大豆U6核小RNA基因启动子及其在植物小RNA基因的组成型表达中的用途的制作方法
本申请要求于2015年3月27日提交的美国临时申请号62/139,075的权益,该申请通过引用以其全文结合在此。
发明领域
本披露涉及植物u6核小rna(snrna)基因启动子及其片段以及它们在植物中以组织独立性或组成型方式改变至少一个异源核苷酸序列的表达的用途。
以电子方式提交的序列表的引用
该序列表的官方副本经由efs-web作为ascii格式的序列表以电子方式提交,文件名为“20160303_bb2332pct_seqlst.txt”,创建于2016年3月3日,且具有68的大小,并与本说明书同时提交。包括在该ascii格式的文件中的序列表是本说明书的一部分并且以其全文通过引用并入本文。
发明背景
植物遗传工程的最新进展已为构建植物打开了新的大门,这些植物具有改善特征或性状,例如植物抗病性、抗虫性、除草剂抗性、产量提高、植物的可食用部分的营养品质的提高、以及从这些植物获得的最终消费品的稳定性或保质期的改善。重组dna技术使得可以在靶标基因组位置处插入dna序列和/或修饰(编辑)特异性内源染色体序列,从而改变生物体的表型。已经使用了采用位点特异性重组系统的位点特异性整合技术以及其他类型的重组技术来在各种生物体中产生目的基因的靶向插入。重要的是,适当的调节信号必须以适当的构型存在,以获得期望的核苷酸序列在植物细胞中的表达。这些调节信号通常包括启动子区、5′非翻译前导序列和3′转录终止/聚腺苷酸化序列。
启动子是非编码基因组dna序列,通常在相关编码序列的上游(5′),rna聚合酶在启动转录前与之结合。该结合与rna聚合酶对齐,使得转录将在特异性转录起始位点启动。启动子包括能够控制另一核酸片段转录的核酸片段。启动子能够控制编码序列或功能性rna的表达。功能性rna包括但不限于,crrna、tracerrna、转移rna(trna)和核糖体rna(rrna)。已经显示某些启动子能够以比其他启动子更高的速率指导rna合成。这些被称为“强启动子”。已经显示某些其他启动子仅以较高的水平在特定类型的细胞或组织中指导rna合成,并且如果所述启动子优选在某些组织中而且还以降低的水平在其他组织中指导rna合成则通常将其称为“组织特异性启动子”或“组织偏好性启动子”。由于使用启动子控制引入植物的一种或多种嵌合基因的表达模式,所以对于分离能够在特定组织类型或特定植物发育阶段中将一种或多种嵌合基因的表达控制在一定水平的新颖启动子具有持续的关注。
某些启动子能够在植物的所有组织以相对相似的水平指导rna合成。这些称为“组成型启动子”或“组织独立性”启动子。根据其指导rna合成的有效性,组成型启动子可分为强、中、弱。由于在许多情况下有必要在植物的不同组织中同时表达一种或多种嵌合基因来获得该一种或多种基因所期望的功能,所以组成型启动子在此考虑中特别有用。
虽然已经从植物和植物病毒中发现了许多组成型启动子并已对其进行了表征,但是对于分离更新颖的组成型启动子,特别是能够控制表达功能性rna分子的启动子仍然具有持续的关注。
概述
本披露涉及包含与如下启动子有效地连接的至少一个异源核苷酸序列的重组dna构建体,其中所述启动子包含seqidno:1、2、3、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、35、38、40中所示核苷酸序列,或所述启动子包含seqidno:1、2、3、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、35、38、40中所示核苷酸序列的功能性片段,或者当与seqidno:1、2、3、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、35、38、40的核苷酸序列比较时,基于clustalv比对方法,使用逐对比对默认参数(ktuple=2、空位罚分(gappenalty)=5、窗口(window)=4和存储的对角线(diagonalssaved)=4),其中所述启动子包含具有至少71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%和100%序列同一性的核苷酸序列。
在一个实施例中,本披露涉及包含如下核苷酸序列的重组dna构建体,所述核苷酸序列包含seqidno:1、2、3、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、35、38、40中所示序列中任何一个或其功能性片段,有效地连接至至少一个异源序列,其中所述核苷酸序列是组成型启动子。
在一个实施例中,本披露涉及包含如下核苷酸序列的重组dna构建体,当与seqidno:1、2、3、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、35、38、40中所示序列比较时,基于clustalv比对方法,使用逐对比对默认参数(ktuple=2、空位罚分=5、窗口=4和存储的对角线=4),该核苷酸序列具有至少95%同一性。
在一个实施例中,本披露涉及包含与本披露的启动子有效地连接的至少一个异源核苷酸序列的重组dna构建体。
在一个实施例中,本披露涉及包含本披露的重组dna构建体的细胞、植物或种子。
在一个实施例中,本披露涉及包含该重组dna构建体的植物和从这种植物获得的种子。
在一个实施例中,本披露涉及重组dna构建体。
附图和序列表简述
从以下详细描述和构成本申请的一部分的附图和序列表可以更全面地理解本披露。
图1是基于其序列相似性在大豆中发现的18种u6snrna基因的系统发育树(基因由gm-u6-1.1、gm-u6-4.1、gm-u6-4.2、gm-u6-6.1、gm-u6-9.1、gm-u6-12.1、gm-u6-13.1、gm-u6-14.1、gm-u6-15.1、gm-u6-15.2、gm-u6-16.1、gm-u6-16.2、gm-u6-19.1、gm-u6-19.2、gm-u6-19.3、gm-u6-19.4、gm-u6-19.5和gm-u6-19.6表示)。第一个数字表示基因所位于的染色体,并且第二个数字表示染色体上的u6snrna的数目。例如,gmu6-9.1是染色体9上的第一个u6snrna。
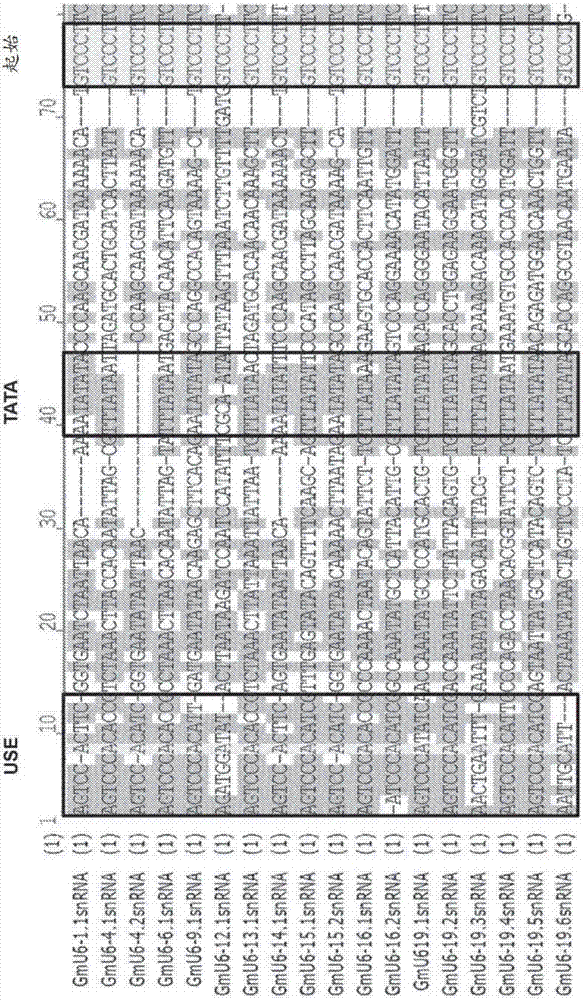
图2是18个大豆u6snrna基因的近似启动子区域的比对(gm-u6-1.1snrna,seqidno:6;gm-u6-4.1snrna,seqidno:8;gm-u6-4.2snrna,seqidno:10;gm-u6-6.1snrna,seqidno:12;gm-u6-9.1snrna,seqidno:14;gm-u6-12.1snrna,seqidno:16;gm-u6-13.1snrna,seqidno:18;gm-u6-14.1snrna,seqidno:20;gm-u6-15.1snrna,seqidno:22;gm-u6-15.2snrna,seqidno:24;gm-u6-16.1snrna,seqidno:26;gm-u6-16.2snrna,seqidno:28;gm-u6-19.1snrna,seqidno:30;gm-u6-19.2snrna,seqidno:32;gm-u6-19.3snrna,seqidno:34;gm-u6-19.4snrna,seqidno:36;gm-u6-19.5snrna,seqidno:38;gm-u6-19.6snrna,seqidno:40)。保守性上游序列元件(use)、tata框和snrna起始区域是加框显示的。
图3a-3b显示质粒qc795和qc819的片段图。图3a示出了包含两个表达盒的qc795的片段图。gm-u6-13.1pro:dd43cr1grna:gm-u6rnaterm盒用于测试表达可通过grna特异性qrt-pcr进行定量的dd43cr1grna的gm-u6-13.1启动子。camv35spro:hpt:piniiterm盒用于表达可选标记和转殖项选择。图3b显示了在qrt-pcr分析中用作对照的含有grna和u6rnapcr扩增子序列的qc819(对照质粒)的片段图。
图4a-4d显示了由四种不同的大豆u6基因启动子(gm-u6-13.1(图4a)、gm-u6-16.1(图4b)、gm-u6-9.1(图4c)和gm-u6-16.2(图4d))表达的dd43cr1指导rna(d43)的相对表达水平。将所有样品标准化为内源性对照基因atp硫酸化酶(atps),并将表达与单一对照进行比较。每列代表独立事件,qpcr确定了所指示的转基因dd43cr1grna盒的拷贝数。
图5a-5b显示质粒qc799和rtw831的片段图。图5a显示了包含两个表达盒的qc799的片段图。gm-u6-9.1pro:dd43cr1grna:gm-u6rnaterm盒用于表达能识别大豆基因组中相应的dd43靶位点的dd43cr1指导rna(grna)。gm-ef1a2pro:cas9(so):piniiterm用于表达大豆密码子优化的cas9蛋白以切割dd43基因组靶位点。图5b显示了含有用于转基因事件选择的gm-samspro:hpt:nosterm表达盒的rtw831的片段图。gm-dd43frag1和gm-dd43frag2两个dna片段与侧翼于dd43位点的基因组dna序列同源,以促进同源重组。
图6是从用qc799和rtw831转化的独立转基因事件检索的dd43cr1序列的比对,以显示通过非同源末端连接(nhej)的dd43grna/cas9介导的dna修复所导致的小序列缺失和插入。20bp的dd43位点是粗体显示的,随后是加框显示的pam序列cgg。小的序列缺失用“-”表示,而序列插入用“^”表示,后面是表示插入序列大小的数字。(seqidno:76=3475_12_1c;seqidno:77=3475_5_6c;seqidno:78=3475_5_3;seqidno:79=3475_12_4c;seqidno:80=3475_1_20b;seqidno:81=3478_5_1a;seqidno:82=3475_5_1a;seqidno:83=3478_12_4b;seqidno:84=3475_5_11b;seqidno:85=3475_5_4a;seqidno:86=33475_12_2a;seqidno:87=3475_5_14a;seqidno:88=3475_1_17a;seqidno:89=3475_5_16c;seqidno:90=3478_8_1a;seqidno:91=3475_5_5c;seqidno:92=3478_5_1b;seqidno:93=3478_6_7c;seqidno:94=3475_1_10a;seqidno:95=3475_1_10c;seqidno:96=3475_5_16a;seqidno:97=3475_5_3b;seqidno:98=3475_1_11a;seqidno:99=3478_1_9b;seqidno:100=3478_1_9;seqidno:101=3478_5_4b;seqidno:102=3478_9_1c;seqidno:103=3478_9_1a)。
序列描述总结了本文所附的序列表。如描述于以下文献中的iupaciub标准中所定义的,序列表包含核苷酸序列字符的单字母代码和氨基酸的单字母和三字母代码:nucleicacidsresearch[核酸研究]13:3021-3030(1985)和biochemicaljournal[生物化学杂志]219(2):345-373(1984)。
seqidno:1是侧翼为xmal(cccggg)和hindiii(aagctt)限制性位点的gm-u6-9.1snrna基因启动子片段。
seqidno:2是侧翼为xma1(cccggg)和hindiii(aagctt)限制性位点的gm-u6-13.1snrna基因启动子片段。
seqidno:3是从xma1(cccgg)限制性位点开始的gm-u6-16.1snrna基因启动子片段。
seqidno:4是从xmal(cccgg)限制性位点开始的gm-u6-16.2snrna基因启动子片段。
seqidno:5是u6snrna基因gm-u6-1.1的预测的转录物序列。
seqidno:6是u6snrna基因gm-u6-1.1的预测的启动子序列。
seqidno:7是u6snrna基因gm-u6-4.1的预测的转录物序列。
seqidno:8是u6snrna基因gm-u6-4.1的预测的启动子序列。
seqidno:9是u6snrna基因gm-u6-4.2的预测的转录物序列。
seqidno:10是u6snrna基因gm-u6-4.2的预测的启动子序列。
seqidno:11是u6snrna基因gm-u6-6.1的预测的转录物序列。
seqidno:12是u6snrna基因gm-u6-6.1的预测的启动子序列。
seqidno:13是u6snrna基因gm-u6-9.1的预测的转录物序列。
seqidno:14是u6snrna基因gm-u6-9.1的预测的启动子序列。
seqidno:15是u6snrna基因gm-u6-12.1的预测的转录物序列。
seqidno:16是u6snrna基因gm-u6-12.1的预测的启动子序列。
seqidno:17是u6snrna基因gm-u6-13.1的预测的转录物序列。
seqidno:18是u6snrna基因gm-u6-13.1的预测的启动子序列。
seqidno:19是u6snrna基因gm-u6-14.1的预测的转录物序列。
seqidno:20是u6snrna基因gm-u6-14.1的预测的启动子序列。
seqidno:21是u6snrna基因gm-u6-15.1的预测的转录物序列。
seqidno:22是u6snrna基因gm-u6-15.1的预测的启动子序列。
seqidno:23是u6snrna基因gm-u6-15.2的预测的转录物序列。
seqidno:24是u6snrna基因gm-u6-15.2的预测的启动子序列。
seqidno:25是u6snrna基因gm-u6-16.1的预测的转录物序列。
seqidno:26是u6snrna基因gm-u6-16.1的预测的启动子序列。
seqidno:27是u6snrna基因gm-u6-16.2的预测的转录物序列。
seqidno:28是u6snrna基因gm-u6-16.2的预测的启动子序列。
seqidno:29是u6snrna基因gm-u6-19.1的预测的转录物序列。
seqidno:30是u6snrna基因gm-u6-19.1的预测的启动子序列。
seqidno:31是u6snrna基因gm-u6-19.2的预测的转录物序列。
seqidno:32是u6snrna基因gm-u6-19.2的预测的启动子序列。
seqidno:33是u6snrna基因gm-u6-19.3的预测的转录物序列。
seqidno:34是u6snrna基因gm-u6-19.3的预测的启动子序列。
seqidno:35是u6snrna基因gm-u6-19.4的预测的转录物序列。
seqidno:36是u6snrna基因gm-u6-19.4的预测的启动子序列。
seqidno:37是u6snrna基因gm-u6-10.5的预测的转录物序列。
seqidno:38是u6snrna基因gm-u6-19.5的预测的启动子序列。
seqidno:39是u6snrna基因gm-u6-19.6的预测的转录物序列。
seqidno:40是u6snrna基因gm-u6-19.6的预测的启动子序列。
seqidno:41是可以通过特异性grna和cas9复合物的碱基配对进行识别的基因组位点dd43-cr1的序列。
seqidno:42是用于grna转基因拷贝数的定量pcr分析和grna转基因表达的定量rt-pcr分析中的正义引物。
seqidno:43是用于grna转基因拷贝数的定量pcr分析和grna转基因表达的定量rt-pcr分析中的fam标记的荧光dna寡核苷酸探针。
seqidno:44是用于grna转基因拷贝数的定量pcr分析和grna转基因表达的定量rt-pcr分析中的反义引物。
seqidno:45是用于35s:hpt转基因拷贝数的定量pcr分析中的正义引物。
seqidno:46是用于35s:hpt转基因拷贝数的定量pcr分析中的fam标记的荧光dna寡核苷酸探针。
seqidno:47是用于35s:hpt转基因拷贝数的定量pcr分析中的反义引物。
seqidno:48是用于作为内源对照的hsp(大豆热休克蛋白基因)的定量pcr分析中的正义引物。
seqidno:49是用于hsp内源对照的定量pcr分析中的vic标记的荧光dna寡核苷酸探针。
seqidno:50是用于hsp内源对照的定量pcr分析中的反义引物。
seoidno:51是用于u6转基因表达的定量rt-pcr分析中的正义引物。
seqidno:52是用于u6转基因表达的定量rt-pcr分析中的fam标记的荧光dna寡核苷酸探针。
seqidno:53是用于u6转基因表达的定量rt-pcr分析中的反义引物。
seqidno:54是用于作为内源对照的atps(大豆atp硫酸化酶基因)的定量pcr和rt-pcr分析中的正义引物。
seqidno:55是用于作为内源对照的atps的定量pcr和rt-pcr分析中的vic标记的荧光dna寡核苷酸探针。
seqidno:56是用于作为内源对照的atps的定量pcr和rt-pcr分析中的反义引物。
seqidno:57是质粒dna片段qc795a的3365bp序列。
seqidno:58是质粒dna片段qc819a的1131bp序列。
seqidno:59是质粒dna片段qc799a的6611bp序列。
seqidno:60是质粒dna片段rtw831a的5719bp序列。
seqidno:61是用于sams:hpt转基因拷贝数的定量pcr分析中的正义引物。
seqidno:62是用于sams:hpt转基因拷贝数的定量pcr分析中的fam标记的荧光dna寡核苷酸探针。
seqidno:63是用于sams:hpt转基因拷贝数的定量pcr分析中的反义引物。
seqidno:64是用于cas9转基因拷贝数的定量pcr分析中的正义引物。
seqidno:65是用于cas9转基因拷贝数的定量pcr分析中的fam标记的荧光dna寡核苷酸探针。
seqidno:66是用于cas9转基因拷贝数的定量pcr分析中的反义引物。
seqidno:67是用于dd43-cr1拷贝数的定量pcr分析中的正义引物。
seqidno:68是用于dd43-cr1拷贝数的定量pcr分析中的fam标记的荧光dna寡核苷酸探针。
seqidno:69是用于dd43-cr1拷贝数的定量pcr分析中的反义引物。
seqidno:70是用于通过pcr扩增dd43-cr1基因组区域的正义引物。
seqidno:71是用于通过pcr扩增dd43-cr1基因组区域的反义引物。
seqidno:72是用于对pcr扩增的dd43-cr1基因组区域进行测序的正义引物。
seqidno:73是用于对pcr扩增的dd43-cr1基因组区域进行测序的反义引物。
seqidno:74是来自野生型大豆基因组的pcr扩增的dd43-cr1基因组区域的全长序列。
seqidno:75是来自野生型大豆基因组的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:76是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:77是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:78是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:79是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:80是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:81是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:82是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:83是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:84是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:85是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:86是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:87是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:88是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seoidno:89是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seoidno:90是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:91是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:92是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:93是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:94是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:95是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seoidno:96是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:97是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:98是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:99是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:100是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seqidno:101是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seoidno:102是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
seoidno:103是已经从转基因大豆转殖项进行修饰的pcr扩增的dd43-cr1基因组区域的部分序列。
发明详述
本文引用的所有专利、专利申请、和出版物的披露内容以其全文通过引用并入。
除非上下文另外明确指示,否则本文和所附权利要求中所使用的单数形式“一个/一种(a/an)”和“该”包括复数指示物。因此,例如,提及“植物”包括多个这样的植物,提及“细胞”包括本领域技术人员已知的一种或多种细胞及其等效物等。
在本披露的上下文中,将使用大量术语。
“分离的多核苷酸”是指单链-或双链的核糖核苷酸(rna)或脱氧核糖核苷酸(dna)的聚合物,其任选地包含合成的、非天然的或改变的核苷酸碱基。dna形式的分离的多核苷酸可以由cdna、基因组dna或合成dna的一个或多个区段组成。
术语“多核苷酸”、“多核苷酸序列”、“核酸序列”、“核酸片段”和“分离的核酸片段”在本文中可互换使用。这些术语涵盖核苷酸序列等。多核苷酸可以是单链-或双链的rna或dna的聚合物,其任选地包含合成的、非天然的或改变的核苷酸碱基。dna聚合物形式的多核苷酸可以由cdna、基因组dna、合成dna或其混合物的一个或多个区段组成。核苷酸(通常以其5′-单磷酸形式被发现)通过单字母名称表示如下:“a”表示腺苷酸或脱氧腺苷酸(分别用于rna或dna),“c”表示胞苷酸或脱氧胞苷酸,“g”表示鸟苷酸或脱氧鸟苷酸,“u”表示尿苷酸,“t”表示脱氧胸苷酸,“r”表示嘌呤(a或g),“y”表示嘧啶(c或t),“k”表示g或t,“h”表示a或c或t,“i”表示肌苷,并且“n”表示任何核苷酸。
crispr基因座(规律间隔成簇短回文重复序列)(又称spidr--间隔区散在同向重复序列)构成dna基因座的家族。crispr基因座由部分回文的短而高度保守的dna重复序列(通常为24至40bp,重复从1至140次,也称为crispr重复序列)组成。重复序列(通常具有物种特异性)由恒定长度的可变序列(通常为20至58,依赖于crispr基因座(wo2007/025097,2007年3月1日公开))间隔。细菌和古细菌已经发展了适应性免疫防御,称为使用短rna直接降解外来核酸的规律间隔成簇短回文重复序列(crispr)/crispr相关(cas)系统(wo2007/025097,2007年3月1日公开)。来自细菌的ii型crispr/cas系统使用crrna(crisprrna)和tracrrna(反式激活crisprrna)来将cas内切核酸酶指导到其dna靶标上。crrna包含与双链dna靶的一条链互补的区域和与tracrrna(反式激活crisprrna)碱基配对的区域,这两个区域形成指导cas内切核酸酶切割dna靶标的rna双链体。
cas基因包括通常与侧翼crispr基因座偶合、相关或相近或相邻的基因。术语“cas基因”、“crispr相关(cas)基因”在本文中可互换使用。对cas蛋白家族的全面综述呈现于以下文献中:haft等人,(2005)computationalbiology[计算生物学],ploscomputbiol[plos计算生物学]1(6):e60.doi:10.1371/期刊.pcbi.0010060。如其中所述,除了四个先前已知的基因家族之外,还描述了41个crispr相关(cas)基因家族。它表明crispr系统属于不同类别,具有不同的重复模式、基因组和种类范围。在给定的crispr基因座处的cas基因数目在物种之间可以不同。
本文中术语crispr相关(“cas”)内切核酸酶是指由cas(crispr相关)基因编码的蛋白。当与合适的rna组分复合时,cas内切核酸酶在某些实施例中能够切割全部或部分特异性dna靶序列。例如,其能够在特异性dna靶序列中引入单链或双链断裂;可替代地其可以被表征为能够切割特异性dna靶序列的一条或两条链。cas内切核酸酶在靶序列处解开dna双链体并切割至少一条dna链,如通过与cas复合的crrna或指导rna识别靶序列所介导的。如果正确的前间区序列邻近基序(pam)位于或相邻于dna靶序列的3′末端,则通过cas内切核酸酶对靶序列进行的此类识别和切割通常会发生。可替代地,本文中的cas蛋白质可能缺乏dna切割或切口活性,但是当与合适的rna组分复合时,仍然可以特异性结合dna靶序列。本文中优选的cas蛋白是cas9。(另见2015年3月19日公开的美国专利申请us2015-0082478a1和2015年2月26日公开的us2015-0059010a1,两者均通过引用以其全文并入本文)。
本文中“cas9”(以前称为cas5、csn1、或csx12)是指与cr核苷酸和tracr核苷酸或与单一指导多核苷酸形成复合物的ii型crispr系统的cas内切核酸酶,用于特异性识别和切割全部或部分的dna靶序列。cas9蛋白包含ruvc核酸酶域和hnh(h-n-h)核酸酶域,其各自在靶序列上切割单个dna链(两个域的协同作用导致dna双链切割,而一个域的活性导致一个缺口)。通常,ruvc域包含子域i、ii和iii,其中子域i位于cas9的n末端附近,并且子域ii和iii位于蛋白质的中间,即位于hnh域的侧翼(hsu等人,cell[细胞],157:1262-1278)。“apo-cas9”是指不与rna组分复合的cas9。apo-cas9可以结合dna,但以非特异性方式进行,且不能切割dna(sternberg等人,nature[自然],507:62-67)。术语“ii型crispr系统”和“ii型crispr-cas系统”在本文中可互换使用,并且是指利用与至少一种rna组分复合的cas9内切核酸酶的dna切割系统。例如,cas9可以与crisprrna(crrna)和反式激活crisprrna(tracrrna)复合。在另一个实例中,cas9可以与单一指导rna复合。因此,crrna、tracrrna和单一指导rna是本文中rna组分的非限制性实例。
在一个实施例中,cas内切核酸酶基因是ii型cas9内切核酸酶基因,例如但不限于,2007年3月1日公开的并通过引用并入本文的wo2007/025097的seqidno:462、474、489、494、499、505和518中列出的cas9基因。在另一个实施例中,cas内切核酸酶基因是植物、玉米或大豆优化的cas9内切核酸酶基因。cas内切核酸酶基因可以有效地连接到cas密码子区域的sv40核靶向信号上游和cas密码子区域的二分型vird2核定位信号(tinland等人,(1992)proc.natl.acad.sci.usa[美国国家科学院院刊]89:7442-6)下游。
术语“功能片段”、“功能上等效的片段”和“功能等效片段”在本文中可互换使用。这些术语是指本披露的cas内切核酸酶序列的部分或子序列,其中在靶位点中产生单链或双链断裂的能力被保留。
术语“功能变体”、“功能上等效的变体”和“功能等效变体”在本文中可互换使用。这些术语是指本披露的cas内切核酸酶的变体,其中保留在靶位点产生单链或双链断裂的能力。片段和变体可以通过诸如定点诱变和合成构建的方法获得。
在一个实施例中,cas内切核酸酶基因是可以识别n(12-30)型ngg的任何基因组序列并且原则上是可以靶向的植物密码子优化的酿脓链球菌cas9基因。
cas内切核酸酶可以通过本领域已知的任何方法(例如但不限于瞬时引入方法、转染和/或局部应用)直接引入细胞。
术语“指导多核苷酸/cas内切核酸酶复合物”、“指导多核苷酸/cas内切核酸酶系统”、“指导多核苷酸/cas复合物”、“指导多核苷酸/cas系统”在本文中可互换使用并且指能够形成复合物的指导多核苷酸和cas内切核酸酶,其中所述指导多核苷酸/cas内切核酸酶复合物可将cas内切核酸酶指导至基因组靶位点,使cas内切核酸酶能够将单链或双链断裂引入基因组靶位点。
如本文中所使用,术语“指导多核苷酸”涉及可以与cas内切核酸酶形成复合物的多核苷酸序列,并且使得cas内切核酸酶能够识别并任选地切割dna靶位点。指导多核苷酸可以是单分子或双分子。指导多核苷酸序列可以是rna序列、dna序列或其组合(rna-dna组合序列)。任选地,指导多核苷酸可以包含至少一种核苷酸、磷酸二酯键或连接修饰,例如但不限于锁核酸(lna)、5-甲基dc、2,6-二氨基嘌呤、2′-氟代a、2’-氟代u、2′-o-甲基rna、硫代磷酸酯键、与胆固醇分子的连接、与聚乙二醇分子的连接、与间隔子18(六乙二醇链)分子的连接、或导致环化的5′至3′共价连接。仅仅包含核糖核酸的指导多核苷酸也被称为“指导rna”或“grna”(也参见2015年3月19日公开的美国专利申请us2015-0082478a1和2015年2月26日公开的us2015-0059010a1,两者均通过引用特此以其全文并入)。
指导多核苷酸可以是包含cr核苷酸序列和tracr核苷酸序列的双分子(也称为双链体指导多核苷酸)。cr核苷酸包括可以与靶dna中的核苷酸序列杂交的第一个核苷酸序列区域(称为可变靶向域或vt域)和作为cas内切核酸酶识别(cer)域的一部分的第二核苷酸序列(也称为tracr配对序列)。tracr配对序列可以沿互补性的区域与tracr核苷酸杂交,并一起形成cas内切核酸酶识别域或cer域。cer域能够与cas内切核酸酶多肽相互作用。双链体指导多核苷酸的cr核苷酸和tracr核苷酸可以是rna、dna和/或rna-dna组合序列。在一些实施例中,双链体指导多核苷酸的cr核苷酸分子被称为“crdna”(当由dna核苷酸的连续延伸构成时)或“crrna”(当由rna核苷酸的连续延伸构成时)或“crdna-rna”(当由dna和rna核苷酸的组合构成时)。该cr核苷酸可以包含天然存在于细菌和古细菌中的crna的片段。可以存在于本文披露的cr核苷酸中的细菌和古细菌中天然存在的crna片段的大小可以是但不限于2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20或更多个核苷酸。在一些实施例中,tracr核苷酸被称为“tracrrna”(当由rna核苷酸的连续延伸构成时)或“tracrdna”(当由dna核苷酸的连续延伸构成时)或“tracrdna-rna”(当由dna和rna核苷酸的组合构成时)。在一个实施例中,指导rna/cas9内切核酸酶复合物的rna是包含双链体crrna-tracrrna的双链体化的rna。
在5’-至-3′方向上,tracrrna(反式激活crisprrna)包含(i)与crisprii型crrna的重复区退火的序列和(ii)含茎环的部分(deltcheva等人,nature[自然]471:602-607)。双链体指导多核苷酸可与cas内切核酸酶形成复合物,其中所述指导多核苷酸/cas内切核酸酶复合物(也称为指导多核苷酸/cas内切核酸酶系统)可将cas内切核酸酶指导至基因组靶位点,使得cas内切核酸酶在基因组靶位点引入单链或双链断裂。(也参见2015年3月19日出版的美国专利申请us2015-0082478a1和2015年2月26日出版的us2015-0059010a1,两者均通过引用特此以其全文并入)。
指导多核苷酸也可以是包含连接至tracr核苷酸序列的cr核苷酸序列的单分子(也称为单指导多核苷酸)。单指导多核苷酸包含可以与靶dna中的核苷酸序列杂交的第一核苷酸序列区域(称为可变靶向域或vt域)和与cas内切核酸酶多肽相互作用的cas内切核酸酶识别域(cer域)。“域”意指可以为rna、dna和/或rna-dna组合序列的核苷酸的连续延伸。单指导多核苷酸的vt域和/或cer域可以包含rna序列、dna序列或rna-dna组合序列。由来自cr核苷酸和tracr核苷酸的序列构成的单指导多核苷酸可以被称为“单指导rna”(当由rna核苷酸的连续延伸构成时)或“单指导dna”(当由dna核苷酸的连续延伸构成时)或“单指导rna-dna”(当由rna和dna核苷酸的组合构成时)。单指导多核苷酸可与cas内切核酸酶形成复合物,其中所述指导多核苷酸/cas内切核酸酶复合物(也称为指导多核苷酸/cas内切核酸酶系统)可将cas内切核酸酶指导至基因组靶位点,使得cas内切核酸酶在基因组靶位点引入单链或双链断裂。(另见2015年3月19日公开的美国专利申请us2015-0082478a1和2015年2月26日公开的us2015-0059010a1,两者均通过引用以其全文并入本文)。
术语“可变靶向域”或“vt域”在本文中可互换使用,并且包括能与双链dna靶位点的一条链(核苷酸序列)杂交(互补)的核苷酸序列。第一个核苷酸序列区域(vt域)与靶序列之间的互补%可以为至少50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、63%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%。可变靶标域可以是至少12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29或30个核苷酸长度。在一些实施例中,可变靶向域包含12至30个核苷酸的连续延伸。可变靶向域可以由dna序列、rna序列、修饰的dna序列、修饰的rna序列或其任意组合构成。
术语指导多核苷酸的“cas内切核酸酶识别域”或“cer域”在本文中可互换使用,并且包括与cas内切核酸酶多肽相互作用的核苷酸序列。cer域包含tracr核苷酸配对序列、随后是tracr核苷酸序列。cer域可以由dna序列、rna序列、修饰的dna序列、修饰的rna序列(参见例如2015年2月26日公开的us2015-0059010a1,其通过引用以其全文并入本文)或其任何组合构成。
连接单指导多核苷酸的cr核苷酸和tracr核苷酸的核苷酸序列可以包含rna序列、dna序列或rna-dna组合序列。在一个实施例中,连接单指导多核苷酸的cr核苷酸和tracr核苷酸的核苷酸序列可以是至少3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99或100个核苷酸的长度。在另一个实施例中,连接单指导多核苷酸的cr核苷酸和tracr核苷酸的核苷酸序列可以包含四核苷酸环序列,例如但不限于gaaa四核苷酸环序列。
指导多核苷酸、vt域和/或cer域的核苷酸序列修饰可以选自但不限于由以下各项组成的组:5′帽、3’聚腺苷酸尾、核糖开关序列、稳定性控制序列、形成dsrna双链体的序列、将指导多核苷酸靶向亚细胞位置的修饰或序列、提供跟踪的修饰或序列、提供蛋白质结合位点的修饰或序列、锁核酸(lna)、5-甲基dc核苷酸、2,6-二氨基嘌呤核苷酸、2’-氟a核苷酸、2’-氟u核苷酸、2′-o-甲基rna核苷酸、硫代磷酸酯键、与胆固醇分子的连接、与聚乙二醇分子的连接、与间隔物18分子的连接、5′至3′共价连接、或其任何组合。这些修饰可以产生至少一个另外的有益特征,其中该另外的有益特征选自由以下各项组成的组:修饰的或调节的稳定性、亚细胞靶向、跟踪、荧光标记、用于蛋白质或蛋白质复合物的结合位点、对互补靶序列的修饰的结合亲和力、修饰的细胞降解抗性和增加的细胞通透性。
如本文使用的,术语“单指导rna”和“sgrna”在本文中可互换使用,并涉及两个rna分子的合成融合,其中包含可变靶向域(与tracrrna杂交的tracr配对序列连接)的crrna(crisprrna)与tracrrna(反式激活crisprrna)融合。单指导rna可以包含可与ii型cas内切核酸酶形成复合物的ii型crispr/cas系统的crrna或crrna片段和tracrrna或tracrrna片段,其中所述指导rna/cas内切核酸酶复合物可将cas内切核酸酶指导至基因组靶位点,使得cas内切核酸酶能够将单链或双链断裂引入基因组靶位点。
术语“指导rna/cas内切核酸酶复合物”、“指导rna/cas内切核酸酶系统”、“指导rna/cas复合物”、“指导rna/cas系统”、“grna/cas复合物”、“grna/cas系统”、“rna指导的内切核酸酶”、“rgen”在本文中可互换使用并且指能够形成复合物的至少一种rna组分和至少一种cas内切核酸酶,其中所述指导rna/cas内切核酸酶复合物可将cas内切核酸酶指导至基因组靶位点,使cas内切核酸酶能够将单链或双链断裂引入基因组靶位点。本文中的指导rna/cas内切核酸酶复合物可以包含四种已知的crispr系统(horvath和barrangou,science[科学]327:167-170)(例如i型、ii型或iii型crispr系统)中任一种的一种或多种cas蛋白和一种或多种合适的rna组分。在优选的实施例中,指导rna/cas内切核酸酶复合物包含cas9内切核酸酶(crisprii系统)和至少一种rna组分(例如,crrna和tracrrna、或grna)。
可以使用本领域已知的任何方法(例如但不限于粒子轰击或局部应用)将指导多核苷酸直接引入细胞。指导多核苷酸还可以通过引入(通过,例如但不限于粒子轰击或农杆菌转化等方法)包含编码指导多核苷酸的异源核酸片段的重组dna分子被间接引入细胞,该重组dna分子有效地连接于能够在所述细胞转录该指导rna的特异性启动子。特异性启动子可以是但不限于rna聚合酶iii启动子,其允许具有精确定义的未修饰的5′-和3′-末端的rna转录(dicarlo等人,nucleicacidsres.[核酸研究]41:4336-4343;ma等人,mol.ther.nucleicacids[核酸分子研究]3:e161)。
术语“靶基因”、“靶序列”、“靶dna”、“靶基因座”、“基因组靶位点”、“基因组靶序列”和“基因组靶基因座”在本文中可互换使用并且是指在此处通过cas内切核酸酶在细胞基因组中诱导单链或双链断裂的细胞的基因组(包括叶绿体dna、线粒体dna、质粒dna)中的多核苷酸序列。靶位点可以是细胞基因组中的内源性位点,或者可替代地,靶位点可以与细胞异源并且从而不是天然存在于细胞的基因组中,或者与在自然界发生的位置相比,可以在异质基因组位置中找到靶位点。如本文所用,术语“内源性靶序列”和“天然靶序列”在本文中可互换使用,指的是对细胞基因组来说是内源的或天然的、并且位于细胞基因组中该靶序列的内源或天然位置处的靶序列。细胞包括但不限于人、动物、细菌、真菌、昆虫和植物的细胞,以及通过本文所述方法产生的植物和种子的细胞。
本文中的“前间区序列邻近基序”(pam)是指由本文中的指导多核苷酸/cas系统识别的短序列。本文中的pam的序列和长度可以取决于所使用的cas蛋白或cas蛋白复合物而不同,但通常为例如2、3、4、5、6、7或8个核苷酸长。
指导多核苷酸/cas内切核酸酶系统可以与共同递送的多核苷酸修饰模板组合使用以允许编辑(修饰)目的基因组核苷酸序列。(也参见2015年3月19日公开的美国专利申请us2015-0082478a1和2015年2月26日公开的wo2015/026886a1,两者均通过引用特此以其全文并入)。
“修饰的核苷酸”或“编辑的核苷酸”是指当与其非修饰的核苷酸序列相比时,包含至少一个改变的目的核苷酸序列。例如,此类“改变”包括:(i)至少一个核苷酸的替代、(ii)至少一个核苷酸的缺失、(iii)至少一个核苷酸的插入、或(iv)(i)-(iii)的任意组合。
术语“多核苷酸修饰模板”包括,当与待编辑的核苷酸序列相比时,包含至少一个核苷酸修饰的多核苷酸。核苷酸修饰可以是至少一个核苷酸取代、添加或缺失。任选地,多核苷酸修饰模板可以进一步包含位于至少一个核苷酸修饰侧翼的同源核苷酸序列,其中侧翼同源核苷酸序列为待编辑的希望的核苷酸序列提供了充足同源性。
在一个实施例中,本披露描述了用于编辑细胞基因组中的核苷酸序列的方法,该方法包括向细胞提供指导多核苷酸、多核苷酸修饰模板和至少一种cas内切核酸酶,其中该cas内切核酸酶能够在所述细胞的基因组中的靶序列处引入单链或双链断裂,其中所述多核苷酸修饰模板包括所述核苷酸序列的至少一个核苷酸修饰。细胞包括但不限于人、动物、细菌、真菌、昆虫和植物的细胞,以及通过本文所述方法产生的植物和种子的细胞。待编辑的核苷酸可以位于由cas内切核酸酶识别和切割的靶位点的内部或外部。在一个实施例中,该至少一个核苷酸修饰不是由cas内切核酸酶识别和切割的靶位点上的修饰。在另一个实施例中,该待编辑的至少一个核苷酸和基因组靶位点之间有至少1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、30、40、50、100、200、300、400、500、600、700、900或1000个核苷酸。
在一个实施例中,本披露描述了用于编辑植物细胞基因组中的核苷酸序列的方法,该方法包括向植物细胞提供指导rna、多核苷酸修饰模板和至少一种植物优化的cas9内切核酸酶,其中该植物优化的cas9内切核酸酶能够在植物基因组中的mocas9靶序列处提供双链断裂,其中所述多核苷酸修饰模板包括所述核苷酸序列的至少一个核苷酸修饰。
可以使用任何可用的基因编辑方法来完成基因组编辑。例如,可以通过向宿主细胞中引入含有宿主细胞基因组内的基因的靶向修饰的多核苷酸修饰模板(有时也称为基因修复寡核苷酸)来完成基因编辑。用于这类方法的多核苷酸修饰模板可以是单链或双链。这类方法的实例通常描述于例如美国公开号2013/0019349中。
在一些实施例中,可以通过在所期望的改变附近的基因组中在限定位置诱导双链断裂(dsb)来促进基因编辑。可以使用可用的任何dsb诱导剂诱导dsb,该诱导剂包括但不限于,talen、大范围核酸酶、锌指核酸酶、cas9-grna系统(基于细菌性crispr-cas系统)等。在一些实施例中,可以将dsb的引入与多核苷酸修饰模板的引入组合。
编辑组合有dsb和修饰模板的基因组序列的过程通常包括:向宿主细胞提供dsb诱导剂或编码dsb诱导剂的核酸(识别染色体序列中的靶序列并且能够诱导基因组序列中的dsb),和与待编辑的核苷酸序列相比时包含至少一个核苷酸改变的至少一个多核苷酸修饰模板。多核苷酸修饰模板还可以包含侧翼于该至少一个核苷酸改变的核苷酸序列,其中侧翼序列与侧翼于dsb的染色体区域基本同源。例如,以下申请中已经描述了使用dsb诱导剂(如cas9-grna复合物)进行基因组编辑:2015年3月19日公开的美国专利申请us2015-0082478a1、2015年2月26日公开的wo2015/026886a1申请、2015年7月1日提交的pct/us15/38767申请和2015年7月21日提交的pct/us15/41256申请,以上各项均通过引用并入本文。
指导rna/cas内切核酸酶系统的另外的用途已进行了描述(参见2015年3月19日公开的美国专利申请us2015-0082478a1、2015年2月26日公开的wo2015/026886a1、2015年2月26日公开的us2015-0059010a1、2015年7月1日提交的pct/us15/38767申请和2015年7月21日提交的pct/us15/41256申请,以上各项均通过引用并入本文),并包括,但不限于修饰或取代目的核苷酸序列(如调节元件)、目的多核苷酸插入、基因敲除、基因敲入、剪接位点的修饰和/或引入交替剪接位点、编码目的蛋白的核苷酸序列的修饰、氨基酸和/或蛋白质融合物、以及通过在目的基因中表达反向重复序列的基因沉默。
“大豆u6启动子”、“gm-u6启动子”或“u6启动子”在本文中可互换使用,并且是指推定的大豆基因的启动子或其功能片段,其与在国家生物技术信息中心(ncbi)数据库中保存的各种植物物种(包括大豆)中鉴定的真核甘油醛-3-磷酸脱氢酶基因具有显著同源性。术语“大豆u6启动子”包括天然大豆启动子(或其功能片段)和至少包含附接dna连接子的天然大豆启动子片段的工程改造的序列,以促进克隆。dna接头可以包含限制性酶位点。
“启动子”指能够控制另一核酸片段转录的核酸片段。启动子能够控制编码序列或功能性rna的表达。功能性rna包括但不限于,crrna、tracerrna、指导rna、转移rna(trna)和核糖体rna(rrna)。启动子序列由近端元件和较远端上游元件组成,后者的元件通常称为增强子。因此,“增强子”是可以刺激启动子活性的dna序列,并且可以是启动子的固有元件或插入的异源元件,用来增强启动子的水平或组织特异性。启动子可以全部来源于天然基因,或者由源自于在自然界存在的不同启动子的不同元件构成,或者甚至包含合成的dna区段。本领域技术人员应当理解,不同的启动子可在不同的组织或细胞类型中,或者在不同的发育阶段,或者响应不同的环境条件而指导基因或功能性rna的表达。不断发现可用于植物细胞中的各种新型启动子;在okamuro和goldberg的编译(biochemistryofplants[植物生物化物]15:182(1989))中可以找到许多实例。还应认识到,因为在大多数情况下还不能完全限定调节序列的确切范围,具有某一变异的启动子dna片段也可以具有启动子活性。
“在植物中具有功能性的启动子”是能够控制植物细胞中的转录的启动子,无论其是否来源于植物细胞。
“组织特异性启动子”和“组织偏好性启动子”可互换使用,用来指主要但不一定仅在一个组织或器官中表达,但也可以在一个特定细胞中表达的启动子。
“受发育调控的启动子”指其活性由发育事件决定的启动子。
“组成型启动子”是指在所有或大多数发育阶段的植物的全部或多数组织或细胞类型中起作用的启动子。与分类为“组成型”(例如泛素)的其他启动子一样,绝对表达水平的某一变异可以存在于不同组织或阶段之间。术语“组成型启动子”或“组织独立性”在本文中可互换使用。
本文披露的启动子核苷酸序列和方法可用于调节宿主植物中任何异源核苷酸序列(例如但不限于rna序列)的组成型表达,以改变植物的表型。
“异源核苷酸序列”是指不与本披露的植物启动子序列一起天然存在的序列。虽然这种核苷酸序列对启动子序列来说是异源的,但相对植物宿主,可以是同源的或天然的、或者异源的、或者外来的。然而,应当认识到,即时启动子可以与其天然编码序列一起使用以增加或减少导致转化的种子中表型变化的表达。术语“异源核苷酸序列”、“异源序列”、“异源核酸片段”和“异源核酸序列”在本文中可互换使用。
本披露涵盖包含本文披露的启动子序列的功能性片段的重组dna构建体。
“功能性片段”是指本披露的启动子序列的部分或子序列,其中启动转录或者驱动基因表达(例如产生某种表型)或功能性rna的表达的能力被保留。可以通过诸如定点诱变和合成构建的方法获得片段。与本文所述的提供的启动子序列一样,功能性片段作用为促进有效地连接的异源核苷酸序列的表达,形成重组dna构建体(也称为嵌合基因)。例如,该片段可用于重组dna构建体的设计中以产生能够与cas内切核酸酶相互作用并形成指导rna/cas内切核酸酶复合物的指导rna。可以通过将相对于异源核苷酸序列而言适当方向上的启动子片段连接起来,针对共抑制或反义设计重组dna构建体。
功能上等效于本披露的启动子的核酸片段是能够以与本披露的启动子相似的方式控制编码序列或功能性rna的表达的任何核酸片段。
在本披露的一个实施例中,可以修饰本文披露的启动子。本领域技术人员可以产生在多核苷酸序列上具有变异的启动子。可以对如1、2、3、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、35、38、40中所示的本披露的启动子的多核苷酸序列进行修饰或改变以增强其控制特征。如本领域普通技术人员将理解的,启动子序列的修饰或改变也可以在不显著影响启动子功能的情况下进行。这些方法是本领域技术人员所熟知的。可以修改序列,例如通过在基于pcr的dna修饰方法中插入、缺失或取代模板序列。
如本文所用的“变体启动子”是启动子的序列或包含变化(原始序列的一个或多个核苷酸发生缺失、添加和/或取代)但是基本上维持启动子功能的启动子的功能性片段的序列。可以向启动子内部插入、缺失或取代一个或多个碱基对。在启动子片段的情况下,变体启动子可以包括影响其有效地连接于的最小启动子转录的变化。可以例如通过标准dna诱变技术或者通过化学合成变体启动子或其部分来产生变体启动子。
用于构建本披露的嵌合和变体启动子的方法包括,但不限于组合不同启动子的控制元件或启动子的重复部分或区域(参见例如,美国专利号4,990,607;美国专利号5,110,732;和美国专利号5,097,025)。本领域技术人员熟悉标准资源材料,这些材料描述了大分子(例如,多核苷酸分子和质粒)的构建、操作和分离以及重组生物的产生和多核苷酸分子的筛选和分离的具体条件和程序。
术语“全互补序列”和“全长互补序列”在本文中互换使用,并且是指给定核苷酸序列的互补序列,其中该互补序列和核苷酸序列由相同数目的核苷酸组成并且100%互补。
如本文所用的术语“基本上相似”和“基本上对应”是指核酸片段,其中一个或多个核苷酸碱基的改变不影响核酸片段介导基因表达或产生某种表型的能力。这些术语还指本披露的核酸片段的修饰,例如相对于初始的、未经修饰的片段,基本上不改变所得核酸片段的功能性质的一个或多个核苷酸的缺失或插入。因此,应当理解(正如本领域技术人员将会理解的),本披露不仅仅涵盖这些具体的示例性序列。
可以修饰包含在本披露的重组dna构建体中的分离的启动子序列,以提供一系列组成型表达水平的异源核苷酸序列。因此,可以利用少于整个启动子的区域并且驱动编码序列表达的能力被保留。然而,认识到,mrna的表达水平可能随着启动子序列部分的缺失而降低。同样,表达的组织独立性、组成性性质可能会改变。
本披露的经分离的启动子序列的修饰可以提供异源核苷酸序列的一系列组成型表达。因此,它们可以被修饰为弱组成型启动子或强组成型启动子。通常,“弱启动子”意指以低水平驱动编码序列表达的启动子。“低水平”意指约1/10,000转录物至约1/100,000转录物至约1/500,000转录物的水平。相反地,强启动子以高水平或者说以约1/10转录物至约1/100个转录物至约1/1,000转录物的水平驱动编码序列的表达。
此外,本领域技术人员认识到,本披露所涵盖的基本相似的核酸序列还通过其在中度严格条件(例如,0.5xssc,0.1%sds,60℃)下与本文示例的序列或本文报道的、与本披露的启动子功能上等效的核苷酸序列的任何部分杂交的能力来定义。这种同源性的估计可以通过如本领域技术人员所熟知的在严格条件下的dna-dna或dna-rna杂交来提供(hames和higgins编辑;nucleicacidhybridization[核酸杂交];英国牛津大学irl出版社,1985)。可以调整严格条件以筛选适度相似的片段(例如来自远缘生物体的同源序列),至高度相似的片段(例如复制来自近缘生物体的功能性酶的基因)。杂交后洗涤部分地确定了严格条件。一套条件使用一系列洗涤,从6xssc、0.5%sds开始,在室温下持续15分钟,然后用2xssc、0.5%sds在45℃下重复30分钟,并且然后用0.2xssc、0.5%sds在50℃下持续30分钟,重复两次。另一组严格条件使用更高的温度,其中洗涤与上述洗涤相同,除了将在0.2xssc、0.5%sds中最后两次30分钟的洗涤中的温度增加至60℃。另一组高度严格条件使用在65℃下在0.1xssc、0.1%sds中两次最终洗涤。
本披露所涵盖的优选的基本上相似的核酸序列是与本文报道的核酸片段80%同一或与本文报道的核苷酸序列的任何部分80%同一的那些序列。更优选的是与本文报道的核酸序列90%同一或与本文报道的核苷酸序列的任何部分90%同一的核酸片段。最优选的是与本文报道的核酸序列95%同一或与本文报道的核苷酸序列的任何部分95%同一的核酸片段。本领域技术人员非常了解,许多水平的序列同一性可用于鉴定相关的多核苷酸序列。同一性百分比的有用实例是上面所列出的,或者也优选是从71%到100%的任何整数百分比,例如71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%和100%。
“基本上同源的序列”是指所披露的序列的变体,例如由定点诱变产生的序列的变体,以及合成衍生的序列。本披露的基本上同源的序列还涉及本文披露的特定启动子核苷酸序列的那些片段,其作用为促进有效地连接的异源核酸片段的组成型表达。这些启动子片段应包含本文披露的特定启动子核苷酸序列的至少约20个连续核苷酸、优选至少约50个连续核苷酸、更优选至少约75个连续核苷酸、甚至更优选至少约100个连续核苷酸。这些片段的核苷酸通常包含特定启动子序列的tata识别序列。可以通过使用限制性酶来切割本文披露的天然存在的启动子核苷酸序列、通过合成来自天然存在的启动子dna序列的核苷酸序列来获得这样的片段,或者可以通过使用pcr技术来获得这样的片段。具体参见mullis等人,methodsenzymol.[酶学方法]55:335-350(1987),和higuchi,r,pcrtechnology:principlesandapplicationsfordnaamplifications[pcr技术:dna扩增的原则和应用];erlich,h.a.,编辑;斯托克顿出版社公司(stocktonpressinc.):纽约,1989。此外,这些启动子片段的变体(例如从定点诱变产生的那些)涵盖于本披露的组合物中。
“密码子简并性”是指允许核苷酸序列变异而不影响经编码的多肽的氨基酸序列的遗传密码上的散度。因此,本披露涉及包含如下核苷酸序列的任何核酸片段,该核苷酸序列编码本文所阐述的全部或大部分氨基酸序列。本领域技术人员非常了解特定宿主细胞在使用核苷酸密码子以指定给定氨基酸时所表现的“密码子偏倚(codon-bia)”。因此,当合成核酸片段使其在宿主细胞中具有改善的表达时,理想的是设计这种核酸片段,使得其密码子使用频率接近宿主细胞的优选密码子的使用频率。
序列比对和同一性百分比计算可以使用设计用于检测同源序列的多种比较方法来确定,这些方法包括但不限于
或者,可以使用比对的clustalw方法。clustalw比对方法(描述于higgins和sharp,cabios.[计算机在生物学中的应用]5:151-153(1989);higgins,d.g.等人,comput.appl.biosci.[计算机应用生物科学]8:189-191(1992))可以在
在一个实施例中,在分子(核苷酸或氨基酸)的整个长度上测定序列同一性%。
通过本领域技术人员对序列的手动评估,或者使用诸如blast(altschul,s.f.等人,j.mol.biol.[分子生物学杂志]215:403-410(1993))和缺口blast(altschul,s.f.等人,nucleicacidsres.[核酸研究]25:3389-3402(1997))等算法的计算机自动序列比较和鉴定,氨基酸或核苷酸序列的“实质部分”包括足够的多肽的氨基酸序列或基因的核苷酸序列,以提供该多肽或基因的推定鉴定。blastn是指将核苷酸查询序列与核苷酸序列数据库进行比较的blast程序。
“基因”包括表达功能性分子(例如但不限于,特定蛋白质)的核酸片段,包括在编码序列之前(5′非编码序列)和之后(3′非编码序列)的调节序列。“天然基因”是指自然界中发现的具有其自身调节序列的基因。
“突变基因”是通过人为干预已经改变的基因。这样的“突变基因”具有通过至少一个核苷酸添加、缺失或取代而与相应的非突变基因的序列不同的序列。在本披露的某些实施例中,突变的基因包含如本文披露的指导多核苷酸/cas内切核酸酶系统导致的改变。突变的植物是包含突变基因的植物。
可互换使用的“嵌合基因”或“重组表达构建体”包括不是天然基因的任何基因,其包含不是在自然界中一起被发现的调节和编码序列。因此,嵌合基因可以包含衍生自不同来源的调节序列和编码序列。
“编码序列”是指编码特异性氨基酸序列的dna序列。“调节序列”是指位于编码序列的上游(5’非编码序列)、内部或下游(3’非编码序列),并且影响相关编码序列的转录、rna加工或稳定性、或者翻译的核苷酸序列。调节序列可包括但不限于启动子、翻译前导序列、内含子和多腺苷酸化识别序列。
“内含子”是转录成rna、但是然后在产生成熟mrna的过程中被切除的基因中的间插序列。该术语也用于切除的rna序列。“外显子”是经转录的基因的序列的一部分,并且在源自该基因的成熟信使rna中被发现,但不一定是编码最终基因产物的序列的一部分。
“翻译前导序列”是指位于基因的启动子序列和编码序列之间的多核苷酸序列。翻译前导序列存在于翻译起始序列的经完全处理的mrna上游。翻译前导序列可能影响初级转录物至mrna的加工、mrna稳定性或翻译效率。已经描述了翻译前导序列的实例(turner,r.和foster,g.d.,molecularbiotechnology[分子生物学]3:225(1995))。
“3’非编码序列”是指位于编码序列下游的dna序列,并且包括聚腺苷酸化识别序列和编码能够影响mrna加工或基因表达的调节信号的其他序列。聚腺苷酸化信号通常表征为影响聚腺苷酸片添加到mrna前体的3′末端。不同的3′非编码序列示例于以下文献中:ingelbrecht等人,plantcell[植物细胞]1:671-680(1989)。
“rna转录物”是指产生自dna序列的rna聚合酶催化的转录产物。当rna转录物是dna序列的完全互补拷贝时,它被称为初级转录物,或者它可以是源自初级转录物的转录后加工的rna序列并被称作成熟rna。“信使rna”(“mrna”)是指无内含子并且可由细胞翻译成蛋白质的rna。“cdna”是指与mrna模板互补并且使用逆转录酶从mrna模板合成的dna。cdna可以是单链或通过使用dna聚合酶i的klenow片段转化成双链。“正义”rna是指包含mrna并且因此可以在细胞内或体外翻译成蛋白质的rna转录物。“反义rna”是指与靶初级转录物或mrna的全部或部分互补,并阻断靶基因的表达或转录物积累的rna转录物(美国专利号5,107,065)。反义rna的互补性可以是相对于特定基因转录物的任何部分而言的,即相对于5′非编码序列、3′非编码序列、内含子、或编码序列而言的。“功能性rna”是指反义rna、核糖酶rna、或可以不进行翻译但是仍对细胞过程具有作用的其他rna。
术语“有效地连接”是指核酸序列在单个核酸片段上的缔合,这样使得一个核酸片段的功能受到另一个的影响。例如,当启动子能够影响编码序列的表达时,它与编码序列有效地连接(即编码序列在启动子的转录控制下)。编码序列可以在正义或反义方向上有效地连接到调节序列上。
术语“启动转录”、“启动表达”、“驱动转录”和“驱动表达”在本文中可互换使用,并且都是指启动子的主要功能。如本披露所详述的,启动子是非编码基因组dna序列,通常在相关编码序列的上游(5′),并且其主要功能是作为rna聚合酶的结合位点,并通过rna聚合酶启动转录。此外,当经转录的rna最终被翻译成相应的多肽时,存在rna(包括功能性rna)的“表达”,或有效地连接的编码核苷酸序列的多肽的表达。
如本文使用的术语“表达”是指功能性终产物(如mrna或蛋白质(前体或成熟的))的产生。
本文所用的术语“表达盒”是指可以将核酸序列或片段移动到其中的离散核酸片段。
基因的表达或过表达涉及该基因的转录并将mrna翻译成前体或成熟蛋白。“反义抑制”是指能够抑制靶蛋白表达的反义rna转录物的产生。“过表达”是指在转基因生物体中的基因产物的生产超过正常或非转化的生物体内的生产水平。“共-抑制”是指能够抑制相同的或基本上相似的外源或内源基因的表达或转录物积累的正义rna转录物的产生(美国专利号5,231,020)。共抑制的机制可能处于dna水平(如dna甲基化)、转录水平或转录后水平。
已通过着眼于在正义取向上与内源mrna具有同源性的核酸序列的过表达,先前设计了植物中的共抑制构建体,其导致与过表达序列具有同源性的所有rna的减少(参见vaucheret等人,plantj.[植物杂志]16:651-659(1998);和gura,nature[自然]404:804-808(2000))。这种现象的总体效率低,并且rna减少的程度广泛变化。最近的工作已经描述了“发夹”结构的用途,该发夹结构在互补方向上将mrna编码序列的全部或部分并入,这产生了已表达的rna的潜在“茎-环”结构(1999年10月21日公开的pct公开号wo99/53050;和2002年1月3日公开的pct公开号wo02/00904)。这增加了回收的转基因植物中共抑制的频率。另一个变异描述了植物病毒序列用于指导近端mrna编码序列的抑制或“沉默”的用途(1998年8月20日公开的pct公开号wo98/36083)。已经获得了遗传和分子证据,表明dsrna介导的mrna切割可能是这些基因沉默现象潜在的保守机制(elmayan等人,plantcell[植物细胞]10:1747-1757(1998);galun,invitrocell.dev.biol.plant[体外细胞与发育生物学-植物]41(2):113-123(2005);pickford等人,cell.mol.lifesci.[细胞与分子生命科学]60(5):871-882(2003))。
如本文所述,当与在具有天然酶或蛋白质的非转基因或野生型植物中可检测的酶活性或蛋白质功能性的水平相比时,“抑制”包括在转基因植物中可检测的酶活性或蛋白质功能性(例如,与蛋白质相关的表型)水平的降低。具有天然酶的植物中的酶活性水平在本文中称为“野生型”活性。具有天然蛋白质的植物中的蛋白质功能性的水平在本文中被称为“野生型”功能性。术语“抑制”包括调低(lower)、降低(reduce)、下调(decline)、减少(decrease)、抑制(inhibit)、消除和预防。这种降低可能是由于天然mrna翻译成活性酶或功能性蛋白的减少。这也可能是由于天然dna转录成mrna的量减少和/或天然mrna的快速降解。术语“天然酶”是指在非转基因或野生型细胞中天然产生的酶。术语“非转基因”和“野生型”在本文中可互换使用。
“改变表达”是指在转基因生物中以与相应的野生型生物体产生的一种或多种基因产物的量显著不同的量或比例产生一种或多种基因产物(即表达增加或减少)。
本文所用的“转化”是指稳定转化和瞬时转化。
“稳定转化”是指将核酸片段引入宿主生物体的基因组中,导致遗传稳定的遗传。一旦经稳定转化,核酸片段稳定地整合入宿主生物体和任何后代的基因组中。包含经转化的核酸片段的宿主生物体被称为“转基因的”生物体。
“瞬时转化”是指将核酸片段引入宿主生物体的细胞核或含dna的细胞器中,导致不具遗传稳定遗传的基因表达。
术语“经引入的”是指向细胞中提供核酸(例如,表达构建体)或蛋白质。经引入的包括提到将核酸合并到真核细胞或原核细胞中,其中核酸可以被并入细胞的基因组中,并且包括提到核酸或蛋白被瞬时提供至细胞中。经引入的包括对稳定或瞬时转化方法以及有性杂交的提及。因此,在将核酸片段(例如,重组dna构建体/表达构建体)插入细胞的上下文中,“引入的”是指“转染”、“转化”或“转导”,并且包括提到将核酸片段并入真核或原核细胞中,其中可以将该核酸片段并入细胞的基因组(例如,染色体、质粒、质体或线粒体dna)中,转化成自主复制子或进行瞬时表达(例如,经转染的mrna)。
“基因组”(当应用于植物细胞时)不仅涵盖在细胞核内发现的染色体dna,而且也涵盖在亚细胞组分(例如线粒体,质体)内发现的细胞器dna。
“植物”包括提到全植物、植物器官、植物组织、种子和植物细胞及其后代。植物细胞包括但不限于得自下列物质的细胞:种子、悬浮培养物、胚、分生区域、愈伤组织、叶、根、芽、配子体、孢子体、花粉和小孢子。
术语“单子叶”和“单子叶植物”在本文中可互换使用。本披露的单子叶植物包括禾本科植物。
术语“双子叶”和“双子叶植物”在本文中可互换使用。本披露的双子叶包括以下家族:十字花科、豆科和茄科。
“子代”包括植物的任何后代。
转基因植物包括例如在其基因组中包含通过转化步骤引入的异源多核苷酸的植物。异源多核苷酸可以稳定地整合到基因组内,这样使得多核苷酸被传递给连续世代。异源多核苷酸可单独地或作为重组dna构建体的部分整合进基因组中。转基因植物还可以在其基因组内包含多于一个异源多核苷酸。各异源多核苷酸均可对所述转基因植物产生不同的性状。异源多核苷酸可以包括起源于外来物种的序列,或者如果来自相同物种,可以是从其天然形式上进行实质修饰的序列。转基因可以包括其基因型已经通过异源核酸的存在改变的任何细胞、细胞系、愈伤组织、组织、植物部分或植物,这些异源核酸包括最初如此改变的那些转基因以及通过有性杂交或无性繁殖从初始转基因产生的那些。通过常规植物育种方法,通过不导致外源多核苷酸的插入的基因组编辑程序,或通过天然存在的事件例如随机异花受精、非重组病毒感染、非重组细菌转化、非重组转座或自发突变的基因组(染色体或染色体外)的改变并不意图被视为转基因。
在本披露的某些实施例中,可育植物是产生有活力的雄性和雌性配子并且自体受精的植物。这样的自体受精的植物可以产生子代植物,而没有来自任何其他植物的配子和其中所含的遗传物质的贡献。本披露的其他实施例可涉及非自体受精植物的用途,因为该植物不产生具有活力或能够以其他方式受精的雄性配子、或雌性配子、或两者。如本文使用的,“雄性不育植物”是不产生有活力的或能够以其他方式受精的雄性配子的植物。如本文使用的,“雌性不育植物”是不产生有活力的或能够以其他方式受精的雌性配子的植物。公认雄性不育和雌性不育植物可以分别是雌性可育的和雄性可育的。进一步认识到,当与雌性可育植物杂交时,雄性可育(但雌性不育)植物可以产生有活力的后代,并且当与雄性可育植物杂交时,雌性可育(但是雄性不育)植物可以产生有活力的后代。
“瞬时表达”是指在选择的某些细胞类型的通过转化方法将转基因基因暂时引入其中的宿主生物体中,常规报道基因如β-葡萄糖苷酸酶(gus)基因,荧光蛋白基因zs-green1、zs-yellow1n1、am-cyan1、ds-red的暂时表达。随后在瞬时基因表达测定后弃去宿主生物体的转化的物质。
本文使用的标准重组dna和分子克隆技术是本领域公知的并且更全面地描述于以下文献中:sambrook,j.等人,molecularcloning:alaboratorymanual[分子克隆:实验室手册];第二版;coldspringharborlaboratorypress:coldspringharbor,newyork,1989[冷泉港实验室出版社:冷泉港,纽约,1989](在下文中“sambrook等人,1989”)或ausubel,f.m.、brent,r.、kingston,r.e.、moore,d.d.、seidman,j.g.、smith,j.a.和struhl,k.,编辑;currentprotocolsinmolecularbiology[分子生物学现代方法];约翰·威利父子出版公司(johnwileyandsons):纽约,1990(在下文中“ausubel等人,1990”)。
“pcr”或“聚合酶链式反应”是用于合成由一系列重复循环(珀金埃尔默塞塔斯仪器公司(perkinelmercetusinstruments),诺沃克(norwalk),康涅狄格州(ct))组成的大量特异性dna区段的技术。通常,使双链dna热变性;将与目标区段的3′边界互补的两条引物在低温下退火,并且然后在中间温度下延伸。一组这三个连续步骤构成一个循环。
术语“质粒”、“载体”和“盒”意指染色体外元件,其通常携带非细胞中心代谢的一部分的基因,并且通常处于环状双链dna片段的形式。此类元件可以是来源于任何来源的单链或双链dna或rna的线性或环状自主复制序列、基因组整合序列、噬菌体或核苷酸序列,其中许多核苷酸序列已连接或重组到单一结构中,该单一结构能够将针对选定基因产物的启动子片段和dna序列连同适当3′未翻译序列引入到细胞中。
术语“重组dna构建体”或“重组表达构建体”可互换使用,并且是指可以将核酸序列或片段移动到其中的离散多核苷酸。优选地,它是包含本披露的启动子的质粒载体或其片段。质粒载体的选择取决于将用于转化宿主植物的方法。本领域技术人员非常了解必须存在于质粒载体上的遗传元件,以便成功地转化、选择和繁殖含有嵌合基因的宿主细胞。技术人员还将认识到,不同的独立转化事件将导致表达的不同水平和模式(jones等人,emboj[欧洲分子生物学学会杂志]4:2411-2418(1985);dealmeida等人,molgengenetics[分子遗传学和普通遗传学]218:78-86(1989)),并且因此必须筛选多个转植项,从而获得显示所希望的表达水平和模式的品系。这种筛选可以通过dna的pcr和southern分析、mrna表达的rt-pcr和northern分析、蛋白质表达的westem分析或表型分析来完成。
表型的各种变化是引人关注的,包括但不限于改变植物中的脂肪酸组成、改变植物的氨基酸含量、改变植物的病原体防御机制等。这些结果可以通过提供异源产物的表达或增加植物中内源产物的表达来实现。或者,可以通过提供一种或多种内源产物,特别是植物中的酶或辅酶因子的表达降低来实现这些结果。这些改变导致转化的植物表型上的变化。
目的基因反映了参与作物发育的那些基因的商业市场和利益。感兴趣的作物和市场的变化,以及随着发展中国家打开国际市场,新作物和技术也将出现。此外,随着我们对农艺学特征和性状(例如产量和杂种优势)的了解增加,用于转化的基因的选择也会相应变化。目的基因的一般类别包括但不限于,参与信息传递(例如锌指)的那些基因、参与通信(例如激酶)的那些基因、以及参与持家(例如热休克蛋白)的那些基因。转基因的更具体类别包括但不限于,编码农艺学重要性状、昆虫抗性、抗病性、除草剂抗性、不育性、谷物或种子特征以及商业产品的基因。目的基因通常包括参与油、淀粉、碳水化合物或营养素代谢的那些基因,以及影响种子大小、植物发育、植物生长调节和产量改善的基因。植物发育和生长调节也指植物各部分(如花、种子、根、叶和嫩芽)的发育和生长调节。
其他商业上期望的性状是赋予冷、热、盐和干旱耐性的基因和蛋白质。
疾病和/或昆虫抗性基因可以编码对具有高产量抑制(例如炭疽病、大豆花叶病毒、大豆异皮线虫、根结线虫、褐斑病、霜霉病、紫斑病、烂种和通常由真菌-腐霉属、疫霉属、丝核菌属、腐皮壳属引起的苗病)的有害生物的抗性。细菌性枯萎病是由大豆细菌性斑点病菌(pseudomonassyringaepv.glycinea)引起的。赋予昆虫抗性的基因包括,例如,苏云金芽胞杆菌毒性蛋白基因(美国专利号5,366,892;5,747,450;5,737,514;5,723,756;5,593,881;以及geiser等人,(1986)gene[基因]48:109);凝集素(vandamme等人,(1994)plantmol.biol.[植物分子生物学]24:825);等等。
除草剂抗性性状可以包括编码针对用于抑制乙酰乳酸合酶(als),特别是磺酰脲类除草剂(例如含有导致这种抗性的突变(特别是s4和/或hra突变)的乙酰乳酸合酶als基因)作用的除草剂的抗性的基因。als基因突变体编码对除草剂氯磺隆的抗性。草甘膦乙酰转移酶(gat)是来自地衣芽孢杆菌的n-乙酰转移酶,其通过基因改组针对广谱除草剂(草甘膦)的乙酰化进行优化,形成转基因植物中草甘膦耐受性的新颖机制的基础(castle等人,(2004)science[科学]304,1151-1154)。
抗生素抗性基因包括例如新霉素磷酸转移酶(npt)和潮霉素磷酸转移酶(hpt)。在经转化的生物体的选择中使用两种新霉素磷酸转移酶基因:新霉素磷酸转移酶i(npti)基因和新霉素磷酸转移酶ii(nptii)基因。第二种使用更广泛。它最初分离自存在于大肠杆菌菌株k12中的转座子tn5。该基因编码氨基糖苷类3′-磷酸转移酶(表示为aph(3′)-ii或nptii),其通过磷酸化使一系列氨基糖苷类抗生素(如卡那霉素、新霉素、遗传霉素和巴龙霉素(paroromycin))失活。nptii广泛用作植物转化的可选标记。它也用于不同生物体中的基因表达和调节研究,部分原因是可以构建保留酶活性的n末端融合物。可通过酶学测定来检测nptii蛋白活性。在其他检测方法中,通过薄层色谱法、斑点印迹分析或聚丙烯酰胺凝胶电泳检测经修饰的底物,即磷酸化抗生素。玉米、棉花、烟草、拟南芥、亚麻、大豆等多种植物已经用nptii基因成功转化。
潮霉素磷酸转移酶(表示为hpt、hph或aphiv)基因最初衍生自大肠杆菌。该基因编码潮霉素磷酸转移酶(hpt),其使氨基环醇抗生素潮霉素b解毒。已经用hpt基因转化了大量的植物,并且已经证明潮霉素b在选择广泛的植物(包括单子叶植物)方面非常有效。大多数植物(例如谷物)对潮霉素b的敏感性高于卡那霉素。同样,hpt基因广泛用于经转化的哺乳动物细胞的选择。hpt基因的序列已被修饰用于植物转化。靠近该酶的羧基(c)-末端的氨基酸残基的缺失和取代增加了某些植物(如烟草)中的抗性水平。同时,该酶的亲水性c-末端已被保持,并且可能对hpt的强活性至关重要。可以使用酶学测定检查hpt活性。非破坏性愈伤组织诱导试验可用于验证潮霉素抗性。
已经在植物中鉴定出参与植物生长和发育的基因。参与细胞分裂素生物合成的一种这样的基因是异戊烯基转移酶(ipt)。细胞分裂素通过刺激细胞分裂和细胞分化在植物生长和发育中起关键作用(sun等人,(2003),plantphysiol.[植物生理学]131:167-176)。
钙依赖性蛋白激酶(cdpk)(主要在植物界中发现的丝氨酸-苏氨酸激酶家族)可能在钙介导的信号通路中起到传感器分子的作用。钙离子是植物生长发育过程中重要的第二信使(harper等人,science[科学]252,951-954(1993);roberts等人,curr.opin.cellbiol.[细胞生物学新观点]5,242-246(1993);roberts等人,annu.rev.plantmol.biol.[植物分子生物学年鉴]43,375-414(1992))。
线虫响应蛋白(nrp)是大豆异皮线虫感染后由大豆产生的。nrp与调味糖蛋白神奇蛋白(miraculin)和参与肿瘤形成和超反应诱导的nf34蛋白具有同源性。据信nrp的功能是作为响应线虫感染的防御诱导剂(tenhaken等人,bmcbioinformatics[bmc生物信息学]6:169(2005))。
种子和谷物的质量反映在诸如饱和和不饱和的脂肪酸或油的水平和类型、必需氨基酸的质量和数量以及碳水化合物的水平等性状上。因此,也可以在如下一种或多种基因上编码商业性状,该一种或多种基因可以增加少量存在于大豆中的两种含硫氨基酸(例如甲硫氨酸和半胱氨酸)。胱硫醚γ-合成酶(cgs)和丝氨酸乙酰转移酶(sat)是分别参与甲硫氨酸和半胱氨酸合成的蛋白质。
其他商业性状可以编码基因以增加例如油籽中的单不饱和脂肪酸,例如油酸。例如,大豆油含有高水平的多不饱和脂肪酸,并且比具有更高水平的单不饱和及饱和脂肪酸的油更易于氧化。高油酸大豆种子可以通过重组操作油酰12-脱水酶(fad2)的活性来制备。高油酸大豆油可用于需要高度氧化稳定性的应用中,例如在高温下长时间烹饪。
棉子糖在如下的许多经济上重要的作物物种的可食用部分中大量积累:例如大豆(soybean或glycinemaxl.merrill)、甜菜(sugarbeet或betavulgaris)、棉花(cotton或gossypiumhirsutuml.)、卡诺拉(油菜属(brassicasp.))以及所有的重要可食用豆科作物(包括豆类(菜豆属(phaseolussp.))、鹰嘴豆(chickpea或cicerarietinum)、豇豆(cowpea或vignaunguiculata)、绿豆(mungbean或vignaradiata)、豌豆(pea或pisumsativum)、小扁豆(lentil或lensculinaris)和羽扇豆(羽扇豆属(lupinussp.)))。虽然在许多物种中大量存在,但棉子糖妨碍了一些经济上重要的作物的有效利用。
参与棉子糖合成的酶(例如肌醇半乳糖苷合成酶)的表达的下调,将是期望的性状。
在某些实施例中,本披露考虑了用不止一个有利的转基因对受体细胞进行转化。可以使用不同的转基因编码载体或并入两种或更多种基因编码序列的单一载体在单一转化事件中供应两种或更多种转基因。可以根据需要使用任何描述的任何两种或更多种转基因,例如赋予除草剂、昆虫、疾病(病毒、细菌、真菌和线虫)或干旱抗性以及油量和油质的那些转基因,或增加产量或营养品质的那些转基因。
核小rna(snrna)可与蛋白质因子结合形成称为核小核糖核蛋白(snrnp)的rna-蛋白复合物。u1、u2、u4、u5和u6snrna是参与rna内含子加工的主要剪接体的组分。尽管其他上述的usnrna由与转录mrna相同的rna聚合酶ii转录,但植物u6snrna由需要鸟苷残基作为起始子和4个或更多个胸苷残基作为终止子的rna聚合酶iii转录。rna聚合酶iii对rna聚合酶ii特异性抑制剂d-鹅膏蕈碱(waibel和filipowicz,nar18:3451-3458(1990))不敏感。rna聚合酶iii的简单起始和终止要求使其启动子能够很好表达小rna分子,例如用于基因表达调节的小发夹rna(shrna)(wang等人,rna14:903-913(2008);kim和nam,plantmol.biol.rep.[植物分子生物学导报]31:581-593(2013)),和使用最近表征的细菌crispr(规律成簇的间隔短回文重复)系统用于基因组修饰的指导rna(grna)(barrangou等人,sci.[科学]315:1709-1712-2007);jinek等人,sci.[科学]337:816-821(2012);mali等人,sci.[科学]339:823-826(2013);cong等人,sci.[科学]339:819-823(2013))。
本披露涉及包含u6snrna基因启动子的重组dna构建体。本披露还涉及包含如下启动子的重组dna构建体,其中所述启动子基本上由seqidno:1、2、3、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、35、38、或40中所示核苷酸序列或seqidno:1、2、3、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、35、38、40的功能性片段组成。本披露还涉及包含如下启动子的分离的多核苷酸,其中所述启动子包含seqidno:1、2、3、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、35、38、40中所示核苷酸序列或seqidno:1、2、3、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、35、38或40的功能性片段。
从本文所阐述的披露内容可以看出,本领域普通技术人员可以进行以下程序:
1)将含有本文所述的u6启动子序列的核酸片段有效地连接到编码合适的功能性rna(例如但不限于crrna、tracerrna或单一指导rna)的核苷酸序列上。
2)将嵌合u6启动子-报道指导rna表达盒转化到合适的植物中以表达该启动子。存在本领域技术人员所熟知的可以用作转化宿主的各种合适的植物,包括双子叶植物(拟南芥、烟草、大豆、油菜、花生、向日葵、红花、棉花、西红柿、马铃薯、可可)和单子叶植物(玉米、小麦、水稻、大麦和棕榈)。
3)测试在各种细胞类型的转基因植物组织(例如叶、根、花、种子)中u6启动子的表达,所述转基因植物组织是通过测定报告基因产物的表达,用嵌合u6启动子:-报道基因表达盒进行转化的。
在另一个实施例中,本披露涉及包含如本文所述的本披露的重组dna构建体或如本文所述的本披露的分离的多核苷酸的宿主细胞。可用于实践本披露的宿主细胞的实例包括但不限于酵母、细菌和植物。
可以构建包含本发明重组dna构建体的质粒载体。质粒载体的选择取决于将用于转化宿主细胞的方法。本领域技术人员充分了解必须存在于质粒载体上的遗传元件,以便成功地转化、选择和繁殖含有嵌合基因的宿主细胞。
已经公开了主要通过使用根癌土壤杆菌转化如下双子叶植物和获得转基因植物的方法,例如棉花(美国专利号5,004,863,美国专利号5,159,135);大豆(美国专利号5,569,834,美国专利号5,416,011);芸苔属(美国专利号5,463,174);花生(cheng等人,plantcellrep.[植物细胞报道]15:653-657(1996),mckently等人,plantcellrep.[植物细胞报道]14:699-703(1995));木瓜(ling等人,bio/technology[生物/技术]9:752-758(1991));和豌豆(grant等人,plantcellrep.[植物细胞报道]15:254-258(1995))。对于其他常用的植物转化方法的综述参见如下文献:newell,c.a.,mol.biotechnol.[分子生物技术]16:53-65(2000)。这些转化方法之一使用发根土壤杆菌(tepfler,m.和casse-delbart,f.,microbiol.sci.[微生物科学]4:24-28(1987))。已经公开了采用如下手段使用dna的直接递送进行的大豆转化:peg融合(pct公开号wo92/17598)、电穿孔(chowrira等人,mol.biotechnol.[分子生物技术]3:17-23(1995);christou等人,proc.natl.acad.sci.u.s.a.[美国国家科学院院刊]84:3962-3966(1987))、显微注射、或粒子轰击(mccabe等人,biotechnology[生物技术]6:923-926(1988);christou等人,plantphysiol.[植物生理学]87:671-674(1988))。
有各种各样的方法用于从植物组织再生植物。特定的再生方法将取决于起始植物组织和待再生的特定植物种类。来自单一植物原生质体转化体或来自各种转化的外植体的植物的再生、发育和培养是本领域所熟知的(weissbach和weissbach编辑;methodsforplantmolecularbiology[植物分子生物学方法];academicpress,inc.:sandiego,ca,1988[学术出版社有限公司:圣地牙哥,加州,1988])。这种再生和生长过程通常包括如下步骤:选择经转化的细胞,通过胚性发育的通常阶段或通过生根苗阶段培养那些个体化细胞。以同样的方式再生转基因胚和种子。然后将所得的转基因生根芽苗种植在合适的植物生长培养基(如土壤)中。优选地,再生植物自花授粉以提供纯合的转基因植物。或者,将得自再生植物的花粉与农学上重要的品系的产生种子的植物进行杂交。相反地,将来自这些重要品系的植物的花粉用于给再生植物授粉。使用本领域技术人员熟知的方法培养含有所期望的多肽的本披露的转基因植物。
除了上述讨论的程序之外,从业者熟悉描述大分子(例如dna分子、质粒等)的构建、操作和分离,重组dna片段和重组表达构建体的产生,以及克隆的筛选和分离的具体条件和程序的标准资源材料(参见例如,sambrook,j.等人,molecularcloning:alaboratorymanual[分子克隆:实验手册];第二版;coldspringharborlaboratorypress:coldspringharbor,newyork,1989[冷泉港实验室出版社:冷泉港,纽约,1989];maliga等人,methodsinplantmolecularbiology[植物分子生物学方法];coldspringharborpress,1995[冷泉港出版社,1995];birren等人,genomeanalysis:detectinggenes,1[基因组分析:检测基因,1];coldspringharbor:newyork,1998[冷泉港:纽约,1998];birren等人,genomeanalysis:analyzingdna,2[基因组分析:分析dna,2];coldspringharbor:newyork,1998[冷泉港:纽约,1998];clark编辑,plantmolecularbiology:alaboratorymanual[植物分子生物学:实验室手册];springer:newyork,1997[斯普林格:纽约,1997])。
技术人员还将认识到,不同的独立转化事件将导致不同水平和模式的嵌合基因表达(jones等人,emboj[欧洲分子生物学学会杂志]4:2411-2418(1985);dealmeida等人,molgengenetics[分子遗传学和普通遗传学]218:78-86(1989))。因此,必须筛选多个事件以便获得显示所期望的表达水平和模式的品系。这种筛选可以通过mrna表达的northern分析、蛋白质表达的western分析或表型分析来完成。还感兴趣的是从显示所期望的基因表达谱的经转化的植物获得的种子。
u6启动子的活性水平弱于许多已知的强启动子,如camv35s启动子(atanassova等人,plantmol.biol.[植物分子生物学]37:275-285(1998);battraw和hall,plantmol.biol.[植物分子生物学]15:527-538(1990);holtorf等人,plantmol.biol.[植物分子生物学]29:637-646(1995);jefferson等人,emboj.[欧洲分子生物学学会杂志]6:3901-3907(1987);wilmink等人,plantmol.biol.[植物分子生物学]28:949-955(1995))、拟南芥泛素延伸蛋白启动子(callis等人,j.biol.chem.[生物化学杂志]265(21):12486-12493(1990))、西红柿泛素基因启动子(rollfinke等人,gene[基因]211:267-276(1998))、大豆热休克蛋白启动子以及玉米h3组蛋白基因启动子(atanassova等人,plantmol.biol.[植物分子生物学]37:275-285(1998))。
可以使用本领域技术人员所熟知的方法(包括但不限于本文所述的方法)来实现转化和选择。
在此披露的方法和组合物的非限制性实例如下:
1.包含有效地连接至至少一个异源核苷酸序列的如下核苷酸序列的重组dna构建体,所述核苷酸序列包含seqidno:1、seqidno:2、seqidno:3、seqidno:4、seqidno:6、seqidno:8、seqidno:10、seqidno:12、seqidno:14、seqidno:16、seqidno:18、seqidno:20、seqidno:22、seqidno:24、seqidno:26、seqidno:28、seqidno:30、seqidno:32、seqidno:34、seqidno:36、seqidno:38、seqidno:40中所示序列中任何一个或其功能性片段,其中所述核苷酸序列是启动子。
2.如实施例1所述的重组dna构建体,其中当与seqidno:1、seqidno:2、seqidno:3、seqidno:4、seqidno:6、seqidno:8、seqidno:10、seqidno:12、seqidno:14、seqidno:16、seqidno:18、seqidno:20、seqidno:22、seqidno:24、seqidno:26、seqidno:28、seqidno:30、seqidno:32、seqidno:34、seqidno:36、seqidno:38或seqidno:40中所示序列中任何一个比较时,基于clustalv比对方法,使用逐对比对默认参数(ktuple=2、空位罚分=5、窗口=4和存储的对角线=4),所述核苷酸序列具有至少95%同一性。
3.包含如实施例1所述的重组dna构建体的载体。
4.包含如实施例1所述的重组dna构建体的细胞。
5.如实施例4所述的细胞,其中该细胞是植物细胞。
6.转基因植物,该转基因植物已经在其基因组中稳定地并入了如实施例1所述的重组dna构建体。
7.如实施例6所述的转基因植物,其中所述植物是双子叶植物。
8.如实施例7所述的转基因植物,其中该植物是大豆。
9.由实施例7所述的转基因植物产生的转基因种子,其中该转基因种子包含重组dna构建体。
10.如实施例1所述的重组dna构建体,其中该至少一个异源序列编码选自由crrna、tracrrna和指导rna组成的组的功能性rna分子。
11.如实施例1所述的重组dna构建体,其中该至少一种异源序列编码能够形成指导rna/cas内切核酸酶复合物的单一指导rna,其中所述指导rna与dna靶位点杂交。
12.在植物中表达功能性rna的方法,该方法包括:
a)将如实施例1所述的重组dna构建体引入植物中,其中该至少一个异源序列编码功能性rna;
b)使步骤a)的植物生长;和
c)选择显示了该重组dna构建体的该功能性rna的表达的植物。
13.如实施例12所述的方法,其中该植物为双子叶植物。
14.如实施例12所述的方法,其中该植物为大豆植物。
15.用重组dna构建体稳定转化的植物,该重组dna构建体包含大豆u6聚合酶iii启动子和有效地连接至所述u6聚合酶iii启动子上的异源核酸片段,其中所述u6聚合酶iii启动子能够在植物细胞中控制所述异源核酸片段的表达,并且此外其中所述u6聚合酶iii启动子包含seqidno:1、seqidno:2、seqidno:3、seqidno:4、seqidno:6、seqidno:8、seqidno:10、seqidno:12、seqidno:14、seqidno:16、seqidno:18、seqidno:20、seqidno:22、seqidno:24、seqidno:26、seqidno:28、seqidno:30、seqidno:32、seqidno:34、seqidno:36、seqidno:38、seqidno:40中所示序列中任何一个。
16.重组dna构建体,其包含驱动编码指导多核苷酸、crrna或tracrrna的异源核酸片段的大豆u6聚合酶iii启动子的重组dna构建体,其中所述启动子包含seqidno:1、seqidno:2、seqidno:3、seqidno:4、seqidno:6、seqidno:8、seqidno:10、seqidno:12、seqidno:14、seqidno:16、seqidno:18、seqidno:20、seqidno:22、seqidno:24、seqidno:26、seqidno:28、seqidno:30、seqidno:32、seqidno:34、seqidno:36、seqidno:38、seqidno:40中所示序列中任何一个或其功能片段。
17.用于修饰植物细胞基因组中的靶位点的方法,该方法包括将指导rna和cas内切核酸酶引入所述植物细胞中,其中所述指导rna和cas内切核酸酶能够形成使cas内切核酸酶在所述靶位点处引入双链断裂的复合物,其中所述指导rna由包含如下启动子的重组dna构建体表达,该启动子包含seqidno:1、seqidno:2、seqidno:3、seqidno:4、seqidno:6、seqidno:8、seqidno:10、seqidno:12、seqidno:14、seqidno:16、seqidno:18、seqidno:20、seqidno:22、seqidno:24、seqidno:26、seqidno:28、seqidno:30、seqidno:32、seqidno:34、seqidno:36、seqidno:38、seqidno:40中所示序列中任何一个或其功能片段。
18.重组dna构建体,其包含驱动编码指导多核苷酸的异源核酸片段的大豆u6聚合酶iii启动子,该重组dna构建体包含:(i)与靶dna中的核苷酸序列互补的第一核苷酸序列区域;和(ii)与cas内切核酸酶相互作用的第二核苷酸序列区域,其中该第一核苷酸序列区域和该第二核苷酸序列区域由脱氧核糖核酸(dna)、核糖核酸(rna)或其组合构成,其中该指导多核苷酸不仅仅包含核糖核酸。
实例
本披露在以下实例中进一步定义,除非另有说明,其中份数和百分数以重量计,并且度数以摄氏度计。除非另有描述,否则本披露中列出的启动子、cdna、衔接子和引物的序列均处于5′至3’方向。分子生物学技术通常如以下文献中所述地进行:ausubel,f.m.等人,currentprotocolsinmolecularbiology[分子生物学现代方法];约翰·威利父子出版公司(johnwileyandsons):纽约,1990或sambrook,j.等人,molecularcloning:alaboratorymanual[分子克隆:实验室手册];第二版;coldspringharborlaboratorypress:coldspringharbor,newyork,1989[冷泉港实验室出版社:冷泉港,纽约,1989](在下文中“sambrook等人,1989”)。应该理解,尽管这些实例说明了本披露的优选实施例,但仅以示例的方式给出。从以上的讨论和这些实例中,本领域的技术人员能够确定本披露的本质特性,并且在不脱离本披露的精神和范围的情况下,可进行本披露的各种变化和修改以使其适应各种用途和条件。因此,从上述说明书来看,除了本文所示出和描述的那些之外,本披露的各种修改对于本领域技术人员来说是显而易见的。此类修改也旨在落入所附权利要求的范围内。
本文所示的每个参考文献的披露内容通过引用整体并入本文。
实例1
大豆u6核小rna基因的鉴定
核小rna(snrna)(如u1、u2、u4、u5和u6)是参与rna内含子加工的主要剪接体的组分。它们与蛋白质因子结合形成称为snrnp(核小核糖核蛋白)的rna-蛋白复合物。虽然其他上述的usnrna由也转录编码蛋白质的mrna的rna聚合酶ii转录,但是植物u6snrna由用鸟苷残基开始转录和通过4个或更多个胸苷残基终止转录的rna聚合酶iii转录(waibel和filipowicz,nucleicacidsres.[核酸研究]18:3451-3458(1990);li等人,j.integrateplantbiol.[植物生态学报]49:222-229(2007);nielsen等人,science[科学]340:1577-1580)。rna聚合酶iii还合成trna、5srrna和其他类型的细胞质和核小rna。rna聚合酶iii对rna聚合酶ii特异性抑制剂α-鹅膏蕈碱不敏感的事实方便地用于确定启动子是否依赖于rna聚合酶iii。
简单的起始和终止要求使得rna聚合酶iii启动子对于精确表达小rna分子是理想的,该小rna分子是例如通过rna干扰进行基因沉默的小发夹rna(shrna)(wang等人,rna14:903-913(2008);kim和nam,plantmol.biol.rep.[植物分子生物学导报]31:581-593(2013))和用于cas9/grna介导的基因组修饰的指导rna(grna)(jinek等人,science[科学]337:816-821(2016);mali等人,science[科学]339:823-826(2013);cong等人,science[科学]339:819-823(2013))。
u6rna约为100个核苷酸长并且跨物种高度保守。已经表征了几种拟南芥属和蒺藜状苜蓿u6基因的启动子(li等人,j.integrateplantbiol.[植物生态学报]49:222-229(2007);kim和nam,plantmol.biol.rep.[植物分子生物学报道]31:581-593(2013))。一个拟南芥u6基因atu6-1用于blast搜索大豆品种williams82的基因组序列(schmutz等人,nature[自然]463:178-183(2010))。
如下所述,来自大豆的18个个体u6snrna同源物被鉴定为位于10个不同的染色体上。u6snrna同源物中有11个是高度保守的,其中102bpu6编码序列中具有不超过2bp的偏差。为了帮助轻松鉴别出同系物,根据它们所在的染色体对它们进行命名。例如,gm-u6-9.1表示染色体9上的一个大豆(glycinemax)u6基因。因此,将18个大豆u6基因命名为gm-u6-1.1、gm-u6-4.1、gm-u6-4.2、gm-u6-6.1、gm-u6-6.1、gm-u6-9.1、gm-u6-12.1、gm-u6-13.1、gm-u6-14.1、gm-u6-15.1、gm-u6-15.2、gm-u6-16.1、gm-u6-16.2、gm-u6-19.1、gm-u6-19.2、gm-u6-19.3、gm-u6-19.4、gm-u6-19.5、和gm-u6-19.6。将每个u6snrna基因的转录序列列出如下:gm-u6-1.1snrna,seqidno:5;gm-u6-4.1snrna,seqidno:7;gm-u6-4.2snrna,seqidno:9;gm-u6-6.1snrna,seqidno:11;gm-u6-9.1snrna,seqidno:13;gm-u6-12.1snrna,seqidno:15;gm-u6-13.1snrna,seqidno:17gm-u6-14.1snrna,seqidno:19;gm-u6-15.1snrna,seqidno:21;gm-u6-15.2snrna,seqidno:23;gm-u6-16.1snrna,seqidno:25;gm-u6-16.2snrna,seqidno:27;gm-u6-19.1snrna,seqidno:29;gm-u6-19.2snrna,seqidno:31;gm-u6-19.3snrna,seqidno:33;gm-u6-19.4snrna,seqidno:35;gm-u6-19.5snrna,seqidno:37;gm-u6-19.6snrna,seqidno:39,并且将每个u6snrna基因的预测启动子序列列出如下:gm-u6-1.1snrna启动子,seqidno:6;gm-u6-4.1snrna启动子,seqidno:8;gm-u6-4.2snrna启动子,seqidno:10;gm-u6-6.1snrna启动子,seqidno:12;gm-u6-9.1snrna启动子,seqidno:14;gm-u6-12.1snrna启动子,seqidno:16;gm-u6-13.1snrna启动子,seqidno:18gm-u6-14.1snrna启动子,seqidno:20;gm-u6-15.1snrna启动子,seqidno:22;gm-u6-15.2snrna启动子,seqidno:24;gm-u6-16.1snrna启动子,seqidno:26;gm-u6-16.2snrna启动子,seqidno:28;gm-u6-19.1snrna启动子,seqidno:30;gm-u6-19.2snrna启动子,seqidno:32;gm-u6-19.3snrna启动子,seqidno:34;gm-u6-19.4snrna启动子,seqidno:36;gm-u6-19.5snrna启动子,seqidno:38;gm-u6-19.6snrna启动子,seqidno:40。
通过使用vectorntialignx(英杰公司(invitrogen))的序列比对来比较包括更多样化的近端启动子区域的18个大豆u6snrna基因。基于其序列相似性,根据系统发育树对其进行分组(图1)。18个snrna基因的近端启动子区域序列的比对鉴定了大多数u6snrna基因(图2)中的保守性上游序列元件(在图2中标记为use)和tata框(在图2中标记为use)。u6snrna转录需要use和tata元件。由于u6启动子还含有较不保守的调节元件,例如在tata框或use元件更靠上游的远端序列元件(dse)和近端序列元件(pse),因此可能需要转录起始位点上游多达约500bp的序列来保持完整的u6启动子功能。此外,相对于彼此或转录起始对这些元件进行适当定位也可以发挥作用(waibel和filipowicz,nucleicacidsres.[核酸研究]18:3451-3458(1990);kim和nam,plantmol.biol.rep.[植物分子生物学导报]31:581-593(2013))。
实例2
大豆u6snrna基因启动子的分离
选择4个u6基因gm-u6-16.1、gm-u6-16.2、gm-u6-13.1和gm-u6-9.1,用于启动子克隆和评价。选择u6snrna转录起始位点gtccct上游的dna序列的大约500bp的基因组片段作为推定的启动子片段,并在5′末端工程改造限制性位点xmaicccggg以方便克隆,得到seqidno:1、2、3、4的启动子序列。gm-u6-9.1和gm-u6-13.1启动子在3′末端均具有hindiii位点,而gm-u6-16.1在3′末端附近具有mfeicaattg,并且gm-u6-16.2在其3′末端附近具有ndei位点catatg。这些限制性位点便于随后克隆这些启动子,以使用crispr系统表达不同的小rna,例如用于靶向基因组修饰的不同的grna。
指导rna/cas内切核酸酶复合物的一个组分是可以由单一指导rna分子组成,或者可以包含嵌合crrna和tracrrna的杂合体的指导rna。引物rna(grna)可以与cas内切核酸酶形成功能性复合物,其中该指导rna将cas内切核酸酶指导到基因组靶位点上。
已经鉴定了许多大豆基因组靶位点,包括染色体4上的dd43-cr1(seqidno:41)。全部四种所选择的u6基因启动子gm-u6-9.1、gm-u6-13.1、gm-u6-16.1和gm-u6-16.2均用于表达dd43-cr1grna,以测试并比较它们的启动子活性。小u6启动子:grna表达盒是由金斯瑞公司(genscript)(genscriptusainc.,860centennialave,piscataway,nj08854,usa[美国金斯瑞有限公司,美国新泽西州(08854)皮斯卡塔韦,纪念大街860号])合成的,并通过限制性消化和连接与植物转化选择标记基因盒35s:hpt连接。
实例3
小rna基因表达构建体和大豆转化
当合成gm-u6-13.1:dd43cr1、gm-u6-16.1:dd43cr1、gm-u6-9.1:dd43cr1和gm-u6-16.2:dd43cr1grna表达盒时,单一限制性位点xmai和noti分别在5’末端和3’末端被工程改造。将上述盒克隆到35s:hpt:pinii植物转化选择标记基因载体的xmai和noti位点上以产生dna构建体qc795、qc796、qc797和qc798(表1)。qc795a的连接的表达盒gm-u6-13.1:dd43-cr1+camv-35spro:hpt:piniiterm的序列(图3a)被列为seoidno:57。qc796a、qc797a和qc798a的连接盒的序列与qc795a仅在u6启动子上不同,即qc796a(seqidno:3)中的gm-u6-16.1、qc797a(seqidno:1)中的gm-u6-9.1和qc798a(seqidno:4)中的gm-u6-16.2。
表1.受控于不同的u6核小rna基因启动子的dd43-cr1grna的转化。
将连接的gm-u6-13.1:dd43-cr1+camv-35spro:hpt:piniiterm盒用asci消化从质粒qc795作为dna片段释放、通过琼脂糖凝胶电泳与载体主链片段分离、并且用dna凝胶提取试剂盒(凯杰公司(qiagen))从该凝胶中纯化。类似地,连接的gm-u6-16.1:dd43-cr1+camv-35spro:hpt:piniiterm、u6-9.1:dd43-cr1+camv-35spro:hpt:piniiterm和u6-16.2:dd43-cr1+camv-35spro:hpt:piniiterm盒分别从质粒qc796、qc797和qc798纯化而来。将纯化的dna片段通过如下详述的粒子枪轰击法(klein等人,nature[自然]327:70-73(1987);美国专利号4,945,050)转化到大豆栽培品种jack中,以研究稳定转化的大豆植物中的u6启动子。
可以将如上文概述的用于gm-u6-13.1:dd43-cr1表达盒构建和转化的相同的方法与其他异源核酸序列(包括靶向不同基因组位点的grna、小发夹rna(shrna)和人造微rna(amirna))一起用于基因表达调节和沉默。
如下诱导来自jack栽培品种的大豆体细胞胚。从表面灭菌的未成熟种子中切下子叶(约3mm长),并在含有0.7%琼脂并补充有10mg/ml2,4-d(2,4-二氯苯氧乙酸)的murashige和skoog(ms)培养基上,在26℃的光照下培养6-10周。然后切下产生次生胚的球状期体细胞胚,并置于含有补充有2,4-d(10mg/ml)的液体ms培养基的烧瓶中,并在旋转振荡器上在光照下培养。在重复选择繁殖为早期的球状期胚的体细胞胚簇之后,在26℃下的16∶8小时(照明/黑暗)的荧光灯方案下,将大豆胚性悬浮培养物保持在150rpm的旋转振荡器上的35ml的液体培养基中。通过将大约35mg的组织接种到35ml相同的新鲜液体ms培养基中,每两周将培养物传代培养。
然后使用杜邦(dupont)biolistictmpds1000/he仪器(伯乐实验室(bio-radlaboratories),赫拉克勒斯,加利福尼亚州(hercules,ca))通过粒子枪轰击法转化大豆胚性悬浮培养物。向50μl的60mg/ml的1.0mm的金色颗粒悬浮液中依次添加以下各项:30μl的30ng/μl的qc795adna片段gm-u6-13.1:dd43-cr1+camv-35spro:hpt:piniiterm或qc796、qc797a、qc798adna片段,20μl的0.1m的亚精胺,以及25μl的5mcacl2。然后将颗粒制剂搅拌3分钟,在离心机中旋转10秒,并且除去上清液。然后将dna包被的颗粒在400μl100%乙醇中洗涤一次,并重悬于45μl100%乙醇中。将dna/颗粒悬浮液超声处理3次,每次1秒。然后将5μl的dna包被的金色颗粒加载到每个宏载体盘(macrocarrierdisk)上。
将约300-400mg的两周龄悬浮培养物置于空的60×15mm的培养皿中,并用移液管从组织中除去残留的液体。对于每个转化实验,轰击大约5至10板的组织。膜破裂压力设定在1100psi,并将该箱抽至28英寸汞柱的真空。将组织放置在距离挡板约3.5英寸处并且轰击一次。轰击后,将该组织分成两半并放回液体培养基中,并如上所述进行培养。
轰击后5至7天,将液体培养基更换为含有作为选择剂的30μg/ml的潮霉素的新鲜培养基。每周更新这种选择性培养基。轰击后7至8周,观察到,从未经转化的、坏死的胚性簇生长为绿色的、经转化的组织。去除分离的绿色组织并接种到单个烧瓶中以产生新的、克隆繁殖的、经转化的胚性悬浮培养物。将每个克隆繁殖的培养物作为独立转化事件处理,并在补充有2,4-d(10mg/ml)和30μg/ml的潮霉素选择剂的相同液体ms培养基中传代培养,以增加质量。然后将胚性悬浮培养物转移到没有补充2,4-d的琼脂固体ms培养基板上,以允许体细胞胚发育。在此阶段收集每个事件的样本进行定量pcr分析。从四个转化项目中产生的转基因事件数列于表1中。
干燥子叶期体细胞胚(通过将它们转移到位于含有一些琼脂凝胶的10cm的培养皿顶部的空的小培养皿中,以允许缓慢干燥)以模拟大豆种子发育的最后阶段。将干燥的胚置于发芽固体培养基上,并使转基因大豆小植株再生。然后将转基因植物转移到土壤中并保持在用于种子生产的生长箱中。
使用dneasy96植物试剂盒(凯杰公司(qiagen))从体细胞胚样品中提取基因组dna,并使用具有基因特异性引物和fam标记的荧光探针的具有7500实时pcr系统(应用生物系统公司(appliedbiosystems))的taqman通用pcr主要混合物进行定量pcr分析,以检查u6启动子:dd43-cr1grna(seqidno:42、43、44)和35s:hpt(seqidno:45、46、47)表达盒的拷贝数(图3a)。qpcr分析是在与作为内源对照的热休克蛋白(hsp)基因(seqidno:48、49、50)和作为校准品的具有已知的35s:hpt转基因的单拷贝的转基因dna样品的双链体反应中进行的。将内源性对照hsp探针用vic标记并将靶基因u6启动子:dd43-cr1grna或35s:hpt探针用fam标记,用于同时检测两种荧光探针(应用生物系统公司(appliedbiosystems))。在50℃孵育2分钟以活化taqdna聚合酶,并在95℃孵育10分钟以使dna模板变性,在这之后,进行40个循环:在95℃下15秒和60℃下1分钟。使用装备有7500实时pcr系统的序列检测软件捕获并分析pcr反应数据,并使用相对定量方法(应用生物系统公司(appliedbiosystems))计算基因拷贝数。通过grnaqpcr测定确定的qc795a、qc796a、qc797a和qc798a转基因事件的转基因拷贝数由代表该事件的每列下的数字表示(图4a-4d)。
qrt-pcr分析中的对照。
图4a-4d中的每一列表示独立事件,其中qpcr确定了所指示的转基因dd43cr1grna盒的拷贝数。a.gm-u6-13.1启动子。b.gm-u6-16.1启动子。c.gm-u6-9.1启动子。d.gm-u6-16.2启动子。
实例4
具有不同的u6启动子的dd43-cr1grna的表达
u6启动子活性由四个项目的不同转基因事件中的dd43-cr1grna的表达水平来反映(表1)。采用
使用与grnaqpcr相同的引物和fam标记的探针通过qrt-pcr检查dd43-cr1grna的表达(seqidno:42、43、44)。使用组成型表达的内源基因atp硫酸化酶(atps)作为内源对照,以使负载引物和vic标记的探针的样品标准化(seqidno:54、55、56)。将每个样品中dd43-cr1grna的相对表达与掺有野生型基因组dna的qc819相同的任意校准品进行比较。重复三次在单项反应(singleplexreaction)中进行qrt-pcr分析以补偿变异。在50℃孵育30分钟以合成第一链cdna并在95℃孵育10分钟以使dna模板变性,在这之后,进行40个循环:在95℃下15秒和60℃下1分钟。使用装备有7500实时pcr(应用生物系统公司(appliedbiosystems))的软件捕获并计算pcr分析数据以进行相对定量。
在所有转基因事件中,通过qrt-pcr检测到dd43-cr1grna表达,只有少数例外,但不包括所有四个项目(具有qc795a的gm-u6-13.1d43(图4a)、具有qc796a的gm-u6-16.1d43(图4b)、具有qc797a的gm-u6-9.1d43(图4c)和具有qc799a的gm-u6-16.2d43(图4d))的任何非转基因事件,表明所有四种启动子(gm-u6-16.1、gm-u6-16.2、gm-u6-13.1和gm-u6-9.1)都是功能性的并且能够表达指导rna。非转基因事件由以前完成的grna特异性qpcr测定的0.0或<0.3拷贝的转基因指示。如果含有1拷贝或2拷贝的转基因的事件的质量较好,则在同一项目的转基因事件中会观察到大量变异。但是,每个数据列顶部的相对较短的标准偏差线(图4a-4d)所示的相同事件的三个重复中的变异是合理范围内小的。这些结果表明,所有四种u6启动子都是功能性的。由于gm-u6-13.1(seqidno:2)、gm-u6-16.1(seqidno:3)、gm-u6-9.1(seqidno:1)和gm-u6-16.2(seqidno:4)的经测试的启动子仅包含在大豆中检测到的天然启动子的片段,因此预期更长版本的启动子gm-u6-13.1(seqidno:18)、gm-u6-16.1(seqidno:26)、gm-u6-9.1(seqidno:14)和gm-u6-16.2(seqidno:28)将与短的版本的启动子(seqidno:1、2、3、4)具有类似的活性。还预期其他u6snrna基因gm-u6-1.1(seqidno:12)、gm-u6-12.1(seqidno:16)、gm-u6-14.1(seqidno:20)、gm-u6-15.1(seqidno:22)、gm-u6-15.2(seqidno:24)、gm-u6-19.1(seqidno:30)、gm-u6-19.2(seqidno:32)、gm-u6-19.3(seqidno:34)、gm-u6-19.4(seqidno:36)、gm-u6-19.5(seqidno:38)和gm-u6-19.6(seqidno:40)的启动子也可能被克隆以表达转基因小rna(如grna)。这些u6启动子中的一些如gm-u6-4.2、gm-u6-12.1、gm-u6-19.3和gm-u6-19.6可能不像其他启动子一样有活性,因为它们缺乏高度保守的use或tata框(图2)。
实例5
用于靶基因组位点修饰的cas9和grna构建体
通过其表达功能性dd43-cr1grna的能力进一步评估u6启动子功能,这与化脓性链球菌cas9内切核酸酶的同步表达相组合,导致靶基因组位点修饰。将qc797a的相同的gm-u6-9.1:dd43-cr1grna盒与gm-ef1a2:cas9(so)盒连接以制备dna构建体qc799(seqidno:59,图5a)。gm-ef1a2启动子是从大豆延伸因子基因中分离的强组成型启动子(美国专利号8,697,857)。cas9(so)是用于在编码化脓性链球菌cas9内切核酸酶的大豆中表达的密码子优化基因。将针对大豆表达进行了密码子优化的sv40核定位信号(nls)添加到cas9羧基末端以帮助将蛋白质运送到细胞核。功能性dd43-cr1grna和cas9可以形成grna-cas9核糖核酸蛋白(riboprotein)复合物,以识别和切割大豆基因组中的dd43靶位点(seqidno:41),以产生双链dna断裂,该断裂将通过非同源末端连接(nhej)或同源重组(hr)自发修复。在nhej或hr期间,可将突变或异源dna引入切割位点。
构建了含有gm-samspro-frt1:hpt-frt87盒的供体dna构建体rtw831,其两侧侧翼分别为约1kb的基因组序列gm-dd43frag1和gm-dd43frag2,用于转基因事件选择(seqidno:60,图5b)。从作为中等组成型启动子的大豆s-腺苷-甲硫氨酸合成酶基因中分离出gm-sams启动子(美国专利号7,217,858)。将frt1和frt87位点并入构建体中,使得hpt基因一旦整合到大豆基因组中仍然可以通过重组酶介导盒交换(rmce)被其他转基因替换(li等人,plantphysiol.[植物生理学]151:1087-1095,(2009))。侧翼dna片段gm-dd43frag1和gm-dd43frag2(与大豆基因组中dd43-cr1位点相邻的序列同一)是促进dna同源重组以将供体盒整合到dd43-cr1位点中的同源序列。
类似地制备了qc799a和rtw831adna片段(图5a-5b),并按照与实例3中所述的相同方法,以相等的量用来共同转化大豆栽培品种93b86。使用dneasy96植物试剂盒(凯杰公司(qiagen))对转基因事件的年轻体细胞胚簇进行取样,用于基因组dna提取,并且与实例3中所述类似地进行qpcr分析。使用引物(seqidno:61、63)和fam标记的探针(seqidno:62)通过qpcr检查rtw831a转基因拷贝数。使用引物(seqidno:64、66)和fam标记的探针(seqidno:65)通过qpcr检查qc799a转基因拷贝数。使用引物(seqidno:67、69)和fam标记的探针(seqidno:68)通过qpcr检查内源性dd43-cr1靶位点的拷贝数。在所有上述qpcr测定的双链体反应中,包含使用引物(seqidno:48、50)和vic标记的探针(seqidno:49)进行的相同的hsp内源对照qpcr。
实例6
采用u6启动子表达的grna引导的基因组位点修饰
野生型大豆基因组dna含有dd43-cr1靶位点(seqidno:41)的两个拷贝,两个同源染色体中每个上各一个拷贝。如果由实例5所述的qpcr测定的dd43-cr1拷贝数减少,则衍生自qc799a+rtw831a转化的任何转基因事件都可能在dd43-cr1位点上被修饰。由于正向引物dd43-f(seqidno:67)和探针dd43-t(seqidno:68)序列与dd43-cr1序列部分重叠,采用引物(seqidno:67、69)和fam标记的探针(seqidno:68)的dd43-cr1特异性qpcr不扩增经修饰的dd43-cr1位点。
如果通过qpcr检测到dd43-cr1靶位点的两个拷贝,那么该位点未被修饰并仍为野生型(wt-homo)。如果仅检测到dd43-cr1的一个拷贝或没有检测到拷贝,那么该位点的一个拷贝(nhej-hemi)或两个拷贝(nhej-无效)被nhej修饰。如表2所总结的,共产生了263个转基因事件,其中53个事件(20.2%)保留了dd43-cr1野生型序列,88个事件(33.5%)具有经修改的dd43-cr1位点的一个拷贝,以及122个事件(46.4%)具有经修改的dd43-cr1位点的两个拷贝。通过由dd43-cr1grna指导的cas9内切核酸酶切割的dna双链断裂引发的nhej,在dd43-cr1位点修饰了多达80%的总事件。表2.采用gm-u6-9.1启动子表达的dd43-cr1grna介导的基因组位点修饰。
通过pcr从野生型和转基因事件扩增基因组dd43-cr1区域,用于序列分析,以评估dd43-cr1靶位点修饰。引物dd43-lb在gm-dd43frag1的上游,并且dd43-rb位于gm-dd43frag2的下游,所以具有dd43-lb和dd43-rb引物的pcr不会扩增来自rtw831a转基因的任何物质(图5b)。pcr循环条件为94℃持续4分钟;在使用铂金高保真taqdna聚合酶(英杰公司(invitrogen))保持在4℃前,进行35个循环:94℃持续30秒,60℃持续1分钟,和68℃持续2分钟;以及最后在68℃持续5分钟。使用琼脂糖凝胶电泳解析pcr反应以鉴定表示预期的2098bp的dd43-cr1区域(seqidno:74)的pcr产物的正确大小。通过ta克隆(英杰公司(invitrogen))将pcr片段克隆到pcr2.1-topo载体中,并对多个克隆进行测序。
dd43-cr1位点修饰大多数发生在pam序列cgg上游3bp的预期的cas9切割位点处(seqidno:75-103,图6)。观察到dd43-cr1位点的小缺失和切割位点处的短插入。结果表明,gm-u6-9.1启动子能够有效表达功能性dd43-cr1grna,以指导位点特异性基因组修饰。其他gm-u6启动子如gm-u6-13.1、gm-u6-16.1和gm-u6-16.2也成功地用于有效表达靶向不同基因组位点的各种grna。由于许多其他大豆u6基因启动子区域含有相似的调节元件,因此预期其他gm-u6启动子虽然尚未被类似地表征但也可用于表达转基因小rna基因。
序列表
<110>e.i.内穆尔杜邦公司
li,zhongsen
<120>大豆u6核小rna基因启动子及其在植物小rna基因的组成型表达中的用途
<130>bb2332pct
<150>62/139,075
<151>2015-03-27
<160>103
<170>patentin3.5版
<210>1
<211>472
<212>dna
<213>大豆
<400>1
cccgggttaagagaattgtaagtgtgcttttatatatttaaaattaatatattttgaaat60
gttaaaatataaaagaaaattcaatgtaaattaaaaataaataaatgtttaataaagata120
aattttaaaacataaaagaaaatgtctaacaagaggattaagatcctgtgctcttaaatt180
tttaggtgttgaaatcttagccatacaaaatatattttattaaaaccaagcatgaaaaaa240
gtcactaaagagctatataactcatgcagctagaaatgaagtgaagggaatccagtttgt300
tctcagtcgaaagagtgtctatctttgttcttttctgcaaccgagttaagcaaaatggga360
atgcgaggtatcttcctttcgttaggggagcaccagatgcatagttagtcccacattgat420
gaatataacaagagcttcacagaatatatagcccaggccacagtaaaagctt472
<210>2
<211>440
<212>dna
<213>大豆
<400>2
cccgggtgtgatttagtataaagtgaagtaatggtcaaaagaaaaagtgtaaaacgaagt60
acctagtaataagtaatattgaacaaaataaatggtaaagtgtcagatatataaaatagg120
ctttaataaaaggaagaaaaaaaacaaacaaaaaataggttgcaatggggcagagcagag180
tcatcatgaagctagaaaggctaccgatagataaactatagttaattaaatacattaaaa240
aatacttggatctttctcttaccctgtttatattgagacctgaaacttgagagagataca300
ctaatcttgccttgttgtttcattccctaacttacaggactcagcgcatgtcatgtggtc360
tcgttccccatttaagtcccacaccgtctaaacttattaaattattaatgtttataacta420
gatgcacaacaacaaagctt440
<210>3
<211>421
<212>dna
<213>大豆
<400>3
cccgggtgccagaagtatttcagtttattttgaaaaatcagaaaaaaaatgtctggaata60
aaatataataagcgatactaataaataattgaacaagataaatggtaaaatgtcaaatca120
aaactaggctacagagtgcagagcagagtcatgatgaatgacagctagttctacttacta180
caccgattcttgtgtacataaaaatattttaaaataattgaatctttctttagccagctt240
tgacaacaatgtacaccgttcgtacttcttactggtaggcaatgcttcttgtttgctttc300
ggtggaaggtgtatatactcaacattacttctttttcagcgtgttttcttacgggagtcc360
cacaccgcccaaaactaatacagtattcttgtttataaagaagtgcaccacttcaattgt420
t421
<210>4
<211>487
<212>dna
<213>大豆
<400>4
cccgggtgcaaatacaacagtttgaaaataaactgctaagttgtgaatctcaccactaaa60
atctattaaaatataattttaaaagataaatttgttattcaataataggcttttaaatat120
aggaaaatgcataatttaactaattaaagaaaataaaaatgcaagtgcggtgacaagaca180
agctagaataaagttgcaaagaaatgacagggctacaaaaggctcacctacttctggatt240
taccaaacttctgtttgtccccatactccaaaaacaaaaccatttttttttatcttcgtt300
tttgtttgctttgactgtgagttgaggcccaactttctgcttctgtccgactctatttga360
tgaattttgtttgcctcctgtgatgtgaaggatgtatcattgaaagggaacgtgtctcaa420
tgatcccacatcggccaaatatgctcattacattgcgtttatatagtcccaggaaaacat480
atggatt487
<210>5
<211>89
<212>dna
<213>大豆
<400>5
gtcccttcggggacatccgaggaaattgaaagcatggcccttgcgcaaggatgacacgct60
tttatcaagaaatggtccaatttattttt89
<210>6
<211>956
<212>dna
<213>大豆
<400>6
aatacaaagaaacaaaacctaaggaatcctaaaggaggaatttgagagaggttaagtcct60
gtgtcttgagagaaaaataatataatatggaattatctcaattctaagaatacggtcttt120
atctttaattactagtagtatataggatggaattttctatatgtaaaactaaatttggat180
tcatttgacaataatattatgcaggcacggaattcttcaatttttaactatgtggtcaca240
cttactacaataatgaccattttacgacacacaaactacgacggttaaaaaaaaaacatc300
gttgaaagatacacggtaacagtgtagtaattttaaagaatgtatacaatgatggttatt360
gaataaccatttttaatagcgagtttaaggagtacaacgacaggttgacaaagaccgtcg420
tagttgtattggtagtacaaagatagttttttccttttgactgtctttgaataagcttac480
ttattattcccaaaatcactaataattaagtagcttcctctccgttgcgctcgcgtaacc540
ttcttctgttcattgttgtccaacttctggattccctcgactgtttgaattcaatgactc600
cctcaaaaccacccgcactctctgactcaagctttgactaggccctttgtctcttgaatc660
tctgcgaagtaacgccaaagcagttcatctgtgaagtctccgtcaaaggtgtctatgtag720
gtaagaacccctgtccgtcaagtttttgttttccttcgatgtggttgaatctagtttgtt780
cttagtcgaaagggtgtctatttgttgttttctggaaccgagtgaagagatcgagttaag840
cttaatgggaatgtgagatatcttcgtttcgttaaggtagcacctggtttgtagcaagtc900
cacttcggtgaatctaattaacaaaaatatataccccaagcaacgataaaaaacat956
<210>7
<211>105
<212>dna
<213>大豆
<400>7
gtcccttcggggacatccgataaaattggaacgatacagagaagattagcatggcccctg60
cgcaaggatgacacgcacaaatcgagaaatggtccaaattttttt105
<210>8
<211>982
<212>dna
<213>大豆
<400>8
ttataaatgagataaaatttatagatcaattaatgttttttttattgtgtaccaattttt60
ttattgatcgaaaaggtaaaatttaggagagttaaatactcttttcatttttattaatta120
gttgtcgtttatggtattttagtagggtgactgctttgataaataaataacaaaggaaaa180
catatttatcctaaattagatgcggatacacattggagattggctaggaagaagtcagta240
gttacaaagttaaaatgatctttaattaatttttatgcaacaagagacaagacctgagca300
ttgttgaagaaacattttagactcataaatggacatacaaagacaaggcataaaactcct360
ctgtttaggtggctggggctatgaccaggataaatggagttggtggtgaaggatgcttga420
ggaaggtggagttgttccagatttagttttcgacttagatgatgcatggaactggctagt480
gacgtggatggtggtaggttactttcaggtcatgattttttgtttctaaatgatactcac540
actcccttccagttttttttttttaaactcagctcccttgcttcctccaccggttatcat600
aatactgaaccaaatcaaacattacagtcaaggtactatgaatatgaaacctgaaatcct660
atgaatgtcataaatttattttaaataataaatttatttagaataatatttttttgggta720
agagttataaaataaaatacaaaaaaaaaacctaatatcaatttttcactgactccgttt780
atattgagacttgagaaagatggttcccgtttgctcccggtggaggctccgaggctgtgt840
atatactcgacattactttagcttgttttgttgtttctttccctttcccacaagactcag900
gtctcgttcgcaaacgagtcccacaccgtctaaacttaccacaatattagcgtttataat960
tagatgcactgcatcacttatt982
<210>9
<211>105
<212>dna
<213>大豆
<400>9
gtcccttcggggacatccaataaaattggaacgatacatagaagattagcatggcccctg60
tgcaaggatgacatgcacaaatcaagaaatggtccaaattttttt105
<210>10
<211>949
<212>dna
<213>大豆
<400>10
caattggacccctcctacctacaaatgctttaaatgcaggcttaagtgaggtcattttgt60
atatacaacatcctgaacaaccaattttagagataacaaatacacaatcaactagataat120
gaaaaaacttatcaagcaagattgcaaaaataaccataatatatgagtacttacccaaca180
ctaccacaataatggatatacgtggtgatagtgtagtaattttaacgaatgtatatgatg240
acgattattgaatatccattttcaatagtgagtttaacctttgaacaaggagtacaacga300
caggttgacaaaaaccgtcataattgtattgctagtacaaagacgatttttatataagac360
cgtctttgaataatcttacttattattcccaaaatcactaattagcttcctctcaatcgc420
gtgtaaccttcttctttacttacgttcatctttgtccaagttgtggattccctcgaaggt480
ttgaattcaatgactccctcaaacctcgcttagttttctatttctgttcgactttctttg540
accctcctagaatttctgcttaaaagtgtgtttgcctttgtgtttacagttgtgaccctc600
gaacccacttgcactctctcactcaagctttgactgggccctctctctcttgaagctttg660
cgaagcaatgctggagtagttcatctgtcaagtctccgtcaaaggtgtctgtgtaggtaa720
gaacccttatctttcaagtgttcgttttccttcgatgtgggtacaatggttgaatccagt780
ttgttctcagtctaaagggtgcttatttgttcttttctacaaccgagtgaagagatcgag840
ttaagcttaatggaaatgtgaggtatcttcctttcgttagggcaacacctagtttgtagc900
aagtccacatcggtgaatataattaaccccaagcaacgataaaaaacat949
<210>11
<211>89
<212>dna
<213>大豆
<400>11
gtcccttcggggacatccgaggaaattgaaagcatggcccttgcgcaaggatgacacgct60
tttatcaagaaatggtccaatttattttt89
<210>12
<211>956
<212>dna
<213>大豆
<400>12
aatacaaagaaacaaaacctaaggaatcctaaaggaggaatttgagagaggttaagtcct60
gtgtcttgagagaaaaataatataatatggaattatctcaattctaagaatacggtcttt120
atctttaattactagtagtatataggatggaattttctatatgtaaaactaaatttggat180
tcatttgacaataatattatgcaggcacggaattcttcaatttttaactatgtggtcaca240
cttactacaataatgaccattttacgacacacaaactacgacggttaaaaaaaaaacatc300
gttgaaagatacacggtaacagtgtagtaattttaaagaatgtatacaatgatggttatt360
gaataaccatttttaatagcgagtttaaggagtacaacgacaggttgacaaagaccgtcg420
tagttgtattggtagtacaaagatagttttttccttttgactgtctttgaataagcttac480
ttattattcccaaaatcactaataattaagtagcttcctctccgttgcgctcgcgtaacc540
ttcttctgttcattgttgtccaacttctggattccctcgactgtttgaattcaatgactc600
cctcaaaaccacccgcactctctgactcaagctttgactaggccctttgtctcttgaatc660
tctgcgaagtaacgccaaagcagttcatctgtgaagtctccgtcaaaggtgtctatgtag720
gtaagaacccctgtccgtcaagtttttgttttccttcgatgtggttgaatctagtttgtt780
cttagtcgaaagggtgtctatttgttgttttctggaaccgagtgaagagatcgagttaag840
cttaatgggaatgtgagatatcttcgtttcgttaaggtagcacctggtttgtagcaagtc900
cacttcggtgaatctaattaacaaaaatatataccccaagcaacgataaaaaacat956
<210>13
<211>107
<212>dna
<213>大豆
<400>13
gtcccttcggggacatctgataaaattggaacgatacagagaagattagcatggcccctg60
cgcaaggatgacacgcacaaatcgagaaatggtccaaattttttttt107
<210>14
<211>950
<212>dna
<213>大豆
<400>14
aaaaggagatgaaatatttttttttaaatctttcgttctaattatatttttatccacatg60
attcaaacttataatcttatttaagatgactaataatatttaaatcaacgatttattgta120
aaaaaaaatatatttcttatttgagaaagagagaaattatacaaagataaatccataaaa180
taaataataaaaaattaatttagttaaagaaatataattaattaatattctaatttatac240
aatatacaatataaatatttgtattctttgatgctgcgaatgaattcggctttgacgcag300
tttgtaacaaagataatatgtatttatacacattatagtattttttaaaataaaattagt360
actaataacattataaaatattattcataaattataaaaaaaattgaattaatattctca420
tatatttttactaaaatgtactagattggggttaaaaaattagcaaatgaaaaaatataa480
taccttaagagaattgtaagtgtgcttttatatatttaaaattaatatattttgaaatgt540
taaaatataaaagaaaattcaatgtaaattaaaaataaataaatgtttaataaagataaa600
ttttaaaacataaaagaaaatgtctaacaagaggattaagatcctgtgctcttaaatttt660
taggtgttgaaatcttagccatacaaaatatattttattaaaaccaagcatgaaaaaagt720
cactaaagagctatataactcatgcagctagaaatgaagtgaagggaatccagtttgttc780
tcagtcgaaagagtgtctatctttgttcttttctgcaaccgagttaagcaaaatgggaat840
gcgaggtatcttcctttcgttaggggagcaccagatgcatagttagtcccacattgatga900
atataacaagagcttcacagaatatatagcccaggccacagtaaaagctt950
<210>15
<211>105
<212>dna
<213>大豆
<400>15
gtcccttggggacatccaataaaattagaaagatatagagaagattagtatggcccatgt60
gcaaggatgacacgcacaaatccagaagatttagaatttgttttt105
<210>16
<211>956
<212>dna
<213>大豆
<400>16
ttattctttcttgtaactaatgatttaatttataactataaccagaacatcaacataaaa60
ctataatatgccataattaagtagttgaactttaggttaattgtaccaataataataata120
ataataatcaataaataaataagaaactgtttagaacacttgacctaaaattttcggtcc180
agtttccaccagtgttaatttcttgagttatacatgtaccgaactcgataagcagatgta240
gtgtggaccacttacttacatgcattatgcatatgatctagttgcaggaatatagaatat300
atgtcactcaaaagtttaacatgcagggatatatacgtacctccactcatttacgttaga360
ctacaaccacttttgctaaccacctgaacaaacatctgaaccaaataataataataatat420
gtttgtcacattgactaatctctcttacgtacgccaattttcgtgtatttccacagcttt480
atccaaaaaggtggccgacattacgaatgaatgctcctcaaaccccaccccctccctttt540
aatgcttttttttcttcttattagggtaaaaacatatacttcatttagctataggaatta600
ggactctcgtatatgtatcaaattaaaaaggaataaataaataaataaataaacaacgaa660
caaaaattagtggctcaaactaaaatattatctacacgtgctgcggggatttccaatcga720
ataatatacggaaatttcattcaatttagggttgttaggtgctaccttgccgttgctacg780
ttagtgaataaaatagctagttgtccggggcaaaatcttacaaaatcttgtgcaattatc840
acttatatatggttccattgacccaagaaaagacaatgttatcaataaagatggatatac900
ttaataagatccaatccatatttcgcaatattataagtttaaatcttgttttgatg956
<210>17
<211>107
<212>dna
<213>大豆
<400>17
gtcccttcggggacatccgataaaattggaacgatacagagaagattagcatggcccctg60
cgcaaggatgacacgcacaaatcgagaaatggtccaaatcttttttt107
<210>18
<211>981
<212>dna
<213>大豆
<400>18
aaaaaagaccttatatgctaataaaaatatcaaatatgttaataaaaaaagtctacacat60
ttaaagtaaaaaaaagaaagaaaagtttaaaaaaattgttttaaacatcacttattgttg120
gaccgacttttcactcaagaaagcagttgaatcactattgagaggctggtttcatttttg180
tttggaaatttgatgtactaagctattctctattctcatgtaagcctaacaaacatatag240
gttaacatttgtctttattttatttccttcaatattctgatttttcagaaacaaaaggcc300
atgtttgattagcagtttgttagctcattccctatattgcactctactttattctttctt360
ttatattttggtcatttttgtttccatcctttaaacctaaaatacttatgccacatacat420
cagtttttacttactgagtactgagtagtatgcagaattgcagatatgacgttgcactgg480
gagctatacttgacacgtgatagtaaaagagtgattgagatttgtttttggttgaacgtt540
tgacatgtgtgatttagtataaagtgaagtaatggtcaaaagaaaaagtgtaaaacgaag600
tacctagtaataagtaatattgaacaaaataaatggtaaagtgtcagatatataaaatag660
gctttaataaaaggaagaaaaaaaacaaacaaaaaataggttgcaatggggcagagcaga720
gtcatcatgaagctagaaaggctaccgatagataaactatagttaattaaatacattaaa780
aaatacttggatctttctcttaccctgtttatattgagacctgaaacttgagagagatac840
actaatcttgccttgttgtttcattccctaacttacaggactcagcgcatgtcatgtggt900
ctcgttccccatttaagtcccacaccgtctaaacttattaaattattaatgtttataact960
agatgcacaacaacaaagctt981
<210>19
<211>88
<212>dna
<213>大豆
<400>19
gtccctttggggacatccgaggaaattggaagcatggcccctgcgcaaggatgacacgct60
tttatcaagcaatggtccaatttttttt88
<210>20
<211>956
<212>dna
<213>大豆
<400>20
ttcagacaactagagatctggttatcttccttaaaccactttaatattcattttctatct60
tcttatctttgattgtttatatctctataattgagtaaagcaagttattaatcataagta120
tcactactacaataatggttatttacgacacactaactacgacgattaagaagaaaatgt180
cgttgaaggatacacggtggcagtgtagtaattttaatgaatatatatacaatgacggtt240
attgaataaccatcttcaatagtgagtttaaggagtacaacgataggttgacaaagacta300
tcgtagttgtattggtagtacaaagatggttttttccttttgactgtctttgaataagct360
tacttattattcctggaatcactactaattaagtagcttcctctcagtcgcgcgcgcgta420
acctttttctctgcttcttccattcatcgttgtccaagttgtcgattccttcaaaggttt480
gaattcaatgactccctcaaaacccccttatgattttgtttctcttcaactttctctgac540
cctcctagaatttctacttaaaagtgtgtttgcctttgtgtttatagtgaccctcgaacc600
catctgcactctctcactaaagctttgactgggccctctgtctcttgaatctctgcgaag660
caacgctggagcagttcatctgtgaagtctccgtcaaaggtgtctgtgtaggtaaaaacc720
cctgtctgtcaagtgtttgttttcctttgatgtgggtactatggttgaatctagtttgtt780
ctcaatcgaaagggtgtctgttttttgttttctgcaatcgagtgaagagaccgagttaag840
cttaatgggaatgtgaggtatcttcatttcattagggcagcacctggtttgtagcaagtc900
cacttcagtgaatataattaacaaaaatatatttcccaagcaacgataaaaaactt956
<210>21
<211>107
<212>dna
<213>大豆
<400>21
gtcccttcggggacatccgataaaattggaacgatacagagaagattagcatggcccctg60
cgcaaggatgacacgcacaaatcgagaaatggtccaaattttttttt107
<210>22
<211>953
<212>dna
<213>大豆
<400>22
accagatagcatcacacactaagtttgttagaaaaaacttaagtttaattgtcacataat60
tcatcattcggtagagatgatgaactttaaatgtaaataagattttaatcgaagattgat120
cccataagatcataattgactcaaaggatcaaatcttgtagaaatatcttggaatttcat180
tgaaaagtcaaagattttaattaggatgattaaaaaaattatttaatcctaaaaatcaat240
cttacaaggaaccactataaaactagttgaacccatcgaaaactaactacagttgacaaa300
atctatttggttggttgattttttttaattaaaaacatccaatcttaatgatataattat360
agcttaatattataagatttttataaaaattatatttattttgtttctaattatgctaag420
agatattattatttgttatttaacttaaatattatcacaaacttgattgaaacttatgtt480
taatttaaaatattatatgtcatgagttatgactccaataatcacaattataaagtgaag540
tttaatttttagtattacaaatatttttttgttgtttaattttaattacttaatgtatta600
tgttaataattaaaaatacaaattatttattattaatgcaatcacagtttgtggatttga660
caaaagaaataggggatctaaaattgtagataagccaaagttaaaacttgaattgactat720
tttttgctctttactctgcaccaactttactattccttcttttagtgtgagcttcatgca780
tcttgttcaccgcaattccgctcggtgaaagttgcacaattcactcacaatctgtttctg840
gtctgttaggtttgttacttggagtgacacgatgacgcaacagtacaagtcccacatcgt900
ttgagtatacagttttcaagcagtttatattcccatagccttagcaagagctt953
<210>23
<211>108
<212>dna
<213>大豆
<400>23
gtcccttcggggacatctgataaaattggaacgatacagagaagattagcatggcccctg60
cgcaaggatgacacgcacaaatcgagaaatggtccaattttttttttt108
<210>24
<211>948
<212>dna
<213>大豆
<400>24
aatgcccgaagaatttgattgatcataatgatgtttttcctcttaattacatttttctca60
ttaaaaaacttaaattcaaaatcttgtataagagtataatattatttttagattaacgat120
ttattggtagatagaatataagaacttgtgttggaaagagagaaattataaataaataag180
agaaaaataattttcctaaaaaatataattaactagtatttgatttatacattatacaaa240
ataattatttgtgttctttgatactgcgaatgaattgggcttaaaccgagtttgtaaaaa300
agatactatgtatttatacaaattatagtatttttaaataaaattagtacaaataacatt360
aaaaaatattattaataaagtataaaatattatttttgactaaaatgtaggagattaggg420
ttaaaaaatagcaaataaaaaaattataataccttaagagaattgtaagtgtgtttttat480
atatttaaaattaatatattttgaaatcttaaaaatgtaaaaaaaatcaatggaaaataa540
aaagaaataaataaatattcagtagataaattttaaaatataaaagaaaatgtccaacaa600
gaggattaagatcctgtgcccttacattttaggtgttgaaatcttagccatacaaaatat660
attttctttaaaacgaagcatgaaaaaagtctctataggccagcacctttgaatctttag720
ctataactcatgcagctagaaatgaggagaagggaatccagtttgttctcagtcgaaagg780
gtgtctctttgttctattctgcaaccgagcgaagagacccagttaagctaaatgggaatg840
tgaggtatcttcctttcgttagggcagcacctggtttgttgcaagtccacatcggtgaat900
ataacaaaaacttaatagaatatatagcccaagcaacgataaaagcat948
<210>25
<211>106
<212>dna
<213>大豆
<400>25
gtcccttcggggacatccgataaaattggaacgatacagagaagattagcatggcccctg60
cgcaaggatgacacgcacaaatcgagaaatggtccaaatttttttt106
<210>26
<211>982
<212>dna
<213>大豆
<400>26
tggagaattgggatcaaggattgaggagataatatccgtcctcgtccagcctcattgcca60
tatctacactgcctaaaaataaaatttttactattctttttaaccaatgatctatcataa120
ttaaaggatgtttcaccattaagtttaaatttgaattagtttttggaaagggattgtgta180
gaatactgaatacgtagattacagatttttctaaattgatcatggtaaaaccattttgtg240
aggattaagtttgttttagaaacaactgttgaacatttttaatttctcttgtagatggtt300
gtatcttaatcaaaatgctaaatgtatgcaagtatcaaccatggctatgaaaatagactt360
gaaacgatgtgggagaagaatttgaagattatgaagaaatgtagatatcattataaaacc420
tattttattatacaaatacacttgttttatcttttcttttctacaaaacattataaaaag480
taaaatataactactttgttttttaataaaaaaaattcaatgggagatactatggattca540
attaccttactgattttatttcatatgtgccagaagtatttcagtttattttgaaaaatc600
agaaaaaaaatgtctggaataaaatataataagcgatactaataaataattgaacaagat660
aaatggtaaaatgtcaaatcaaaactaggctacagagtgcagagcagagtcatgatgaat720
gacagctagttctacttactacaccgattcttgtgtacataaaaatattttaaaataatt780
gaatctttctttagccagctttgacaacaatgtacaccgttcgtacttcttactggtagg840
caatgcttcttgtttgctttcggtggaaggtgtatatactcaacattacttctttttcag900
cgtgttttcttacgggagtcccacaccgcccaaaactaatacagtattcttgtttataaa960
gaagtgcaccacttcaattgtt982
<210>27
<211>105
<212>dna
<213>大豆
<400>27
gtcccttcggggacatctgataaaattggaacgatacagagaagattagcatggcccctg60
cgcaaggatgacacgcacaaatcgagaaatggtccaaattttttt105
<210>28
<211>999
<212>dna
<213>大豆
<400>28
ttaaaaaaattatctttatcaaaataattaaatataagagaaaaagtgcactaacaatat60
aaaagtattttatgttgtcttttacttataaattaccatttatgataaattagttgattt120
ttacaaaattgtcctaaaagtcatacaaacgataatttgtgattgaatgacaattttaca180
ttgactggacatcactattaaactctaaatttaaaatgtttttaaactcaaagatatata240
taagtatacactacataataatggaggtgtaagattaacttgtgatataaagaaaaagaa300
agaagaaaaaaatcataaatttaattctttctgccaataaaaaaattaataattaatgtt360
tgcccataaaagaaactatacagtactattagagtgtttgtaaaataacatgaaatagaa420
gtaaataaaaataaaaattatgaattaaaataagatgtaaaaatatgaattttatctcat480
tttattgtttatttcatttctttttaactcacctttggtgcaaatacaacagtttgaaaa540
taaactgctaagttgtgaatctcaccactaaaatctattaaaatataattttaaaagata600
aatttgttattcaataataggcttttaaatataggaaaatgcataatttaactaattaaa660
gaaaataaaaatgcaagtgcggtgacaagacaagctagaataaagttgcaaagaaatgac720
agggctacaaaaggctcacctacttctggatttaccaaacttctgtttgtccccatactc780
caaaaacaaaaccatttttttttatcttcgtttttgtttgctttgactgtgagttgaggc840
ccaactttctgcttctgtccgactctatttgatgaattttgtttgcctcctgtgatgtga900
aggatgtatcattgaaagggaacgtgtctcaatgatcccacatcggccaaatatgctcat960
tacattgcgtttatatagtcccaggaaaacatatggatt999
<210>29
<211>107
<212>dna
<213>大豆
<400>29
gtccctttgggggcatcctatacaaatggaatgatacagagaagattagcatggcccccg60
tgcaaggatgacacgcacaaattgagaaatggtccaaattttgtttt107
<210>30
<211>995
<212>dna
<213>大豆
<400>30
gtaatttttccacagcagacagaagtagtaataaaaggcaactataggaataataggatt60
attttctctgccctgatacgtagtttcaagtttcgagctttcctttgtttgatggtgagt120
acttgtactgaacttataattgattagcctttgcatggtaagcagaattattcttgctaa180
cagaactctgatgcttggatttgatgttgttagtcattatacatgatgagcttatcgaga240
aatcgttctcaaatctatagtcacaaatctacactacttttttcttcttttttatatcaa300
ttgacctcataaattataacagttaacttgtttgtggttgatttggttgctttttttatt360
cttcctctaaccgcatccaattacaccactaattatactttacagtgtttgaaagaaatt420
tgaatgtaactaacaatattgaaatttaaattagggaagaagtattcttacgttgcaaac480
gagtatgacaatggcaaagtatccttaatttacactgcaaggataacaactttacataac540
tcggaatttcatatctttgaaatgaggaagcaaggttttgtattttacaaaagtttatgc600
aaatataatgaggtaattgaactagttagttgtgttatgtatcatacactattacatggc660
actgtttgtccgctcattacatgattcagcttatagatgaaagagtgaaggaacttgctc720
ttaaattataggacaagttgaactaatttgttaggtgattggttggctgcatataaatga780
taataatagaatacattgaaatatctgtatctttctgtgtatcattccatctttcatctt840
gcatgttgtttgccaccaatgaagggctgtgcaggactcaagcatttggctccagctagt900
gattgcaatgtgactgtgtctgtctccacagtcccatatcaaccaaatatgctccatgca960
ctgtgtttatataacaccaggggaatacattaatt995
<210>31
<211>106
<212>dna
<213>大豆
<400>31
gtcccttcggggacatccgataaaattggaacgatacagagaagattagcatggcccctg60
cgcaaggatgacacgcacaaatcgagaaatggtccaaatttttttt106
<210>32
<211>1059
<212>dna
<213>大豆
<400>32
atattaaagagtatgttgctaaacacttttttatttttaataaaccatgatagttatctc60
agcagaaaccatgtatccaaagcaaaaatttcaacaagttcaatgctgattcagaaacat120
cttatttttcacaatttttaacaaataatgaaatggcatgtttacaatattaatagtaat180
agtattaaattaaataataaattatatcaaaagactaattttttaatacatatattactt240
ataaaatattattaataatgttgataattattattgttgaaaatattttttttaagtatg300
attacttaaatcttattaaaaaaatgtcacaatgacacatgtccaacttctgaacaatta360
tatatatatatatatatatatatatatatatatatattaaaatatagttattatttaagt420
tctgttaatgattattaatctttatcaaagaaggatgattattgataatggaatttaatt480
tgtttccaattatatttttttatttgtaagacttaaatatgatacttaagaaagttgagt540
tctatttcatttgaccaatcacatattgataaagttttagtgtttgaaaaaaaaagtata600
gataaatataaaatgttttataaaatataaaacgataaaaatgttttaaacgatatatat660
tataaaaaaaaacgtttcaaaaataaatacaaaaatgtttttaaatatatataatttaac720
tcattaaagaaaataaaaatgcaagtgcggtgacaagacaagctaaaagttgcaaaagaa780
atggcagggctataaggctcacctactcctggatttaccaaattttggttcgtccctata840
ctcgaaaaataaaacaaaataaatttcagtatcttcgtttttgtatgctttgactgtgag900
gcgaggccaactttcttcttctgtctgagatgaattttgtttgcctcctgtgaaggatgt960
atcattcaaagtgaatgttttgcaactgccagtagtcccacatcgaccaaatattcttat1020
tacagtgtgtttatatagcacctggagaaggaatgggtt1059
<210>33
<211>117
<212>dna
<213>大豆
<400>33
gtcccttcgaagaatggggacatatgataaaattggaaccatacagagaagattagcgtt60
gccctgtgcaaggatgacacgcacaaatcaagaaatgctccaattttttgttttttt117
<210>34
<211>936
<212>dna
<213>大豆
<400>34
ccgatggaaatatttagattttctacagaaattaatctatggataaaattccatccgtaa60
ttttaactttctgatggaaaaattgtccatcggtaaattccatcaataattcaaaaattt120
ctatacaaaattttcaaaatttttgacgaaatttattcttttgataaattctatcaacaa180
tttaaatgtttctgatgaaatatttttcgttggaaataataatttttcttgtagtgtttg240
agttgttcttaggctacaacaatatgtgacgatgatgtgagacatatttaggttgcgaca300
atgatgtgctcaagctgtgatgaggtgcgacaatgaaagattattattgtgattttgaaa360
tcatgcagtgtgggatgaggagcggatggttatgccttttccctctttattttagttttt420
gataaaaagaatttgttttcaattattaaaaaaaatattactcattaaaatatatcacag480
gcggatgtagtatggagggatgttaatacagttcttattctcaactgtgaaaggggttgc540
tgcagccaatcgaaccacttcattccatgctggggaggttgcaatgaaaagagatttagg600
ccccgccaaaccctcataacattcgtgcatctaaagaatatattgcaccttggccatgta660
tcatcatcagaacaccttggccgtataaaattttgaatttaactcaaaagttacgcaaaa720
ttaattctaacacaaaaataattctgaatttttctctcatgtgaaattaaacatctaaaa780
atatattcaaaatcaatttcagacttagaatcaaatttttaacaccaaatcaaaccgaaa840
cccaagatactttctcaaattgttaataaactgaatttgaaaaaatatagacaatttacg900
tgtttatataacaaaagacaaacatagggatcgtct936
<210>35
<211>106
<212>dna
<213>大豆
<400>35
gtcccttcggggacatccgataaaattggaacgatacagagaagattagcatggcccctg60
cgcaaggatgacacgcacaaatcgagaaatggtccaaatttttttt106
<210>36
<211>982
<212>dna
<213>大豆
<400>36
ctagtggagcctgcgattcaactgctgctcctgctgccaccttgctgcaaccctgaaaaa60
gctgagctgcaaccctgaaatagctgaggagccctgctacctgcaaatcgccagtttgac120
tggctagtcccatgccacgtgttgacgccagcccaaacccgccaatagggctggcgattt180
tcttcttccatgcatgcactctcgtacaaaacagcccctcattaaaaaatctttacaaac240
aacctcattttagaaaatagtttgtaaaaagatctccaatgggaaaatttgccttagttt300
ataattaaaacttatttatactcaaattttagtagcttctgacttatagttgagtgttct360
ttgaaatttcaacgtggctaaatggcatacgtaatcatgtcaagcataaaatttaagaat420
tattttatttatataatatttttatatcataatctattttattgtacacttttttttttg480
cttttgttttctacaaaacattataaaaagtaagatataacaactttttttttaaaaaaa540
tcaataggaaatattatcggttcagttaatttacagagagatatttatttcatatgtgcc600
agaagtatttcagttccttatgaaaaatcagaaaaatgtatggaataaaatataataatc660
gatactaataatagaacaaaataaatggtaaaatgtcaaatcaaaactaggctgcagtat720
gcagagcagagtcatgatgatactacttactacaccgattcttgtgtgcagaaaaatatg780
ttaaaataattgaatctttctctagccaaatttgacaacaatgtacaccgttcatattga840
gagacgatgcttcttgtttgctttcggtggaagctgcatatactcaacattactccttca900
gcgagttttccaactgagtcccacattgcccagacctaacacggtattcttgtttataat960
gaaatgtgccaccacatggatt982
<210>37
<211>106
<212>dna
<213>大豆
<400>37
gtcccttcggggacatccgataaaattggaacgatacagagaagattagcatggcccctg60
cgcaaggatgacacgcacaaatcgagaaatggtccaaatttttttt106
<210>38
<211>1021
<212>dna
<213>大豆
<400>38
tgaggaataaataaagcacatatttattttttattttatttaactaaatgacaaatttat60
ttaattattttttcaattaccaaaatgagaaaaattaatgtaaaaatttaatatttttta120
attcacaaaaattccatgtcaacataacatatgtcaagtgagcataaagattagtgtgga180
acatagataggtgataattgtgtattcatatcaacatataacatagagatagacagaaag240
aacgaaaatgtgtaacggaataaaagattaatgatgttgatcaattaaaaatttgtttga300
ggattaaaactcaaaatttcaaaaaatttaaatactaaaaacttagttaacctaaaaatt360
aaaggaaagagaaaaaaaaaatctaacagagagggtgaatcatataaacaatgatatata420
aaagtggtttattatagaatcacgtcatatgtttttcctcggtaaccttgtttcttttat480
tctaagttcttgttgccatgtttttgcgcttctctatccaagtatccatttatattcata540
attttttttttttgtttttttatacaaggacggctgattcaatcatcacaccacacgtca600
tattaaaaaaatatagtagatttattttaaaatagagagaatcgttaagaaaaaaataaa660
tagtaaagtaaatgaaaacccaaataatatcattattatgtcaataagtcggagaggata720
gtaatcaaatggtctatgaggtggtggttcattcaacatatagcacctattcattgttcc780
taaaacataatttaagaacaaaaacttaaacttaaataataataataaaagagtacatcg840
aagtatctgtgttctctatccttctgactaacattcatgttgtttgtattcagcaaaggg900
ccgtgcaggatttgtgcgtcgcgctccggttagttattgcagtgaccgtctctttagtcc960
cacatcgagtaattatgcttcatacagtctgtttatataacagagatggaacaaactggt1020
t1021
<210>39
<211>110
<212>dna
<213>大豆
<400>39
gtccctgtgggggtatctgacaaaattggaactttcatagaatgattaacagcatgactc60
ctctgcaaggatgatacgcacacaaatcgaaaaatgatttaatttttttt110
<210>40
<211>947
<212>dna
<213>大豆
<400>40
tattttttaacaaatattagttatcgataaaataaatattatggactaaaaatataatta60
aaaataataataatgaagaagtagaaataattaattaaaattaaactttcaaaaatttaa120
gttaaactatttaactaaaatacattgatctaaataaacaatatcaaaataaatatgcaa180
aactttcaaaattctttattgtttctttgttaagtaatggagaaattttatcattttcaa240
aatttttattattttcaaaatttttattattttttattattttcaacaatattatttaaa300
aactatccttatgaaaatatgtttcttttaaacaaaaggtagtaatttttttaatagaaa360
aatatagtaaatgtttaaaaactattctcattaaatttagctactgattgttttaaaaaa420
ttattttatttcctccacatacatattgtttctcttcttctttatttattatatttttct480
actataaaactcacagcaccaataaagcccaagacttagaattgggctataagtccaaac540
cataactgcacagattgattttggtggtaatatcaacctatttatgaaaaagagtaaaag600
attaaacattcacaatctttcaacctacaagttcatagtctatctataacatcagccaat660
aatagaaaaagagtaactgtatctaatattctaatttcattgatgtttcatatgaatttc720
ctaccgtcacccatcatgtatttaaagccactatctcgtcattttcagggaaagacaact780
aaatgtgtaaaggcacgtggaagaggtaggttcattttcaaggcagccaacttaacatta840
acaactaaatgtgtatctaataagacttgaccctttcagctagtaattgcattactaaat900
ataactagttccctatctttatatagcaccaggcgtaacaatgaata947
<210>41
<211>20
<212>dna
<213>大豆
<400>41
gtcccttgtacttgtacgta20
<210>42
<211>28
<212>dna
<213>人工序列
<220>
<223>grna正向引物grna-f
<400>42
cccttgtacttgtacgtagttttagagc28
<210>43
<211>23
<212>dna
<213>人工序列
<220>
<223>am标记的grna探针grna-t
<400>43
caagttaaaataaggctagtccg23
<210>44
<211>21
<212>dna
<213>人工序列
<220>
<223>grna反向引物grna-r
<400>44
cggtgccactttttcaagttg21
<210>45
<211>21
<212>dna
<213>人工序列
<220>
<223>35s启动子正向引物35s-277f
<400>45
gacagtggtcccaaagatgga21
<210>46
<211>21
<212>dna
<213>人工序列
<220>
<223>fam标记的35s启动子探针35s-299t
<400>46
ccccacccacgaggagcatcg21
<210>47
<211>22
<212>dna
<213>人工序列
<220>
<223>35s启动子反向引物35s-345r
<400>47
cgtggttggaacgtcttctttt22
<210>48
<211>24
<212>dna
<213>人工序列
<220>
<223>hsp正向引物hsp-f1
<400>48
caaacttgacaaagccacaactct24
<210>49
<211>20
<212>dna
<213>人工序列
<220>
<223>vic标记的hsp探针hsp-t1
<400>49
ctctcatctcatataaatac20
<210>50
<211>21
<212>dna
<213>人工序列
<220>
<223>hsp反向引物hsp-r1
<400>50
ggagaaattggtgtcgtggaa21
<210>51
<211>25
<212>dna
<213>人工序列
<220>
<223>u6rna正向引物u6rna-f1
<400>51
tggaacgatacagagaagattagca25
<210>52
<211>16
<212>dna
<213>人工序列
<220>
<223>fam标记的u6rna探针u6rna-t1
<400>52
tcatccttgcgcaggg16
<210>53
<211>22
<212>dna
<213>人工序列
<220>
<223>u6rna反向引物u6rna-r1
<400>53
ttggaccatttctcgatttgtg22
<210>54
<211>24
<212>dna
<213>人工序列
<220>
<223>atps正向引物atps-87f
<400>54
catgattgggagaaaccttaagct24
<210>55
<211>20
<212>dna
<213>人工序列
<220>
<223>vic标记的atps探针atps-117t
<400>55
ttctcctgctaagacatact20
<210>56
<211>20
<212>dna
<213>人工序列
<220>
<223>atps反向引物atps-161r
<400>56
agattgggccagaggatcct20
<210>57
<211>3365
<212>dna
<213>人工序列
<220>
<223>质粒片段,qc795a
<400>57
cgcgccggtacccgggtgtgatttagtataaagtgaagtaatggtcaaaagaaaaagtgt60
aaaacgaagtacctagtaataagtaatattgaacaaaataaatggtaaagtgtcagatat120
ataaaataggctttaataaaaggaagaaaaaaaacaaacaaaaaataggttgcaatgggg180
cagagcagagtcatcatgaagctagaaaggctaccgatagataaactatagttaattaaa240
tacattaaaaaatacttggatctttctcttaccctgtttatattgagacctgaaacttga300
gagagatacactaatcttgccttgttgtttcattccctaacttacaggactcagcgcatg360
tcatgtggtctcgttccccatttaagtcccacaccgtctaaacttattaaattattaatg420
tttataactagatgcacaacaacaaagcttgtcccttgtacttgtacgtagttttagagc480
tagaaatagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagt540
cggtgcttttttttgcggccgcaattggatccaattccaatcccacaaaaatctgagctt600
aacagcacagttgctcctctcagagcagaatcgggtattcaacaccctcatatcaactac660
tacgttgtgtataacggtccacatgccggtatatacgatgactggggttgtacaaaggcg720
gcaacaaacggcgttcccggagttgcacacaagaaatttgccactattacagaggcaaga780
gcagcagctgacgcgtacacaacaagtcagcaaacagacaggttgaacttcatccccaaa840
ggagaagctcaactcaagcccaagagctttgctaaggccctaacaagcccaccaaagcaa900
aaagcccactggctcacgctaggaaccaaaaggcccagcagtgatccagccccaaaagag960
atctcctttgccccggagattacaatggacgatttcctctatctttacgatctaggaagg1020
aagttcgaaggtgaaggtgacgacactatgttcaccactgataatgagaaggttagcctc1080
ttcaatttcagaaagaatgctgacccacagatggttagagaggcctacgcagcaggtctc1140
atcaagacgatctacccgagtaacaatctccaggagatcaaataccttcccaagaaggtt1200
aaagatgcagtcaaaagattcaggactaattgcatcaagaacacagagaaagacatattt1260
ctcaagatcagaagtactattccagtatggacgattcaaggcttgcttcataaaccaagg1320
caagtaatagagattggagtctctaaaaaggtagttcctactgaatctaaggccatgcat1380
ggagtctaagattcaaatcgaggatctaacagaactcgccgtgaagactggcgaacagtt1440
catacagagtcttttacgactcaatgacaagaagaaaatcttcgtcaacatggtggagca1500
cgacactctggtctactccaaaaatgtcaaagatacagtctcagaagaccaaagggctat1560
tgagacttttcaacaaaggataatttcgggaaacctcctcggattccattgcccagctat1620
ctgtcacttcatcgaaaggacagtagaaaaggaaggtggctcctacaaatgccatcattg1680
cgataaaggaaaggctatcattcaagatgcctctgccgacagtggtcccaaagatggacc1740
cccacccacgaggagcatcgtggaaaaagaagacgttccaaccacgtcttcaaagcaagt1800
ggattgatgtgacatctccactgacgtaagggatgacgcacaatcccactatccttcgca1860
agacccttcctctatataaggaagttcatttcatttggagaggacacgctcgagctcatt1920
tctctattacttcagccataacaaaagaactcttttctcttcttattaaaccatgaaaaa1980
gcctgaactcaccgcgacgtctgtcgagaagtttctgatcgaaaagttcgacagcgtctc2040
cgacctgatgcagctctcggagggcgaagaatctcgtgctttcagcttcgatgtaggagg2100
gcgtggatatgtcctgcgggtaaatagctgcgccgatggtttctacaaagatcgttatgt2160
ttatcggcactttgcatcggccgcgctcccgattccggaagtgcttgacattggggaatt2220
cagcgagagcctgacctattgcatctcccgccgtgcacagggtgtcacgttgcaagacct2280
gcctgaaaccgaactgcccgctgttctgcagccggtcgcggaggccatggatgcgatcgc2340
tgcggccgatcttagccagacgagcgggttcggcccattcggaccgcaaggaatcggtca2400
atacactacatggcgtgatttcatatgcgcgattgctgatccccatgtgtatcactggca2460
aactgtgatggacgacaccgtcagtgcgtccgtcgcgcaggctctcgatgagctgatgct2520
ttgggccgaggactgccccgaagtccggcacctcgtgcacgcggatttcggctccaacaa2580
tgtcctgacggacaatggccgcataacagcggtcattgactggagcgaggcgatgttcgg2640
ggattcccaatacgaggtcgccaacatcttcttctggaggccgtggttggcttgtatgga2700
gcagcagacgcgctacttcgagcggaggcatccggagcttgcaggatcgccgcggctccg2760
ggcgtatatgctccgcattggtcttgaccaactctatcagagcttggttgacggcaattt2820
cgatgatgcagcttgggcgcagggtcgatgcgacgcaatcgtccgatccggagccgggac2880
tgtcgggcgtacacaaatcgcccgcagaagcgcggccgtctggaccgatggctgtgtaga2940
agtactcgccgatagtggaaaccgacgccccagcactcgtccgagggcaaaggaatagtg3000
agaacctagacttgtccatcttctggattggccaacttaattaatgtatgaaataaaagg3060
atgcacacatagtgacatgctaatcactataatgtgggcatcaaagttgtgtgttatgtg3120
taattactagttatctgaataaaagagaaagagatcatccatatttcttatcctaaatga3180
atgtcacgtgtctttataattctttgatgaaccagatgcatttcattaaccaaatccata3240
tacatataaatattaatcatatataattaatatcaattgggttagcaaaacaaatctagt3300
ctaggtgtgttttgcgaattcgatatcaagcttatcgataccgtcgagggggggcccggt3360
accgg3365
<210>58
<211>1131
<212>dna
<213>人工序列
<220>
<223>质粒片段,qc819a
<400>58
cgcgccggtacccgggttaagagaattgtaagtgtgcttttatatatttaaaattaatat60
attttgaaatgttaaaatataaaagaaaattcaatgtaaattaaaaataaataaatgttt120
aataaagataaattttaaaacataaaagaaaatgtctaacaagaggattaagatcctgtg180
ctcttaaatttttaggtgttgaaatcttagccatacaaaatatattttattaaaaccaag240
catgaaaaaagtcactaaagagctatataactcatgcagctagaaatgaagtgaagggaa300
tccagtttgttctcagtcgaaagagtgtctatctttgttcttttctgcaaccgagttaag360
caaaatgggaatgcgaggtatcttcctttcgttaggggagcaccagatgcatagttagtc420
ccacattgatgaatataacaagagcttcacagaatatatagcccaggccacagtaaaagc480
ttgtcccttgtacttgtacgtagttttagagctagaaatagcaagttaaaataaggctag540
tccgttatcaacttgaaaaagtggcaccgagtcggtgcttttttttgcggccgctcgagt600
gccagaagtatttcagtttattttgaaaaatcagaaaaaaaatgtctggaataaaatata660
ataagcgatactaataaataattgaacaagataaatggtaaaatgtcaaatcaaaactag720
gctacagagtgcagagcagagtcatgatgaatgacagctagttctacttactacaccgat780
tcttgtgtacataaaaatattttaaaataattgaatctttctttagccagctttgacaac840
aatgtacaccgttcgtacttcttactggtaggcaatgcttcttgtttgctttcggtggaa900
ggtgtatatactcaacattacttctttttcagcgtgttttcttacgggagtcccacaccg960
cccaaaactaatacagtattcttgtttataaagaagtgcaccacttcaattgttgtccct1020
tcggggacatccgataaaattggaacgatacagagaagattagcatggcccctgcgcaag1080
gatgacacgcacaaatcgagaaatggtccaaatttttgggcccggtaccgg1131
<210>59
<211>6611
<212>dna
<213>人工序列
<220>
<223>质粒片段,qc799a
<400>59
cgcgccggtacccgggttaagagaattgtaagtgtgcttttatatatttaaaattaatat60
attttgaaatgttaaaatataaaagaaaattcaatgtaaattaaaaataaataaatgttt120
aataaagataaattttaaaacataaaagaaaatgtctaacaagaggattaagatcctgtg180
ctcttaaatttttaggtgttgaaatcttagccatacaaaatatattttattaaaaccaag240
catgaaaaaagtcactaaagagctatataactcatgcagctagaaatgaagtgaagggaa300
tccagtttgttctcagtcgaaagagtgtctatctttgttcttttctgcaaccgagttaag360
caaaatgggaatgcgaggtatcttcctttcgttaggggagcaccagatgcatagttagtc420
ccacattgatgaatataacaagagcttcacagaatatatagcccaggccacagtaaaagc480
ttgtcccttgtacttgtacgtagttttagagctagaaatagcaagttaaaataaggctag540
tccgttatcaacttgaaaaagtggcaccgagtcggtgcttttttttgcggccgcaattgg600
atcgggtttacttattttgtgggtatctatacttttattagatttttaatcaggctcctg660
atttctttttatttcgattgaattcctgaacttgtattattcagtagatcgaataaatta720
taaaaagataaaatcataaaataatattttatcctatcaatcatattaaagcaatgaata780
tgtaaaattaatcttatctttattttaaaaaatcatataggtttagtatttttttaaaaa840
taaagataggattagttttactattcactgcttattacttttaaaaaaatcataaaggtt900
tagtatttttttaaaataaatataggaatagttttactattcactgctttaatagaaaaa960
tagtttaaaatttaagatagttttaatcccagcatttgccacgtttgaacgtgagccgaa1020
acgatgtcgttacattatcttaacctagctgaaacgatgtcgtcataatatcgccaaatg1080
ccaactggactacgtcgaacccacaaatcccacaaagcgcgtgaaatcaaatcgctcaaa1140
ccacaaaaaagaacaacgcgtttgttacacgctcaatcccacgcgagtagagcacagtaa1200
ccttcaaataagcgaatggggcataatcagaaatccgaaataaacctaggggcattatcg1260
gaaatgaaaagtagctcactcaatataaaaatctaggaaccctagttttcgttatcactc1320
tgtgctccctcgctctatttctcagtctctgtgtttgcggctgaggattccgaacgagtg1380
accttcttcgtttctcgcaaaggtaacagcctctgctcttgtctcttcgattcgatctat1440
gcctgtctcttatttacgatgatgtttcttcggttatgtttttttatttatgctttatgc1500
tgttgatgttcggttgtttgtttcgctttgtttttgtggttcagttttttaggattcttt1560
tggtttttgaatcgattaatcggaagagattttcgagttatttggtgtgttggaggtgaa1620
tcttttttttgaggtcatagatctgttgtatttgtgttataaacatgcgactttgtatga1680
ttttttacgaggttatgatgttctggttgttttattatgaatctgttgagacagaaccat1740
gatttttgttgatgttcgtttacactattaaaggtttgttttaacaggattaaaagtttt1800
ttaagcatgttgaaggagtcttgtagatatgtaaccgtcgatagtttttttgtgggtttg1860
ttcacatgttatcaagcttaatcttttactatgtatgcgaccatatctggatccagcaaa1920
ggcgattttttaattccttgtgaaacttttgtaatatgaagttgaaattttgttattggt1980
aaactataaatgtgtgaagttggagtatacctttaccttcttatttggctttgtgatagt2040
ttaatttatatgtattttgagttctgacttgtatttctttgaattgattctagtttaagt2100
aatccatggacaaaaagtactcaatagggctcgacatagggactaactccgttggatggg2160
ccgtcatcaccgacgagtacaaggtgccctccaagaagttcaaggtgttgggaaacaccg2220
acaggcacagcataaagaagaatttgatcggtgccctcctcttcgactccggagagaccg2280
ctgaggctaccaggctcaagaggaccgctagaaggcgctacaccagaaggaagaacagaa2340
tctgctacctgcaggagatcttctccaacgagatggccaaggtggacgactccttcttcc2400
accgccttgaggaatcattcctggtggaggaggataaaaagcacgagagacacccaatct2460
tcgggaacatcgtcgacgaggtggcctaccatgaaaagtaccctaccatctaccacctga2520
ggaagaagctggtcgactctaccgacaaggctgacttgcgcttgatttacctggctctcg2580
ctcacatgataaagttccgcggacacttcctcattgagggagacctgaacccagacaact2640
ccgacgtggacaagctcttcatccagctcgttcagacctacaaccagcttttcgaggaga2700
acccaatcaacgccagtggagttgacgccaaggctatcctctctgctcgtctgtcaaagt2760
ccaggaggcttgagaacttgattgcccagctgcctggcgaaaagaagaacggactgttcg2820
gaaacttgatcgctctctccctgggattgactcccaacttcaagtccaacttcgacctcg2880
ccgaggacgctaagttgcagttgtctaaagacacctacgacgatgacctcgacaacttgc2940
tggcccagataggcgaccaatacgccgatctcttcctcgccgctaagaacttgtccgacg3000
caatcctgctgtccgacatcctgagagtcaacactgagattaccaaagctcctctgtctg3060
cttccatgattaagcgctacgacgagcaccaccaagatctgaccctgctcaaggccctgg3120
tgagacagcagctgcccgagaagtacaaggagatctttttcgaccagtccaagaacggct3180
acgccggatacattgacggaggcgcctcccaggaagagttctacaagttcatcaagccca3240
tccttgagaagatggacggtaccgaggagctgttggtgaagttgaacagagaggacctgt3300
tgaggaagcagagaaccttcgacaacggaagcatccctcaccaaatccacctgggagagc3360
tccacgccatcttgaggaggcaggaggatttctatcccttcctgaaggacaaccgcgaga3420
agattgagaagatcttgaccttcagaattccttactacgtcgggccactcgccagaggaa3480
actctaggttcgcctggatgacccgcaaatctgaagagaccattactccctggaacttcg3540
aggaagtcgtggacaagggcgcttccgctcagtctttcatcgagaggatgaccaacttcg3600
ataaaaatctgcccaacgagaaggtgctgcccaagcactccctgttgtacgagtatttca3660
cagtgtacaacgagctcaccaaggtgaagtacgtcacagagggaatgaggaagcctgcct3720
tcttgtccggagagcagaagaaggccatcgtcgacctgctcttcaagaccaacaggaagg3780
tgactgtcaagcagctgaaggaggactacttcaagaagatcgagtgcttcgactccgtcg3840
agatctctggtgtcgaggacaggttcaacgcctcccttgggacttaccacgatctgctca3900
agattattaaagacaaggacttcctggacaacgaggagaacgaggacatccttgaggaca3960
tcgtgctcaccctgaccttgttcgaagacagggaaatgatcgaagagaggctcaagacct4020
acgcccacctcttcgacgacaaggtgatgaaacagctgaagagacgcagatataccggct4080
ggggaaggctctcccgcaaattgatcaacgggatcagggacaagcagtcagggaagacta4140
tactcgacttcctgaagtccgacggattcgccaacaggaacttcatgcagctcattcacg4200
acgactccttgaccttcaaggaggacatccagaaggctcaggtgtctggacagggtgact4260
ccttgcatgagcacattgctaacttggccggctctcccgctattaagaagggcattttgc4320
agaccgtgaaggtcgttgacgagctcgtgaaggtgatgggacgccacaagccagagaaca4380
tcgttattgagatggctcgcgagaaccaaactacccagaaagggcagaagaattcccgcg4440
agaggatgaagcgcattgaggagggcataaaagagcttggctctcagatcctcaaggagc4500
accccgtcgagaacactcagctgcagaacgagaagctgtacctgtactacctccaaaacg4560
gaagggacatgtacgtggaccaggagctggacatcaacaggttgtccgactacgacgtcg4620
accacatcgtgcctcagtccttcctgaaggatgactccatcgacaataaagtgctgacac4680
gctccgataaaaatagaggcaagtccgacaacgtcccctccgaggaggtcgtgaagaaga4740
tgaaaaactactggagacagctcttgaacgccaagctcatcacccagcgtaagttcgaca4800
acctgactaaggctgagagaggaggattgtccgagctcgataaggccggattcatcaaga4860
gacagctcgtcgaaacccgccaaattaccaagcacgtggcccaaattctggattcccgca4920
tgaacaccaagtacgatgaaaatgacaagctgatccgcgaggtcaaggtgatcaccttga4980
agtccaagctggtctccgacttccgcaaggacttccagttctacaaggtgagggagatca5040
acaactaccaccacgcacacgacgcctacctcaacgctgtcgttggaaccgccctcatca5100
aaaaatatcctaagctggagtctgagttcgtctacggcgactacaaggtgtacgacgtga5160
ggaagatgatcgctaagtctgagcaggagatcggcaaggccaccgccaagtacttcttct5220
actccaacatcatgaacttcttcaagaccgagatcactctcgccaacggtgagatcagga5280
agcgcccactgatcgagaccaacggtgagactggagagatcgtgtgggacaaagggaggg5340
atttcgctactgtgaggaaggtgctctccatgcctcaggtgaacatcgtcaagaagaccg5400
aagttcagaccggaggattctccaaggagtccatcctccccaagagaaactccgacaagc5460
tgatcgctagaaagaaagactgggaccctaagaagtacggaggcttcgattctcctaccg5520
tggcctactctgtgctggtcgtggccaaggtggagaagggcaagtccaagaagctgaaat5580
ccgtcaaggagctcctcgggattaccatcatggagaggagttccttcgagaagaacccta5640
tcgacttcctggaggccaagggatataaagaggtgaagaaggacctcatcatcaagctgc5700
ccaagtactccctcttcgagttggagaacggaaggaagaggatgctggcttctgccggag5760
agttgcagaagggaaatgagctcgcccttccctccaagtacgtgaacttcctgtacctcg5820
cctctcactatgaaaagttgaagggctctcctgaggacaacgagcagaagcagctcttcg5880
tggagcagcacaagcactacctggacgaaattatcgagcagatctctgagttctccaagc5940
gcgtgatattggccgacgccaacctcgacaaggtgctgtccgcctacaacaagcacaggg6000
ataagcccattcgcgagcaggctgaaaacattatccacctgtttaccctcacaaacttgg6060
gagcccctgctgccttcaagtacttcgacaccaccattgacaggaagagatacacctcca6120
ccaaggaggtgctcgacgcaacactcatccaccaatccatcaccggcctctatgaaacaa6180
ggattgacttgtcccagctgggaggcgactctagagccgatcccaagaagaagagaaagg6240
tgtaggttaacctagacttgtccatcttctggattggccaacttaattaatgtatgaaat6300
aaaaggatgcacacatagtgacatgctaatcactataatgtgggcatcaaagttgtgtgt6360
tatgtgtaattactagttatctgaataaaagagaaagagatcatccatatttcttatcct6420
aaatgaatgtcacgtgtctttataattctttgatgaaccagatgcatttcattaaccaaa6480
tccatatacatataaatattaatcatatataattaatatcaattgggttagcaaaacaaa6540
tctagtctaggtgtgttttgcgaattcgatatcaagcttatcgataccgtcgaggggggg6600
cccggtaccgg6611
<210>60
<211>5719
<212>dna
<213>人工序列
<220>
<223>质粒片段,rtw831a
<400>60
cgcgcctctagttgaagacacgttcatgtcttcatcgtaagaagacactcagtagtcttc60
ggccagaatggccatctggattcagcaggcctagaaggccatttaaatcctgaggatctg120
gtcttcctaaggacccgggatatcgctatcaactttgtatagaaaagttgggccgaattc180
gagctcggtacggccagaatccggtaagtgactagggtcacgtgaccctagtcacttaaa240
ttcggccagaatggccatctggattcagcaggcctagaaggcccggaccgattaaacttt300
aattcggtccgggttacctcgagatcttgttcccctccttggtttggcataaattgattt360
tcatggctcttctcggtcgaaactggagctaattcacccttagtctctcttaaaattctg420
gctgtaagaaacaccacagaacacataaattataaactaattataatttgaagagtaaaa480
tatgtttttactcttatgatttaattagtgtagttttaattttctcctttttttaaaaaa540
ttttggtattcataaatttcaattttttaaaaataattgttgttacccgttaatgataac600
gggatatgttatgttaccactaaatcggacaaaaaaaattcaaaacttttataaggatta660
aaattaacaaaaatattttaaaaaaatctaacctcaataaagttaaatttataagcacaa720
aataatacttttaagcctaatttggcaagacacaagcaagctcacctgtagcattaatag780
aaaggaagcaaagcaagagaaaagcaaccagaaggaagcgtttgcttggtgacacagcca840
tcttacttgaatttatggtattactgagaaaccttgatcttgcttcaaaatcttctagtt900
accctctttttataggcagaaagagaactagctagttgccaataggatatgaggacatgt960
ggtgcaatgcactcactcttcaaggacaagaaaaacaatggctacaattgtggttcaaat1020
caatgtctcctgctctgtcctgcctgaaaatgacacccttttgcttggaaaagaggatca1080
aagctaagaacaggagtggcttcattcccttcatgtaaccaaacactttcgcattctgtc1140
attcgtgaatcagcaaaatctgcaaccaaaaatatatggtgcctaaataaaagaaataaa1200
ataatttagagttgcggactaaaataataaacaaaagaaatatattataatctagaatta1260
atttaggactaaaagaagaggcagactccaattcctcttttctagaataccctccgtacg1320
tacactagtggtcacctaagtgactagggtcacgtgaccctagtcacttattcccaaaca1380
ctagtaacggccgccagtgtgctggaattcgcccttcccaagctttgctctagatcaaac1440
tcacatccaaacataacatggatatcttccttaccaatcatactaattattttgggttaa1500
atattaatcattatttttaagatattaattaagaaattaaaagattttttaaaaaaatgt1560
ataaaattatattattcatgatttttcatacatttgattttgataataaatatatttttt1620
ttaatttcttaaaaaatgttgcaagacacttattagacatagtcttgttctgtttacaaa1680
agcattcatcatttaatacattaaaaaatatttaatactaacagtagaatcttcttgtga1740
gtggtgtgggagtaggcaacctggcattgaaacgagagaaagagagtcagaaccagaaga1800
caaataaaaagtatgcaacaaacaaatcaaaatcaaagggcaaaggctggggttggctca1860
attggttgctacattcaattttcaactcagtcaacggttgagattcactctgacttcccc1920
aatctaagccgcggatgcaaacggttgaatctaacccacaatccaatctcgttacttagg1980
ggcttttccgtcattaactcacccctgccacccggtttccctataaattggaactcaatg2040
ctcccctctaaactcgtatcgcttcagagttgagaccaagacacactcgttcatatatct2100
ctctgctcttctcttctcttctacctctcaaggtacttttcttctccctctaccaaatcc2160
tagattccgtggttcaatttcggatcttgcacttctggtttgctttgccttgctttttcc2220
tcaactgggtccatctaggatccatgtgaaactctactctttctttaatatctgcggaat2280
acgcgtttgactttcagatctagtcgaaatcatttcataattgcctttctttcttttagc2340
ttatgagaaataaaatcactttttttttatttcaaaataaaccttgggccttgtgctgac2400
tgagatggggtttggtgattacagaattttagcgaattttgtaattgtacttgtttgtct2460
gtagttttgttttgttttcttgtttctcatacattccttaggcttcaattttattcgagt2520
ataggtcacaataggaattcaaactttgagcaggggaattaatcccttccttcaaatcca2580
gtttgtttgtatatatgtttaaaaaatgaaacttttgctttaaattctattataactttt2640
tttatggctgaaatttttgcatgtgtctttgctctctgttgtaaatttactgtttaggta2700
ctaactctaggcttgttgtgcagtttttgaagtataacaacagaagttcctattccgaag2760
ttcctattctctagaaagtataggaacttccactagtccatgaaaaagcctgaactcacc2820
gcgacgtctgtcgagaagtttctgatcgaaaagttcgacagcgtctccgacctgatgcag2880
ctctcggagggcgaagaatctcgtgctttcagcttcgatgtaggagggcgtggatatgtc2940
ctgcgggtaaatagctgcgccgatggtttctacaaagatcgttatgtttatcggcacttt3000
gcatcggccgcgctcccgattccggaagtgcttgacattggggaattcagcgagagcctg3060
acctattgcatctcccgccgtgcacagggtgtcacgttgcaagacctgcctgaaaccgaa3120
ctgcccgctgttctgcagccggtcgcggaggccatggatgcgatcgctgcggccgatctt3180
agccagacgagcgggttcggcccattcggaccgcaaggaatcggtcaatacactacatgg3240
cgtgatttcatatgcgcgattgctgatccccatgtgtatcactggcaaactgtgatggac3300
gacaccgtcagtgcgtccgtcgcgcaggctctcgatgagctgatgctttgggccgaggac3360
tgccccgaagtccggcacctcgtgcacgcggatttcggctccaacaatgtcctgacggac3420
aatggccgcataacagcggtcattgactggagcgaggcgatgttcggggattcccaatac3480
gaggtcgccaacatcttcttctggaggccgtggttggcttgtatggagcagcagacgcgc3540
tacttcgagcggaggcatccggagcttgcaggatcgccgcggctccgggcgtatatgctc3600
cgcattggtcttgaccaactctatcagagcttggttgacggcaatttcgatgatgcagct3660
tgggcgcagggtcgatgcgacgcaatcgtccgatccggagccgggactgtcgggcgtaca3720
caaatcgcccgcagaagcgcggccgtctggaccgatggctgtgtagaagtactcgccgat3780
agtggaaaccgacgccccagcactcgtccgagggcaaaggaatagtgaggtacctaaaga3840
aggagtgcgtcgaagcagatcgttcaaacatttggcaataaagtttcttaagattgaatc3900
ctgttgccggtcttgcgatgattatcatataatttctgttgaattacgttaagcatgtaa3960
taattaacatgtaatgcatgacgttatttatgagatgggtttttatgattagagtcccgc4020
aattatacatttaatacgcgatagaaaacaaaatatagcgcgcaaactaggataaattat4080
cgcgcgcggtgtcatctatgttactagatcgatgtcgacccgggccctaggaggccggcc4140
cagctgatgatcccggtgaagttcctattccgaagttcctattctccagaaagtatagga4200
acttcactagagcttgcggccgcgcatgctgacttaatcagctaacgccactcgacctgc4260
aggcatgcccgcggatatcgatgggccccggccgaagcttcaagtttgtacaaaaaagca4320
ggctggcgccggaaccaattcagtcgactggatccggtaccgaattcgcggccgcactcg4380
agatatctagacccagctttcttgtacaaagtggccgttaacggatcggccagaatccgg4440
taagtgactagggtcacgtgaccctagtcacttaaattcggccagaatggccatctggat4500
tcagcaggcctagaaggcccggaccgattaaactttaattcggtccgggttacctctaga4560
aagcttgtcgacctgcaggtacaagtacaagggacttgtgagttgtaaggctgtatttac4620
aatagtgaaaagagaatcatctgggtgattgggtttttagtccccagtgacgaattaaag4680
gtttgaattcttagtatgtttgggaatcaattaggaatttcgttttggactttccaaagc4740
aattattcactttttcattcattaaatgtgactaaaaaattgttatttctccattggcca4800
ggatgcatcgtttatataaacataaccttagtgaaagcagtgttttcatgtgacagcggc4860
agactatatcttaaacaaaattacttgtaaagaaagataccgttaggaaaaaaatgaaaa4920
gaaaattgaagctatcacttgtttactttcctaatatctttcaagaatacaatgtggtga4980
atttcaattttccctacatatgtataccgtcagcctgacgcaacttatgaaacttctctt5040
tctttcatttgatgtatatataaagacacattatatataaagaaactttatatatatctc5100
catcatattttagtacttgctactatgtaaaattagctgttggaagtatctcaagaaaca5160
tttaatttattgaaccaagcattaaccattcatctacatttgagttctaaaataaatctt5220
aaatgatgtggaggaagggaaattgttaattatttccctcttctcctacatggatatacc5280
tgaaacatgcaatggatggattagattttaacatttgcagcctgagaagttcactgactt5340
tcctccagctattttatgtgtgcccgccaccatttatagctcatgattgtagctgaactg5400
caaaaactgcatcgattgcaaactgaaattgagaatctcttttcaactttatatgctgat5460
tgatgcatgctgagcatgctatactagtactcgaagttcctatatgtagactttgttact5520
gcctaatatactttgtgtttgttctcaagttcttattttatttcatattttttcctataa5580
aaggttaatggctctataaaggttgagtgacggatccggtcacctaagtgactagggtca5640
cgtgaccctagtcacttattcccgggcaactttattatacaaagttgatagatctcgaat5700
tcattccgattaatcgtgg5719
<210>61
<211>22
<212>dna
<213>人工序列
<220>
<223>sams:hpt正向引物sams-76f
<400>61
aggcttgttgtgcagtttttga22
<210>62
<211>22
<212>dna
<213>人工序列
<220>
<223>fam标记的sams:hpt探针frt1i-63t
<400>62
tggactagtggaagttcctata22
<210>63
<211>21
<212>dna
<213>人工序列
<220>
<223>sams:hpt反向引物frt1i-41f
<400>63
gcggtgagttcaggctttttc21
<210>64
<211>19
<212>dna
<213>人工序列
<220>
<223>cas9正向引物cas9-f
<400>64
ccttcttccaccgccttga19
<210>65
<211>19
<212>dna
<213>人工序列
<220>
<223>fam标记的cas9探针cas9-t
<400>65
aatcattcctggtggagga19
<210>66
<211>21
<212>dna
<213>人工序列
<220>
<223>cas9反向引物cas9-r
<400>66
tgggtgtctctcgtgcttttt21
<210>67
<211>26
<212>dna
<213>人工序列
<220>
<223>dd43正向引物dd43-f
<400>67
ttctagaataccctccgtacgtacaa26
<210>68
<211>19
<212>dna
<213>人工序列
<220>
<223>fam标记的dd43探针dd43-t
<400>68
caagggacttgtgagttgt19
<210>69
<211>26
<212>dna
<213>人工序列
<220>
<223>反向引物dd43-r;
<400>69
cccagatgattctcttttcactattg26
<210>70
<211>26
<212>dna
<213>人工序列
<220>
<223>正向引物dd43-lb
<400>70
gtgtagtccattgtagccaagtcacc26
<210>71
<211>24
<212>dna
<213>人工序列
<220>
<223>反向引物dd43-rb
<400>71
caaaccggagagagaggaagaacc24
<210>72
<211>23
<212>dna
<213>人工序列
<220>
<223>正向引物dd43lb-1
<400>72
caatgtctcctgctctgtcctgc23
<210>73
<211>24
<212>dna
<213>人工序列
<220>
<223>反向引物dd43rb-1
<400>73
agtttcataagttgcgtcaggctg24
<210>74
<211>2098
<212>dna
<213>大豆
<400>74
gtgtagtccattgtagccaagtcaccaatatcttgttcccctccttggtttggcataaat60
tgattttcatggctcttctcggtcgaaactggagctaattcacccttagtctctcttaaa120
attctggctgtaagaaacaccacagaacacataaattataaactaattataatttgaaga180
gtaaaatatgtttttactcttatgatttaattagtgtagttttaattttctccttttttt240
aaaaaattttggtattcataaatttcaattttttaaaaataattgttgttacccgttaat300
gataacgggatatgttatgttaccactaaatcggacaaaaaaaattcaaaacttttataa360
ggattaaaattaacaaaaatattttaaaaaaatctaacctcaataaagttaaatttataa420
gcacaaaataatacttttaagcctaatttggcaagacacaagcaagctcacctgtagcat480
taatagaaaggaagcaaagcaagagaaaagcaaccagaaggaagcgtttgcttggtgaca540
cagccatcttacttgaatttatggtattactgagaaaccttgatcttgcttcaaaatctt600
ctagttaccctctttttataggcagaaagagaactagctagttgccaataggatatgagg660
acatgtggtgcaatgcactcactcttcaaggacaagaaaaacaatggctacaattgtggt720
tcaaatcaatgtctcctgctctgtcctgcctgaaaatgacacccttttgcttggaaaaga780
ggatcaaagctaagaacaggagtggcttcattcccttcatgtaaccaaacactttcgcat840
tctgtcattcgtgaatcagcaaaatctgcaaccaaaaatatatggtgcctaaataaaaga900
aataaaataatttagagttgcggactaaaataataaacaaaagaaatatattataatcta960
gaattaatttaggactaaaagaagaggcagactccaattcctcttttctagaataccctc1020
cgtacgtacaagtacaagggacttgtgagttgtaaggctgtatttacaatagtgaaaaga1080
gaatcatctgggtgattgggtttttagtccccagtgacgaattaaaggtttgaattctta1140
gtatgtttgggaatcaattaggaatttcgttttggactttccaaagcaattattcacttt1200
ttcattcattaaatgtgactaaaaaattgttatttctccattggccaggatgcatcgttt1260
atataaacataaccttagtgaaagcagtgttttcatgtgacagcggcagactatatctta1320
aacaaaattacttgtaaagaaagataccgttaggaaaaaaatgaaaagaaaattgaagct1380
atcacttgtttactttcctaatatctttcaagaatacaatgtggtgaatttcaattttcc1440
ctacatatgtataccgtcagcctgacgcaacttatgaaacttctctttctttcatttgat1500
gtatatataaagacacattatatataaagaaactttatatatatctccatcatattttag1560
tacttgctactatgtaaaattagctgttggaagtatctcaagaaacatttaatttattga1620
accaagcattaaccattcatctacatttgagttctaaaataaatcttaaatgatgtggag1680
gaagggaaattgttaattatttccctcttctcctacatggatatacctgaaacatgcaat1740
ggatggattagattttaacatttgcagcctgagaagttcactgactttcctccagctatt1800
ttatgtgtgcccgccaccatttatagctcatgattgtagctgaactgcaaaaactgcatc1860
gattgcaaactgaaattgagaatctcttttcaactttatatgctgattgatgcatgctga1920
gcatgctatactagtactcgaagttcctatatgtagactttgttactgcctaatatactt1980
tgtgtttgttctcaagttcttattttatttcatattttttcctataaaaggttaatggct2040
ctataaaggttgagtgacatatatatactataaaggttcttcctctctctccggtttg2098
<210>75
<211>60
<212>dna
<213>大豆
<400>75
agccttacaactcacaagtcccttgtacttgtacgtacggagggtattctagaaaagagg60
<210>76
<211>59
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_12_1c
<400>76
agccttacaactcacaagtcccttgtacttgtactacggagggtattctagaaaagagg59
<210>77
<211>59
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_5_6c
<400>77
agccttacaactcacaagtcccttgtacttgtagtacggagggtattctagaaaagagg59
<210>78
<211>58
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_5_3a
<400>78
agccttacaactcacaagtcccttgtacttgtgtacggagggtattctagaaaagagg58
<210>79
<211>58
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_12_4c
<400>79
agccttacaactcacaagtcccttgtacttgcgtacggagggtattctagaaaagagg58
<210>80
<211>57
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_1_20b
<400>80
agccttacaactcacaagtcccttgtacttggtacggagggtattctagaaaagagg57
<210>81
<211>57
<212>dna
<213>人工序列
<220>
<223>pcr片段,3478_5_1a
<400>81
agccttacaactcacaagtcccttgtacttgttacggagggtattctagaaaagagg57
<210>82
<211>56
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_5_1a
<400>82
agccttacaactcacaagtcccttgtacttgtacggagggtattctagaaaagagg56
<210>83
<211>55
<212>dna
<213>人工序列
<220>
<223>pcr片段,3478_12_4b
<400>83
agccttacaactcacaagtcccttgtactttacggagggtattctagaaaagagg55
<210>84
<211>55
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_5_11b
<400>84
agccttacaactcacaagtcccttgtactgtacggagggtattctagaaaagagg55
<210>85
<211>54
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_5_4a
<400>85
agccttacaactcacaagccccttgtacttacggagggtattctagaaaagagg54
<210>86
<211>52
<212>dna
<213>人工序列
<220>
<223>pcr片段,33475_12_2a
<400>86
agccttacaactcacaagtcccttgtatacggagggtattctagaaaagagg52
<210>87
<211>52
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_5_14a
<400>87
agccttacaactcacaagtcccttgtgtacggagggtattctagaaaagagg52
<210>88
<211>50
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_1_17a
<400>88
agccttacaactcacaagtcccttgtacggagggtattctagaaaagagg50
<210>89
<211>49
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_5_16c
<400>89
agccttacaactcacaagtccctttacggagggtattctagaaaagagg49
<210>90
<211>48
<212>dna
<213>人工序列
<220>
<223>pcr片段,3478_8_1a
<400>90
agccttacaactcacaagtcccttacggagggtattctagaaaagagg48
<210>91
<211>47
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_5_5c
<400>91
agccttacaactcacaagtccctacggagggtattctagaaaagagg47
<210>92
<211>43
<212>dna
<213>人工序列
<220>
<223>pcr片段,3478_5_1b
<400>92
agccttacaactcacaagtcccttgtacttgtaagaaaagagg43
<210>93
<211>49
<212>dna
<213>人工序列
<220>
<223>pcr片段,3478_6_7c
<400>93
agccttacaactcacaagtcctaaattaaaggttattctagaaaagagg49
<210>94
<211>227
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_1_10a
<400>94
agccttacaactcacaagtcccttgtacttgtagaatccagttcataaaacaagtgacac60
acaacagatatgaactggactacgtcgaacccacaaatcccacaaagcgcgtgaaatcaa120
atcgctcaaaccacaaaaaagaacaacgcgtttgttacacgctaataccaaaattatacc180
caaatcttaagctatttatgcgtacggagggtattctagaaaagagg227
<210>95
<211>97
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_1_10c
<400>95
agccttacaactcacaagtcccttgtacttgtaatgctcccctctaaactcgtatcgctt60
cagagttgagagtacggagggtattctagaaaagagg97
<210>96
<211>183
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_5_16a
<400>96
agccttacaactcacaagtcccttgtatatagatacccacaaaataagtaaacccgatcc60
aaaatcttaaatgatgtggaggaagggaaattgttaattattcccctcttctcctacatg120
gatatacctgaaacatgcaatggatggattagattttgtacggagggtattctagaaaag180
agg183
<210>97
<211>234
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_5_3b
<400>97
agccttacaactcacaagtcccttgtacttgtaccaggggatgttttttatttacattca60
cgtcttttggaaagagccgctaaattaagttctcagttaggcgaaggaagtatgactgct120
ttaccaatagttgaaactcaatcgggagatgtttcagcttatattcctactaatgtaatt180
tccattacagatggccaaatattcttacgtacggagggtattctagaaaagagg234
<210>98
<211>280
<212>dna
<213>人工序列
<220>
<223>pcr片段,3475_1_11a
<400>98
agccttacaactcacaagtcccttgtacttgtaccgaaaatttcagccataaaaaaagtt60
ataatagaatttaaagcaaaagtttcattttttaaacatatatactgacacgctccgata120
aaaatagaggcaagtccgacaacgtcccctccgaggaggtcgtgaagaagatgaaaaact180
actggagacagctcttgaacgccaagctcatcacccagcgtaagctcgacaacctgacta240
aggctgagagaggtgtacggagggtattctagaaaagagg280
<210>99
<211>250
<212>dna
<213>人工序列
<220>
<223>pcr片段,3478_1_9b
<400>99
agccttacaactcacaagtcccttgtacttgtactggatttggtgagggatgcttccgtt60
gtcgaaggttctctgcttcctcaacaggtcctctctgttcaacttcaccaacagctcctc120
ggtaccgtccatcttctcaaggatgaagatcgagtgcttcgactccgtcgagatctctgg180
tgtcgaggacaggttcaacgcctcccttgggacttgccacgatcgtacggagggtattct240
agaaaagagg250
<210>100
<211>161
<212>dna
<213>人工序列
<220>
<223>pcr片段,3478_1_9a
<400>100
agccttacaactcacaagtcccttatgacctcaaaaaaaagattcacctccaacacacca60
aataactcgaaaatctctttcctattctctagaaagtataggaacttccactagtccatg120
aaaaagcctgaactcgtacggagggtattctagaaaagagg161
<210>101
<211>185
<212>dna
<213>人工序列
<220>
<223>pcr片段,3478_5_4b
<400>101
agccttacaactcacaagtcccttgtacttgtacacctggggcatggagagcaccttcct60
cacagtagcgaaatccctccctttgtcccacacgatctctccagtctcaccgttggtctc120
gatcagtgggcgcttcctgatctcaccgttggcgagagtgtacggagggtattctagaaa180
agagg185
<210>102
<211>212
<212>dna
<213>人工序列
<220>
<223>pcr片段,3478_9_1c
<400>102
agccttacaactcacaagtcccttgtacttgtgctaggttagccgaaagatggttatcgg60
ttcaaggacgcaaggtgcccctgctttttcagggtaataaggggtagagaaaatgcctcg120
agccaaagttcgagtaccaggcgctacagcgctgaagtaatccatgccatactcccagga180
aaagccgtacggagggtattctagaaaagagg212
<210>103
<211>231
<212>dna
<213>人工序列
<220>
<223>pcr片段,3478_9_1a
<400>103
agccttacaactcacaagtcccttgtacttgtactcaagttcttattttatttcatattt60
tttcctataaaaggttaatggctctataaaggttgagtgacggatccggtcacctaagtg120
actagggtcacgtgaccctagtcacttattcccgggcaactttattatacaaagttgata180
gatctcgaattcattccgattaatcgtggcgagggtattctagaaaagagg231
- 还没有人留言评论。精彩留言会获得点赞!