用于产生苄基异喹啉生物碱(BIA)的重组宿主细胞和苄基异喹啉生物碱(BIA)的新的制造方法
本发明涉及用于产生苄基异喹啉生物碱(bia)的重组宿主细胞和苄基异喹啉生物碱(bia)的新的制造方法。
背景技术:
苄基异喹啉生物碱(bia)衍生物是包含吗啡、可待因等镇痛药、黄连素等抗菌剂之类的有用药品的多样的化合物组。这些苄基异喹啉生物碱衍生物大部分是在各种植物中由酪氨酸经由四氢罂粟林(thp,papaveroline)、去甲乌药碱(norcoclaurine)、网脉番荔枝碱(reticuline)等苄基异喹啉生物碱(bia)而合成的。即,四氢罂粟林(thp)、去甲乌药碱、网脉番荔枝碱也是多种苄基喹啉生物碱衍生物的生物合成途径中的重要的中间体。这样的四氢罂粟林(thp)、去甲乌药碱、网脉番荔枝碱虽然不直接用于疾病的治疗,但在工业上用作药品原料,可制造氧可酮、羟吗啡酮、纳布啡、纳洛酮、纳曲酮、丁丙诺啡、埃托啡等。
一直以来苄基异喹啉生物碱(bia)及其衍生物的生产基本上依赖于从植物中提取。另外,一些苄基异喹啉生物碱(bia)通过全合成而被化学合成(参照非专利文献1)。然而,从生产的稳定性、高效性的观点出发,需要开发出其它制造方法。例如,使用了微生物的生物生产(bioproduction)由于不包含其它植物代谢产物,因此能够高效地生产所需的苄基异喹啉生物碱(bia),而受到注目(参照非专利文献2~4)。然而,其收率每升低于10mg,为了满足工业上的需求,要求对利用生物生产的方法进行进一步优化。
现有技术文献
非专利文献
非专利文献1:gatesm.etal,thesynthesisofmorphine,jamchemsoc74,1109-1110(1952)
非专利文献2:galanie,s.,thodey,k.,trenchard,i.j.,filsingerinterrante,m.&smolke,c.d.completebiosynthesissofopioidsinyeast,science349,1095-1100(2015)
非专利文献3:nakagawa,a.etal.(r,s)-tetrahydropapaverolineproductionbystepwisefermentationusingengineeredescherichiacoli.sci.rep.4,6695(2014)
非专利文献4:nakagawa,a.etal.totalbiosynthesisofopiatesbystepwisefermentationusingengineeredescherichiacoli.nat.commun.7,10390(2016)
技术实现要素:
发明要解决的问题
在这样的情况下,本发明的目的在于,提供能够高效地生产苄基异喹啉生物碱(bia)的微生物、以及使用了其的苄基异喹啉生物碱(bia)的制造方法。具体而言,提供能够高效地且容易地生产作为多种苄基异喹啉生物碱(bia)衍生物的生物合成途径的中间体的四氢罂粟林(thp)、去甲乌药碱、网脉番荔枝碱等苄基异喹啉生物碱(bia)的重组宿主细胞,此外,目的在于提供使用该宿主细胞高效地且容易地制造四氢罂粟林(thp)、去甲乌药碱、网脉番荔枝碱等苄基异喹啉生物碱(bia)的方法。
用于解决问题的方案
本发明人等为了解决上述课题,在使用了微生物的四氢罂粟林(thp)、去甲乌药碱、网脉番荔枝碱等苄基异喹啉生物碱(bia)的制造方法中,使用基于合成生物学方法设计出新的的生物合成途径,成功地鉴定了作为双功能酶的芳香族醛合成酶(aas)。另外发现,通过在酪氨酸脱羧酶(tydc)、多巴脱羧酶(ddc)等芳香族氨基酸脱羧酶(aaad)的特定的残基中导入突变,从而这些酶也显示出4-hpaas、dhpaas样的活性。即,本发明的主旨如下所示。
[1]一种用于产生苄基异喹啉生物碱(bia)的重组宿主细胞,其表达了异种的芳香族醛合成酶(aas)、芳香族氨基酸脱羧酶(aaad)的野生型或突变体。
[2]根据[1]所述的重组宿主细胞,其中,苄基异喹啉生物碱(bia)为四氢罂粟林(thp)、去甲乌药碱、3-羟基乌药碱、3-羟基-n-甲基乌药碱和/或网脉番荔枝碱。
[3]根据[1]或[2]所述的重组宿主细胞,其中,上述异种的种属为昆虫、植物或微生物。
[4]根据[3]所述的重组宿主细胞,其中,上述异种的种属为选自由家蚕(bombyxmori)、佛罗里达弓背蚁(camponotusfloridanus)、西方蜜蜂(apismellifera)、埃及伊蚊(aedesaegypti)、及黑腹果蝇(drosophilamelanogaster)组成的组中的昆虫、罂粟(papaversomniferum)或恶臭假单胞菌(pseudomonasputida)。
[5]根据[1]至[4]中任一项所述的重组宿主细胞,其中,宿主细胞为大肠杆菌。
[6]根据[1]至[5]中任一项所述的重组宿主细胞,其中,芳香族醛合成酶(aas)为3,4-二羟基苯基乙醛合成酶(dhpaas)、4-羟基苯基乙醛合成酶(4-hpaas)。
[7]根据[6]所述的重组宿主细胞,其中,芳香族醛合成酶(aas)为昆虫来源,且芳香族醛合成酶(aas)的突变体中的突变为选自由asn192his、phe79tyr和tyr80phe组成的组中的至少1个。
[8]根据[6]所述的重组宿主细胞,其中,芳香族氨基酸脱羧酶(aaad)为植物来源的酪氨酸脱羧酶(tydc),且酪氨酸脱羧酶(tydc)的突变体中的突变为选自由leu205asn、phe99tyr和tyr98phe组成的组中的至少1个,或为选自由his203asn、phe101tyr和tyr100phe组成的组中的至少1个。
[9]根据[6]所述的重组宿主细胞,其中,芳香族氨基酸脱羧酶(aaad)为微生物来源的多巴脱羧酶(ddc),且多巴脱羧酶(ddc)的突变体中的突变为选自由tyr79phe、phe80tyr和his181asn组成的组中的至少1个。
[10]根据[1]至[9]中任一项所述的重组宿主细胞,其还表达了去甲乌药碱合成酶(ncs)。
[11]根据[1]至[10]中任一项所述的重组宿主细胞,其还表达了选自由去甲乌药碱6-o-甲基转移酶(6’omt)、3’-羟基-n-甲基-(s)-乌药碱-4’-o-甲基转移酶(4’omt)、乌药碱-n-甲基转移酶(cnmt)、及n-甲基乌药碱3-羟化酶组成的组中的至少1种酶。
[12]一种苄基异喹啉生物碱(bia)的制造方法,其包括在含有l-多巴或酪氨酸的培养基中培养[1]至[11]中任一项所述的重组宿主细胞的工序。
[13]一种苄基异喹啉生物碱(bia)的制造方法,其包括在无细胞体系中使芳香族醛合成酶(aas)、芳香族氨基酸脱羧酶(aaad)的野生型或突变体作用于l-多巴或酪氨酸的工序。
[14]根据[13]所述的制造方法,其特征在于,芳香族醛合成酶(aas)、芳香族氨基酸脱羧酶(aaad)的野生型或突变体是由[1]至[11]中任一项所述的重组宿主细胞得到的酶。
发明的效果
根据本发明,通过使用表达作为双功能酶的芳香族醛合成酶(aas)等的重组宿主细胞,从而能够高效地且容易地生产四氢罂粟林(thp)、去甲乌药碱、3-羟基乌药碱、3-羟基-n-甲基乌药碱、网脉番荔枝碱等苄基异喹啉生物碱(bia)。
附图说明
图1是示出m-途径检索中发现的用于产生网脉番荔枝碱的thp合成途径的图。
图2是示出对称性ddc-dhpaas途径和mao介导非对称性途径中的thp的预测收率的图。
图3是示出aaad和dhpaas的结构分析的图。左边示出与plp形成了复合物的黑腹果蝇(d.melanogaster)来源的ddc,中间示出与plp-4-hpaa形成了复合物的罂粟(p.somniferum)tydc1,右边示出与plp-dopa形成了复合物的家蚕(b.mori)来源dhpaas的结构。
图4是对昆虫的dhpaa的序列进行系统发生学分类的结果。
图5中,图5-1和图5-2是关于家蚕(b.mori)的野生型和突变体dhpaas的功能的比较的图。
图6是对利用家蚕(b.mori)的野生型和突变体dhpaas由l-多巴产生h2o2进行动力学分析的图。
图7是示出利用家蚕(b.mori)的野生型和突变体dhpaas在体外产生多巴胺、dhpaa和thp的图。
图8是对利用突变体dhpaas由l-多巴产生thp的机理进行说明的图。
图9中,图9-1是示出利用dhpaas在体内产生多巴胺、dhpaas和thp的图。图9-2是示出所产生的(r,s)-thp的手性lc-ms分析的结果的图。
图10是示出在体内产生thp和网脉番荔枝碱的图。
图11是示出在体内产生thp、网脉番荔枝碱和2种中间体的图。
图12是示出在体内产生thp、多巴胺的图。
图13是示出在体内产生去甲乌药碱的图。
图14是示出在体内产生去甲乌药碱的图。
图15是示出在体内产生4-hpaa、l-多巴、thp、去甲乌药碱、网脉番荔枝碱的路线的图。
图16是示出图15的路线图中的4-hpaa、l-多巴、thp、去甲乌药碱、网脉番荔枝碱的体内产生量的图。
图17是示出在体内产生thp、3hnmc、网脉番荔枝碱的路线的图。
图18是示出图17的路线图中的thp、3hnmc、网脉番荔枝碱的体内产生量的图。
具体实施方式
以下对本发明的用于产生苄基异喹啉生物碱(bia)的重组宿主细胞、及苄基异喹啉生物碱(bia)的新的制造方法进行详细地说明。需要说明的是,本说明书中,dna、载体的制备等分子生物学方法只要没有特别声明则可以利用本领域技术人员公知的一般实验书中记载的方法或依据其的方法而进行。另外,本说明书中使用的术语只要没有特别声明则可解释为该技术领域中通常使用的意思。需要说明的是,本发明中苄基异喹啉生物碱(bia)是指具有苄基异喹啉结构的化合物。例如可列举出各种植物中的四氢罂粟林(thp)、去甲乌药碱、3-羟基乌药碱、3-羟基-n-甲基乌药碱、网脉番荔枝碱等,但不限定于这些。
<重组宿主细胞>
本发明的重组宿主细胞是表达芳香族醛合成酶(aas)、芳香族氨基酸脱羧酶(aaad)的野生型或突变体的、用于产生苄基异喹啉生物碱(bia)、特别是四氢罂粟林(thp)、去甲乌药碱、3-羟基乌药碱、3-羟基-n-甲基乌药碱和/或网脉番荔枝碱的重组宿主细胞。以下对本发明的重组宿主细胞进行详细地说明。
本发明的重组宿主细胞所表达的芳香族醛合成酶(aas)是指催化芳香族氨基酸的脱羧基化和氨基氧化的双功能酶。具体而言,是具有催化由l-多巴或酪氨酸转化为多巴胺、及dhpaa或4-hpaa的功能的酶。上述中得到的多巴胺、及dhpaa或4-hpaa彼此结合而生成thp或去甲乌药碱。根据系统发生分析,可认为aas是从芳香族氨基酸脱羧酶(aaad、ec4.1.1.28)分支出的酶,两者具有结构上的相似性,且在依赖吡哆醛5’-磷酸(plp)作为辅因子方面是共通的。
作为本发明中的aas,只要具有上述功能就没有特别限定,例如可列举出苯基乙醛合成酶(paas、keggec4.1.1.109)、4-羟基苯基乙醛合成酶(4-hpaas、keggec4.1.1.108)等在植物中被研究、根据kegg而分类的植物来源aas、昆虫来源的3,4-二羟基苯基乙醛合成酶(dhpaas、keggec4.1.1.107)酶、这些以外的例如iaas(indole-3-acetaldehydesynthase;吲哚-3-乙醛合成酶)等。需要说明的是,种属不受限定,包括包含动物、植物、细菌在内的多个种属。3,4-二羟基苯基乙醛合成酶催化l-多巴的氧化性脱羧基化而产生dhpaa。另外,催化l-多巴的氨基氧化而产生多巴胺。作为aas,从由l-多巴向thp转化的效率的观点出发,上述中优选昆虫来源的dhpaas。昆虫来源的dhpaas与l-多巴的结合特异性高,可认为因此产生dhpaa、产生多巴胺(da)的效率增高。另外,作为aas,从由酪氨酸向去甲乌药碱转化效率的观点出发,也优选植物来源的4-hpaas。
作为上述昆虫,可列举出家蚕、佛罗里达弓背蚁、西方蜜蜂、埃及伊蚊、黑腹果蝇等,这些当中,从本发明的效果的观点出发,优选家蚕。
作为上述植物,可列举出罂粟、拟南芥(arabidopsisthaliana)、琴叶拟南芥(arabidopsislyrata)、芜青(brassicarapa)、亚麻荠(camelinasativa)、长蒴黄麻(corchorusolitorius)、甘蓝(brassicaoleracea)、树树菜(brassicacretica)、欧洲油菜(brassicanapus)、荠菜(capsellarubella)、山嵛菜(eutremasalsugineum)、糙叶山黄麻(parasponiaandersonii)、欧芹(petroselinumcrispuma)、欧洲甜樱桃(prunusavium)、东京樱花(prunusyedoensis)、扁桃(prunusdulcis)、梅花(prunusmume)、桃(prunuspersica)、东京樱花(prunusyedoens)、萝卜(raphanussativus)、醉蝶花(tarenayahassleriana)、异色山黄麻(tremaorientale)、枣(ziziphusjujuba)、苹果(malusdomestica)、枇杷(eriobotryajaponica)、黄麻(corchoruscapsularis)、川桑(morusnotabilis)、白梨(pyrusxbretschneideri)、银白杨(populusalba)、胡桃(juglansregia)、温州蜜柑(citrusunshiu)、橙子(citrussinensis)、欧洲栓皮栎(quercussuber)、土瓶草(cephalotusfollicularis)、大桉(eucalyptusgrandis)、野草莓(fragariavesca)、毛果杨(populustrichocarpa)、榴莲(duriozibethinus)、木薯(manihotesculenta)、榴梿(duriozibethinus)、毛果杨(populustrichocarpa)、胡桃(juglansregia)、木薯(manihotesculenta)、橡胶树(heveabrasiliensis)、甜橙(citrussinensis)、大桉(eucalyptusgrandis)、榴莲(duriozibethinus)、木薯(manihotesculenta)、橡胶树(heveabrasiliensis)、克莱门柚(citrusclementina)、川桑(morusnotabilis)、番木瓜(caricapapaya)、月季(rosachinensis)、葡萄(vitisvinifera)、胡杨(populuseuphratica)、月季(rosachinensis)、葡萄(vitisvinifera)、猕猴桃(actinidiachinensis)、胡杨(populuseuphratica)、牵牛花(ipomoeanil)、矮牵牛(petuniahybrid)等,这些当中,从本发明的效果的观点出发,优选罂粟。
作为上述微生物,可列举出恶臭假单胞菌(p.putida)、詹氏甲烷球菌(methanocaldococcusjannaschii)等,这些当中,从本发明的效果的观点出发,优选恶臭假单胞菌(p.putida)。
作为本发明中的aas,优选为活性中心附近的氨基酸残基被置换为在多巴脱羧酶(ddc:dopadecarboxylase)中可见的氨基酸残基的突变体。
具体而言,例如昆虫来源的dhpaas中,优选phe79tyr、tyr80phe、asn192his的突变,可以具有这些突变中的任意1个,可以具有任意2个,也可以具有全部3个突变。这些当中,从由l-多巴向thp转化的效率的观点出发,优选:具有上述全部3个突变的phe79tyr-tyr80phe-asn192hisdhpaas、具有phe79tyr-tyr80phe这2个突变的phe79tyr-tyr80phedhpaas、仅具有asn192his的突变的asn192hisdhpaas,更优选phe79tyr-tyr80phe-asn192hisdhpaas、asn192hisdhpaas。
本发明的重组宿主细胞所表达的芳香族氨基酸脱羧酶(aaad)是催化芳香族氨基酸的脱羧基化的酶。具体而言,是具有催化由l-多巴或酪氨酸转化为多巴胺或4-hpaa的功能的酶。具体而言,可列举出酪氨酸脱羧酶(tydc)、多巴脱羧酶(ddc)、苯基丙氨酸脱羧酶(pdc)、色氨酸脱羧酶(tdc)等。
作为本发明的重组宿主细胞所表达的芳香族氨基酸脱羧酶(aaad)的种属,可以适宜列举出与关于上述aas而记载的种属相同的种属。
本发明的重组宿主细胞所表达的芳香族氨基酸脱羧酶(aaad)为植物来源的tydc1时,优选phe99tyr、tyr98phe、leu205asn的突变,可以具有这些突变中的任意1个,可以具有任意2个,也可以具有全部3个突变。这些当中,从由酪氨酸转化为去甲乌药碱的效率的观点出发,优选具有上述全部3个突变的phe99tyr-tyr98phe-leu205asntydc1。另一方面,为tydc3的情况下,优选phe101tyr、tyr100phe、his203asn的突变,可以具有这些突变中的任意1个,可以具有任意2个,也可以具有全部3个突变。这些当中,从由酪氨酸向去甲乌药碱转化的效率的观点出发,优选具有上述全部3个突变的phe101tyr-tyr100phe-his203asntydc3。
需要说明的是,作为昆虫的家蚕(bombyxmori)的dhpaas的第79、80和192位的活性位点残基在芳香族氨基酸脱羧酶(aaad)、芳香族醛合成酶(aas)、dhpaas及其它相关蛋白中全部在结构上是保守的。其中,残基的编号根据蛋白质的尺寸差异、根据种属而不同。例如,家蚕(bombyxmori)的dhpaas的phe79对应于恶臭假单胞菌(pseudomonasputida)的ddc的tyr79、罂粟(papaversomniferum)的tydc1的tyr98、罂粟(papaversomniferum)的tydc3的tyr100。家蚕(bombyxmori)的dhpaas的tyr80对应于恶臭假单胞菌(pseudomonasputida)的ddc的phe80、罂粟(papaversomniferum)的tydc1的phe99、罂粟(papaversomniferum)的tydc3的phe101。家蚕(bombyxmori)的dhpaas的asn192对应于恶臭假单胞菌(pseudomonasputida)的ddc的his181、罂粟(papaversomniferum)的tydc1的leu205、罂粟(papaversomniferum)的tydc3的his203。需要说明的是,例如,罂粟的tydc中还有tydc2、4~9,但是对应于tydc1的leu205的,在tydc5、tydc6、tydc8、tydc9中是his205,在tydc2、tydc7中是his203。
本说明书中,时常会提及家蚕(bombyxmori)的3,4-二羟基苯基乙醛合成酶(dhpaas)的氨基酸残基的编号,但本发明适用于与上述结构上保守的残基相对应的所有氨基酸位置。为了确定该结构上保守的残基而可以参照结构图。另外,对于所对应的位置的氨基酸的编号不同的例子,可以参照序列比对图(图3和4)。
本发明的重组宿主细胞具有编码上述的aas(野生型和各种突变体)的基因。作为这样的基因,昆虫来源的dhpaas的情况,例如可列举出具有序列号1(dhpaas野生型)、序列号2(asn192hisdhpaas突变体)、序列号3(phe79tyr-tyr80phedhpaas突变体)、序列号4(phe79tyr-tyr80phe-asn192hisdhpaas突变体)所示的核苷酸序列的基因。另外,所对应的蛋白的氨基酸序列分别由序列号5(dhpaas野生型)、序列号6(asn192hisdhpaas突变体)、序列号7(phe79tyr-tyr80phedhpaas突变体)、序列号8(phe79tyr-tyr80phe-asn192hisdhpaas突变体)表示。需要说明的是,为了改善本发明的重组宿主细胞中上述野生型和突变体dhpaas的蛋白生产效率,可以使用sumo标签表达系统。作为此时的各自的氨基酸序列,可以采用序列号9(dhpaas野生型)、序列号10(asn192hisdhpaas突变体)、序列号11(phe79tyr-tyr80phedhpaas突变体)、序列号12(phe79tyr-tyr80phe-asn192hisdhpaas突变体)所示的氨基酸序列。
即,本发明的重组宿主细胞所具有的aas基因为dhpaas时,优选为以下(a)、(b)或(c)的dna。
(a)由序列号1~4中任一项的核苷酸序列组成的dna。
(b)与由下述核苷酸序列组成的dna在严格条件下进行杂交、且编码具有dhpaas的酶活性(双功能)的蛋白质的dna,所述核苷酸序列为与由(a)的核苷酸序列组成的dna互补的核苷酸序列。
(c)由与序列号1~4中任一项的核苷酸具有70%以上、优选为80%以上、更优选为90%以上、进一步优选为95%以上、特别优选为98%以上的同源性的核苷酸序列组成、在野生型的序列中导入了上述突变、且编码具有dhpaas的酶活性(双功能)的蛋白质的dna。
另外,本发明的重组宿主细胞具有编码上述的芳香族氨基酸脱羧酶(aaad)的基因。作为这样的基因,芳香族氨基酸脱羧酶(aaad)为植物来源tydc1的情况,作为野生型,可列举出具有序列号15的氨基酸序列、且具有与其对应的序列号16的核苷酸序列者。对于上述的突变,通过使用序列号17和序列号18的引物,从而能够合成导入了phe99tyr、tyr98phe的突变的核苷酸,另外,通过使用序列号19和序列号20的引物,从而能够合成导入了leu205asn的突变的核苷酸。另外,作为本发明的重组宿主细胞所具有的基因,芳香族氨基酸脱羧酶(aaad)为植物来源的tydc3的情况,作为野生型,可列举出具有序列号21的氨基酸序列、且具有与其对应的序列号22的核苷酸序列者。对于上述的突变,通过使用序列号23和序列号24的引物,从而能够合成导入了phe101tyr、tyr100phe的突变的核苷酸,另外,通过使用序列号25和序列号26的引物,从而能够合成导入了his203asn的突变的核苷酸。
本发明的重组宿主细胞优选:除了具有编码上述的aas(野生型和各种突变体)、或aaad(野生型和各种突变体)的基因之外,还具有编码用于由thp、去甲乌药碱合成网脉番荔枝碱所需的酶的基因。
作为这样的酶,例如可列举出去甲乌药碱合成酶(ncs)。去甲乌药碱合成酶(ncs)是由多巴胺和dhpaa、或由多巴胺和4-hpaa合成去甲乌药碱、thp的酶。本发明的重组宿主细胞优选包含编码去甲乌药碱合成酶(ncs)的基因。
进而,作为这样的酶,可列举出去甲乌药碱6-o-甲基转移酶(6’omt)、3’-羟基-n-甲基-(s)-乌药碱-4’-o-甲基转移酶(4’omt)、乌药碱-n-甲基转移酶(cnmt)、n-甲基乌药碱3-羟化酶(nmch)等。本发明的重组宿主细胞更优选具有编码去甲乌药碱6-o-甲基转移酶(6’omt)、3’-羟基-n-甲基-(s)-乌药碱-4’-o-甲基转移酶(4’omt)和乌药碱-n-甲基转移酶(cnmt)的基因中的全部。
严格条件是指仅发生特异性杂交而不发生非特异性杂交的条件。这样的条件通常是6m脲、0.4%sds、0.5xssc程度。通过杂交而得到的dna优选与由上述(a)的核苷酸序列组成的dna具有60%以上的高度同源性,进一步优选具有80%以上的同源性。
同源性是指2个多肽或多核苷酸之间的序列的相似程度,通过对比较对象的氨基酸序列或碱基序列的整个区域以最佳状态(序列处于最大匹配度的状态)对齐的2个序列进行比较,从而确定。同源性的数值(%)可如下算出:确定两个(氨基酸或碱基)序列中存在的相同的氨基酸或碱基,从而确定匹配位点的数量,接着用该匹配位点的数除以比较对象的序列区域内的氨基酸或碱基的总数,用得到的数值乘以100,从而算出。作为用于得到最佳的比对和同源性的算法,可列举出本领域技术人员通常可利用的各种算法(例如blast算法、fasta算法等)。氨基酸序列的同源性例如可使用blastp、fasta等的序列解析软件进行确定。碱基序列的同源性可使用blastn、fasta等软件进行确定。
上述基因可以通过本领域技术人员公知的pcr或杂交技术、或使用dna合成机等的人工合成方法而获得。基因序列的确定可以通过本领域技术人员公知的方法并使用测序仪而进行。
本发明中使用的宿主细胞可以是对于本领域技术人员而言公知的任意宿主细胞,包括原核细胞、真核细胞、例如细菌细胞、菌类细胞、酵母细胞、哺乳动物细胞、昆虫细胞或植物细胞。示例性细菌细胞中,包括埃希氏菌(escherichia)、沙门菌(salmonella)、链霉菌(streptomyces)、假单胞菌(pseudomonas)、金黄色葡萄球菌(staphylococcus)、或芽胞杆菌(bacillus)的任意种,上述中,例如包括大肠杆菌(escherichiacoli)、乳酸乳球菌(lactococcuslactis)、枯草菌(bacillussubtilis)、蜡样芽孢杆菌(bacilluscereus)、鼠伤寒沙门菌(salmonellatyphimurium)、荧光假单胞菌(pseudomonasfluorescens)等。
作为本发明中使用的宿主细胞,从对各种应激具有抗性且也容易进行基因重组的角度出发,优选大肠杆菌细胞。
本发明中“多核苷酸”这一术语是指单一核酸和多个核酸这两者,包括mrna等核酸分子、质粒rna、全长的cdna及其片段等。多核苷酸由任意的多聚核糖核苷酸或多聚脱氧核糖核苷酸构成,可以是修饰、非修饰的任意者。可以是单链或双链,也可以是两者的混合。
本发明中“表达异种的芳香族醛合成酶(aas)的野生型或突变体的、用于产生苄基异喹啉生物碱(bia)的重组宿主细胞。”时的“异种”是指:表达与本发明的重组宿主细胞不同种属来源的蛋白质、编码其的多核苷酸的细胞。例如,本发明的重组宿主细胞为大肠杆菌细胞时,作为异种蛋白、异种多核苷酸,可列举出昆虫、植物等的蛋白、多核苷酸。在本发明的重组宿主细胞中导入编码异种蛋白的多核苷酸的目的在于:由异种中,导入编码该宿主细胞原本所不具有的酶等蛋白质的多核苷酸,从而发挥目标代谢途径、即由l-多巴产生thp和/或网脉番荔枝碱的代谢途径的作用。
(多核苷酸的导入方法)
为了使宿主细胞表达异种的芳香族醛合成酶(aas)的野生型或突变体,必须使宿主细胞表达编码异种的芳香族醛合成酶(aas)的野生型或突变体的多核苷酸,例如,用包含该多核苷酸的表达载体转化细胞即可。表达编码用于由thp合成网脉番荔枝碱所需的酶的多核苷酸的情况也是同样的。表达载体只要包含处于能表达状态的本发明的基因就没有特别限定,可以使用适于每种宿主的载体。
本发明的表达载体可以通过如下方式制作:在上述异种多核苷酸的上游插入转录启动子、根据情况在下游插入终止子而构建表达盒,将该表达盒插入表达载体中,从而制作。或者,在表达载体上已经具有转录启动子和/或终止子的情况下,则无需构建表达盒,直接利用载体中的启动子和/或终止子、在之间插入该异种多核苷酸即可。
为了在载体上插入上述异种多核苷酸,可以利用使用限制性酶的方法、使用拓扑异构酶的方法等。另外,如果需要还可以在插入时添加适当的连接体。另外,作为对于翻译成氨基酸而言重要的碱基序列,已知sd序列、kozak序列等核糖体结合序列,也可以将这些序列插入基因的上游。伴随着插入也可以置换基因所编码的氨基酸序列的一部分。
本发明中使用的载体只要保持本发明的基因就没有特别限定,可以使用适于每种宿主的载体。作为载体,例如可列举出质粒dna、噬菌体dna、逆转录转座子dna、人工染色体dna等。
将表达载体导入宿主中的方法只要是适于宿主的方法就没有特别限定。作为能利用的方法,例如可列举出电穿孔法、使用钙离子的方法、原生质球法、乙酸锂法、磷酸钙法、脂质体转染法等。重组宿主细胞中的该多核苷酸的表达可以根据本领域技术人员公知的方法来定量化。例如,可以通过该多核苷酸所编码的多肽在细胞蛋白质整体中的百分比来表示。另外,可以通过使用转化后细胞的细胞提取液、并使用了能检测该多核苷酸所编码的多肽的抗体的蛋白质印迹法加以确认,或者可以通过使用了特异性检测该多核苷酸的引物的实时pcr等加以确认。
<本发明的苄基异喹啉生物碱(bia)的制造方法>
本发明也提供使用了上述的本发明的重组宿主细胞的四氢罂粟林(thp)、去甲乌药碱、3-羟基乌药碱、3-羟基-n-甲基乌药碱、及/或网脉番荔枝碱的制造方法。作为本发明的制造方法,大致分为2种方法。
1种方法是包括下述工序的方法,所述工序为:将上述的本发明的重组宿主细胞在含有l-多巴和/或酪氨酸的培养基中培养。对于已摄入了培养基中的l-多巴和/或酪氨酸的本发明的重组宿主细胞,使用在细胞内表达的aas等而能够高效地生成thp、去甲乌药碱、3-羟基乌药碱、3-羟基-n-甲基乌药碱、及/或网脉番荔枝碱。所生成的thp、去甲乌药碱、3-羟基乌药碱、3-羟基-n-甲基乌药碱、及/或网脉番荔枝碱被分泌到培养基中。
另1种方法是包括下述工序的方法,所述工序为:在无细胞体系中使芳香族醛合成酶(aas)、芳香族氨基酸脱羧酶(aaad)的野生型或突变体作用于l-多巴和/或酪氨酸。该方法中,例如,在体外使芳香族醛合成酶(aas)、芳香族氨基酸脱羧酶(aaad)的野生型或突变体直接作用于l-多巴和/或酪氨酸而生成多巴胺、及dhpaa或4-hpaa等苯基醛,多巴胺与dhpaa或4-hpaa彼此结合,由此生成thp或去甲乌药碱。进而,通过与由thp、去甲乌药碱合成网脉番荔枝碱所需的酶反应而生成网脉番荔枝碱。此时,作为芳香族醛合成酶(aas)、芳香族氨基酸脱羧酶(aaad)的野生型或突变体,优选使用由上述的本发明的重组宿主细胞得到的酶。
实施例
通过以下的实施例对本发明进行具体地说明,但本发明不受实施例限定。需要说明的是,在用于说明实施例的一部分附图中,一部分氨基酸符号为单字母符号。
1.用于生物合成网脉番荔枝碱的通过dhpaas的对称性thp产生途径的选择
m-途径酶检索依据araki等人的方法(araki,etal.m-path:acompassfornavigatingpotentialmetabolicpathways.bioinformatics31,905-911(2015).)使用了基于网络的版本。m-途径得分以tanimoto系数的形式计算。作为m-途径数据库,使用对来自kegg的最新的底物、制品及酶信息进行了更新的2016版本。为了调查介导酪氨酸(pubchemcid:6057)转化为4-hpaa(cid:440113)、介导l-多巴(cid:6047)转化为dhpaa(cid:119219)、介导酪氨酸转化为2’-去甲小檗宁(norberbamunine)(cid:441063)、介导组氨酸(cid:6274)转化为咪唑-4-乙醛(cid:150841)、介导4-氨基苯基丙氨酸(cid:151001)转化为4-氨基苯基乙醛(cid:20440853)的酶,而使用了策划(curation)模式。另外,在由酪氨酸转化为高香草酸(cid:1738)时以原始模式使用了m-途径。
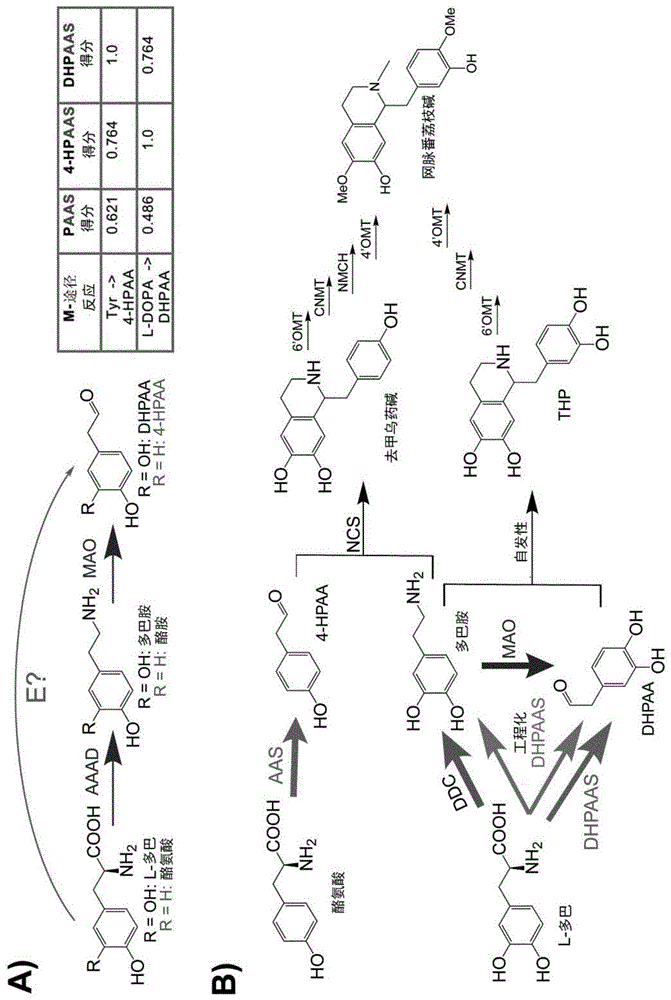
m-途径酶检索与已知的对酶网络进行检索的工具不同,其优点在于能够基于底物和产物的相似性来预测未知的酶反应。为了探索由l-多巴生产bia的优化,依据araki等人的方法测试了m-途径酶检索算法。在结合了来自brenda(https://www.brenda-enzymes.org/)和kyotoencyclopediagenesandgenomes(kegg、http://www.kegg.jp)的最新酶数据库的数据库中使用m-途径时,作为用于由l-酪氨酸(tyr)生产4-羟基苯基乙醛(4-hpaa或4-hpa)、由3,4-二羟基苯丙氨酸(l-多巴)生产3,4-二羟基苯基乙醛(dhpaa、dhpa或dopal)的推测的捷径,已鉴定出昆虫来源的3,4-二羟基苯基乙醛合成酶(dhpaas)和植物来源的芳香族醛合成酶(aas;paas、4-hpaas)(图1的a)。另外,在组合上述dhpaas或芳香族醛合成酶(aas;paas、4-hpaas)与3,4-二羟基苯基丙氨酸脱羧酶(ddc)时,发现了与以往报道的mao介导途径不同的新的对称性thp和去甲乌药碱生成途径(图1的b)。
上述芳香族醛合成酶(aas;paas、4-hpaas)和dhpaas是催化芳香族氨基酸的脱羧基化和氨基氧化的双功能酶。包括苯基乙醛合成酶(paas、keggec4.1.1.109)和4-羟基苯基乙醛合成酶(4-hpaas、keggec4.1.1.108)在内在植物中发现的这些酶被统称为aas。近些年,在昆虫中发现的酶dhpaas(ec4.1.1.107)由于催化l-多巴的氧化脱羧基化而被认为是aas相关蛋白。需要说明的是,“aas”在广义上是芳香族醛合成酶,是包括昆虫来源的3,4-二羟基苯基乙醛合成酶(dhpaas)和植物来源的芳香族醛合成酶(aas;paas、4-hpaas)中任意者的概念,但在狭义上从酶发现的经过出发是指植物来源的芳香族醛合成酶。系统发生分析表明:上述的植物来源的aas和昆虫来源的dhpaas从芳香族氨基酸脱羧酶(aaad、ec4.1.1.28)起开始分支。因此,上述aas、dhpaas和aaad具有结构相似性、且依赖于吡哆醛5’-磷酸(plp)作为辅因子。上述aas和dhpaas根据kegg而归属于ec4.1.1.-,但由于双功能作用而不易分类,关于这些相对较新表征的酶仍存在不清楚的地方。
由上述aas、dhpaas所介导的对称性bia产生途径与mao介导非对称性途径(图1)相比也具有优点。作为这样的优点,包括可溶性的dhpaas对l-多巴的特异性高于mao这点。使用数学模型和数值模拟来比较通过非对称性(ddc-mao)和对称性(ddc-dhpaas)途径的thp生成。非对称途径中,mao识别各种胺,因此将来自其它底物的竞争性抑制导入mao反应速率vmao8中。对称途径中,对于ddc(vddc)和dhpaas(vdhpaas)的反应速率,构建了没有反馈的模型及考虑了反馈抑制的2个模型。模型内的可能的参数值范围参照placzek,s.etal.brendain2017:newperspectivesandnewtoolsinbrenda.nucleicacidsres.45,d380~d388(2017)。为了预测各途径的性能,在相关范围内随机生成参数值,实施了蒙特卡洛模拟。迭代次数为10000次,模拟时间为0~50小时。将l-多巴作为基于随机生成的参数的常数项而提供。在达到100mml-多巴的最大量时,停止向系统中供给底物。使用scipy.integrate.odeint作为数值模拟求解器在python3.0中执行人工编程而得的程序。
之后的体外和体内试验的结果启示出:反应性高的dhpaa通过与细胞内或增殖培养基中存在的竞争性亲核试剂的反应而被降解消耗。在动态模型中包括该dhpaa的损失时,可得到稍低的thp收率(图2),与实验收率良好地吻合。然而,包括增殖培养基的缓冲液组成、ph、温度、潜在的抑制剂和代谢通量在内的许多变量也应被考虑成用于改善thp收率的训练数据。若同时考虑到由产物所致的反馈抑制和dhpaa的损失,对称性ddc-dhpaas途径示出明显高于mao介导非对照的途径的thp预测收率(图2)。这些模型启示出:与以前报道的mao介导thp产生量(最高1mm)相比,dhpaas所介导的途径可能产生更高水平的thp。进而,基于反馈抑制模型可知:多巴胺与dhpaa的平衡对于最佳的thp生成是重要的。因此,进一步研究了基于dhpaas的dhpaa产生与多巴胺产生的平衡调节。
2.基于结构的新的dhaas突变体的鉴定和工程化
为了选择对生产bia最佳的序列,比较了推测的植物来源aas和昆虫来源dhpaas的结构。通过在嵌合体内操作的建模器建立与共价键合有plp辅因子的芳香族氨基酸底物形成了复合物的推测的aas、dhpaas的二聚体同源模型,通过moe改进了结构(图3)。
m-途径将4-hpaas鉴定为生成4-hpaa的酶,所述4-hpaa为植物bia合成的重要的中间体。因此假定p.somniferum(罂粟)在天然的4-hpaa生物合成中利用了aas活性,并且由p.somniferum的序列检索潜在的aas酶。有趣的是,基于与卡比多巴(pdbid:1js3)形成了复合物的susscrofaddc的结构而重建的p.somniferum(罂粟)酪氨酸脱羧酶(tydc1)在aaad活性位点his192所对应的位置包含新的异亮氨酸残基(图3,中间的图),该位置作为重要的催化残基而受到注目。然而,除了新的tydc1leu205外,所有的p.somniferumtydc1序列与标准的aaad序列非常相似。与此相对,与推测的昆虫dhpaas序列相比,观察到更明确的活性位点的差异(图3)。因此,将焦点转移到昆虫dhpaas上,以便选择最佳的bia生产系统。
关于包括昆虫dhpaas的进化和所有的必要的催化残基的阐明在内的其氧化脱羧基化机理,仍然存留很多疑问。为了阐明这些疑问并得到基于dhpaas的机制的见解,与结构解析相结合进行了系统分类。
通过建模器和嵌合体制作b.mori(家蚕)的dhpaas和p.somniferum(罂粟)的tydc1的二聚体同源性模型。使用黑腹果蝇(d.melanogaster)的ddc(pdbid:3k40)和组氨酸脱羧酶(4e1o)的晶体结构作为家蚕(b.mori)的dhpaas重建的模板。使用与卡比多巴(pdbid:1js3)形成了复合物的susscrofa的ddc的结构作为tydc1的模板。通过分子操作环境(molecularoperatingenvironment:moe)进行了plp与芳香族氨基酸底物的共价键合和结构的精密化。通过pymol分析了完成的结构。
通过检索昆虫序列np_476592.1、np_724162.1、xp_319838.3、eds39158.1、eat37246.1和eat37247.1,从而由蛋白质blast非重复数据库收集了昆虫的aaad和aas序列。去除重复的序列和超过700个氨基酸长的序列,对得到的序列进行排列,使用分裂值0.12制作了系统树。通过用moe制作序列同一性表,从而鉴定了簇。通过738个昆虫aaad相关序列的系统发生解析,鉴定出247个推测的dhpaas序列和5个dhpaas组(图4)。
为了获得针对dhpaas机制的新见解而选择了构成中央系统发生群(图4)的性状不明确的鳞翅目dhpaas。若对昆虫dhpaas的结构进行分析,则由gly353~arg324形成的新的环,即使使用黑腹果蝇3,4-二羟基苯基丙氨酸脱羧酶(ddc、pdbid:3k40)的结构作为模板也未能容易地模型化。通过使用与组氨酸甲基酯(pdbid:4e1o)的复合物中的人组氨酸脱羧酶的模板,而使与交叉二聚体活性位点形成和底物结合相关的该320~350环更良好地模型化。
由ddc和dhpaas活性位点的比较明确了:第192位(家蚕(b.mori)和黑腹果蝇(d.melanogaster)dhpaas的编号)是确定脱羧酶或醛合成酶的催化活性时的重要的残基(图3和4)。该192残基可以与通过aas机制而被氧化的plp-芳香族氨基酸复合物的外部醛亚胺形成氢键。之前已经报道了含有asn192的埃及伊蚊和黑腹果蝇dhpaas的特性,但在这次的研究中,通过结构性解析和功能性解析,也将asn192鉴定/确认为重要的催化位点。
通过对ddc和dhpaas的结构进行仔细比较,示出dhpaas的phe79和tyr80在区分dhpaas活性和ddc活性方面发挥进一步的作用(图3和4)。tyr79-phe80在昆虫ddc中保守,但该79-80基序在昆虫dhpaas中通常与phe79-tyr80相反,这些残基也围绕plp-底物复合物的外部醛亚胺(图3)。因此,我们假设这些残基与dhpaas的催化机制有关且在dhpaas的分类中是有用的。在已鉴定出的5个dhpaas组中,phe79-tyr80在apis(蜜蜂)和蚊中保守。果蝇的dhpaas序列中,被称为亚型x1者中phe79-tyr80保守,被称为亚型x2者(包括np476592.126)中tyr79-tyr80保守。鳞翅目和蚁类的dhpaas组中,混合有phe79-tyr80、tyr79-tyr80和tyr79-phe80(图4)。
以下的实验中,选择家蚕(b.mori)序列xm_004930959.2作为包含三个dhpaas特异性残基phe79、tyr80和asn192的全部、且也包含报道了针对l-多巴的底物特异性增加的gly353的、典型的dhpaas序列。进而,phe79tyr、tyr80phe和asn192hisdhpaas催化位点的氨基酸突变体是为了调查多巴胺和dhpaa的产生调节机制而设计的(图5的a)。
3.重组家蚕(b.mori)dhpaas的制备
通过geneart(invitrogen)合成全长的野生型家蚕(b.mori)dhpaas的cdna序列(xm_004930959.2;序列号1),通过bsai限制性酶位点克隆至具有卡那霉素抗药性(lifesensorsinc.)的pe-sumo载体。使用重叠pcr生成了氨基酸突变体的cdna(序列号2~4)。将dhpaas表达载体导入在添加了50μg/ml的卡那霉素的lb培养基中维持的bl21(de3)中、或导入在添加了50μg/ml的卡那霉素和34μg/ml的氯霉素的lb培养基中维持的bl21(de3)plyss中,从而进行转化。通过将0.2~0.45mm的iptg添加至在lb培养基中在需氧条件下增殖的大肠杆菌中,从而诱导了重组dhpaas的表达。诱导后,将培养温度降低至14~16℃。孵育一夜后,通过将细胞离心分离而颗粒化,再次悬浮于磷酸缓冲生理盐水(pbs)中,边在冰上冷却边通过超声波处理使其溶解。将裂解物离心分离,并将澄清的裂解物进样至hitraptalon和histraphp柱(gelifesciences)上,用pbs和10~20mm咪唑对其进行清洗。用450~1000mm咪唑洗脱重组dhpaas。然后,使用milliporeamiconultra-15离心过滤器,将缓冲液替换为补充了plp的pbs。
4.dhpaas底物和反应产物的分析
对于l-多巴与dhpaas的反应的进展,利用薄层色谱法(tlc)非定量地分析了底物和产物。在涂覆有硅胶60f254(merckmillipore)的铝板上进行tlc。使用1-丁醇:乙酸:h2o=7:2:1的比例的混合物作为流动相。在uv下分析了dhpaas反应的成分,接着进行加热并进行了茚三酮染色。
通过用shimadzulcms-8050esi三重四极杆方式得到的质谱鉴定了dhpaas反应的底物和产物。对于定量分析,使用nexerax2uhplc系统以及以多反应监测(mrm)模式操作的shimadzulcms-8050来进行。l-多巴(tci)、多巴胺(tci)、dhpaa(santacruzbiotechnology)和thp(sigma)分别使用198.10>152.10(+)、154.10>91.05(+)、151.30>123.15(-)和288.05>164.15(+)的qualifiermrm转换。对于多巴胺、dhpaa和thp,分别使用了154.10>137.05(+)、151.30>122.10(-)和288.05>123.15(+)的qualifiermrm转换。网脉番荔枝碱使用了330.10>177.20(+)的qualifiermrm转换。使用加热至40℃的discoveryhsf5-3柱(3μm、2.1mm×150mm、sigma-aldrich),并使用0.1%甲酸水溶液和0.1%甲酸乙腈的浓度梯度作为流动相,以0.25ml/分钟进行了分离。使用相同的lc-ms系统,用加热的asteccyclobondi2000柱(5μm、2.1mm×150mm、sigma-aldrich)以90%乙腈-50mmnh4oac(ph4.5)的流动相梯度、洗脱速度0.3ml/分钟进行洗脱,进行了(r,s)-thp的手性分析。
5.通过氨基酸置换进行的家蚕(b.mori)dhpaas的功能转换
如阴离子m/z151.10的检出和主要的多巴胺离子的缺失所示,重组家蚕(b.mori)xm_004930959.2野生型蛋白质产生dhpaa作为与l-多巴的主要产物(图5)。该家蚕(b.mori)dhpaas的鉴定启示出与dhpaas系统群相关的上述的分析是正确的。由结构解析的结果得出如下假设:phe79tyr-tyr80phe-asn192his三重突变体具有ddc样活性,但asn192his和phe79tyr-ty80phe突变体具有dhpaas和ddc活性这两者。为了验证该假设并获得针对dhpaas作用的全面见解,评价了家蚕(b.mori)dhpaas的野生型、asn192his突变体、phe79tyr-ty80phe突变体以及phe79tyr-tyr80phe-asn192hisdhpaas突变体的酶活性(图5、图6)。
基于tlc后的茚三酮染色,确认了phe79tyr-tyr80phe-asn192hisdhpaas突变体的主要产物为多巴胺,支持了上述假设(图5的b)。若对通过更长时间孵育而得到的产物进行分析,则检测出thp为l-多巴和phe79tyr-tyr80phe-asn192hisdhpaas反应产物的主要阳离子(图5的d)。
接着,通过h2o2的产生而评价了dhpaas的活性。使用过氧化氢荧光定量分析试剂盒(sigma)在96孔板中对h2o2进行了定量。将0.6-0.8μg的dhpaas溶解于pbs(20μl)中,与各种浓度的l-多巴(10μl)混合,接着添加了30μl的过氧化物酶混合物(sigma)。使用spectramaxparadigm酶标仪(moleculardevices)检测了荧光。其结果,可知:asn192对于维持dhpaas的活性是最重要的,phe79和tyr80均对dhpaas的活性产生影响(图6)。
6.在体外通过dhpaas产生thp
由于确认了通过phe79tyr-tyr80phe-asn192hisdhpaas可以直接产生thp,因此使用野生型及设计出的3种家蚕(b.mori)dhpaas突变体,评价了体外的thp产生(图7)。
具体的试验方法如下所述。将溶解于pbs的dhpaas(2~3μg)与l-多巴水溶液混合而使最终体积为40μl。在其中加入最终浓度1.875mm的l-多巴、及2.5mm的抗坏血酸钠。在室温(23~24℃)开始进行反应,8小时后将温度设为4℃。在各个时间点收集反应液2μl,用包含抗坏血酸和樟脑磺酸的meoh98μl进行稀释。将该稀释反应液立即保存在-30℃,保存至lc-ms分析为止。
使用在mrm模式下操作的lc-ms对多巴胺、dhpaa和thp的产生进行了监测。如被氧化的thp离子m/z284.10和m/z306.15的检出所示,thp的收率对氧化是极其敏感的。thp-醌([thp-3h]+=284.0917)主要对应于离子m/z284.10。鉴定出的阳离子m/z306.15可以对应于thp的n-氧化物([thp+oh]+=306.1336)。
为了抑制由h2o2所致的产物的氧化性分解而添加抗坏血酸时,可显著改善体外的thp的收率。若添加2.5mm的抗坏血酸钠,通过phe79tyr-tyr80phe-asn192hisdhpaas突变体由l-多巴转化为thp的转化率增加至23.9%(219μm)。该值超过了多巴胺在体内转化为thp的最高转化率15.9%(nakagawa,a.etal.sci.rep.4,6695(2014)))。抗坏血酸未抑制由dhpaas产生dhpaa的事实说明:dhpaa并不是由h2o2氧化而产生的多巴胺的二次产物,而是l-多巴的直接的酶反应的产物。
正如预测的那样,dhpaa产生量在使用了野生型酶和phe79tyr-tyr80phe突变体的情况下最高,在使用了asn192his突变体和phe79tyr-tyr80phe-asn192his突变体的情况下最低(图7、图8)。多巴胺产生如预期的那样可观察到相反的倾向,在phe79tyr-tyr80phe-asn192his突变体中最高、在野生型dhpaas中最低,但基于asn192his突变体的多巴胺产生高于phe79tyr-tyr80phe突变体。这些体外试验的结果支持了上述假设、即phe79、tyr80和asn192在dhpaas的功能转换中的效果是由立体结构所引起的(图8)。
7.在体内通过dhpaas产生thp
为了克隆至作为表达载体的ptrchis2b上,使用包含ncoi和xhoi限制性酶位点的引物对dhpaas序列进行了pcr扩增。将得到的无标签的表达载体导入bl21(de3)plyss中,由此进行转化。为了进行生物生产,使用包含15.6mm抗坏血酸钠、100μg/ml氨苄青霉素和34μg/ml氯霉素的m9培养基3.5ml,边以200rpm振荡边使大肠杆菌在37℃下增殖。在od600达到0.2~0.4时,以最终浓度0.97mm添加iptg来诱导dhpaas的表达,将培养温度降低至25℃。自诱导起1小时13分钟后,在各培养液中添加3.4mg的l-多巴(0.97mg/ml),接着以4.86μm的最终浓度添加plp。在添加l-多巴的12.9小时后,将培养温度降低至16℃。在4个时间点收集培养样品(300~500μl),通过具有3000da的截留分子量的milliporeamiconultra0.5ml离心过滤器进行过滤。在添加底物22.7小时后,在各培养中添加约4~5mg的抗坏血酸,将培养物移至4℃中。在添加底物49.8小时后,以4500g将培养物离心分离,为了进行最终测定而回收了上清液。用meoh稀释培养上清液,对l-多巴、多巴胺、dhpaa和thp进行了定量。
在使用了在lb培养基中增殖的大肠杆菌(e.coli)的初始尝试中,thp的产生量通常极低,但phe79tyr-tyr80phe突变体稍高,其次是野生型dhpaas。然而,若将培养基更换为m9基础培养基,则thp产生量大幅增加(图9)。
多巴胺和dhpaa在体内的生物生产与基于dhpaas结构的假设完全一致,通过phe79、tyr80和asn192的置换而产生。与体外的结果相比,phe79tyr-tyr80phe突变体的thp产生量为0.902μm,在体内显示出最强的thp生产。野生型dhpaas显示出第二高的产生量,其次是phe79tyr-tyr80phe-asn192hisdhpaas、asn192his突变体的顺序。如手性lc-ms分析(图9-2)所示,在体内通过dhpaas而生成了(r,s)-thp的非对映体混合物。
8.体内的网脉番荔枝碱的产生
在表达dhpaas的同时在大肠杆菌中表达进行由thp转化为网脉番荔枝碱的3种酶,并确认了在体内产生网脉番荔枝碱。具体而言,使用表达山茶(c.japonica)来源的去甲乌药碱6-o-甲基转移酶(6’omt)、3’-羟基-n-甲基-(s)-乌药碱-4’-o-甲基转移酶(4’omt)、以及乌药碱-n-甲基转移酶(cnmt)基因的pacyc184载体(序列号13)、及实施例7中得到的dhpaas表达载体ptrchis2b(序列号14),将bl21(de3)plyss共转化,并用氨苄青霉素和氯霉素对得到的产网脉番荔枝碱大肠杆菌进行选择。在补充了2%葡萄糖的m9基础培养基中测试了网脉番荔枝碱产生。使大肠杆菌增殖直至od600达到0.2-0.3为止,在其中添加0.5mm的iptg、450μm的l-多巴和4.54mm的抗坏血酸钠。进而在添加底物17.2小时后,追加444μm的抗坏血酸盐。在25℃、200rpm振荡下培养大肠杆菌,产生了网脉番荔枝碱。为了进行多巴胺、dhpaa、thp和网脉番荔枝碱的定量,用包含樟脑磺酸和抗坏血酸的meoh将培养液稀释。对于phe79tyr-tyr80phe和phe79tyr-tyr80phe-asn192his介导性网脉番荔枝碱产生,进行重复测定,对于野生型和asn192his介导性网脉番荔枝碱产生,进行4次测定。将结果示于图10。
9.体内的thp、网脉番荔枝碱和中间体的产生
使用导入了野生型的dhpaas的表达载体ptrchis2b、导入了phe79tyr-tyr80phe-asn192his突变dhpaa的表达载体pe-sumo、导入了山茶(c.japonica)来源的去甲乌药碱6-o-甲基转移酶(6’omt)、3’-羟基-n-甲基-(s)-乌药碱-4’-o-甲基转移酶(4’omt)、以及乌药碱-n-甲基转移酶(cnmt)基因的pacyc184载体,将bl21(de3)plyss共转化。在thp生产的最初步骤中,在添加了1.5%葡萄糖、100μg/ml氨苄青霉素、及50μg/ml卡那霉素的不含甘油的tb中以37℃培养该3个质粒系统。在od600达到0.38后,添加iptg使最终浓度成为0.5mm。诱导后1.5小时,将温度降低至25℃。自诱导起5.5小时后,通过4000×g的离心分离回收细胞,在-80℃下保存一夜,将来自约43ml的培养物的颗粒再次悬浮于包含低钙、0.2%tritonx-100、1.5%葡萄糖、10μmplp、10mm抗坏血酸钠、1mml-多巴的m9中,使最终体积为6.5ml。混合后,将培养物在24~25℃下维持1.5小时,以5000×g进行离心分离,将上清液中的多巴胺和dhpaa浓缩。在添加底物25小时后,以5000×g再次进行离心分离,将含有thp的上清液用于以下的bia生产工序。
在bia生产的第2阶段中,将包含山茶(c.japonica)的4-omt和罂粟(p.somniferum)的6-omt和cnmt的pet23a导入bl21(de3)中。将该细胞在葡萄糖为1.5%、氨苄青霉素为100μg/ml且无甘油的tb中最初以37℃进行培养。在od600达到0.78后,添加iptg使最终浓度成为0.5mm。在诱导的1.5小时后,将温度降低至25℃。在诱导的5.5小时后,通过4000×g的离心分离回收细胞,在-80℃下保存两夜,将46ml的来自培养物的颗粒再次悬浮于最初步骤的上清液中。然后,25℃下边振荡边测定了bia生产量(3hc、3hnmc及网脉番荔枝碱)。
使用lc-ms和mrm分析了培养基的稀释样品。将结果示于图11。需要说明的是,在添加l-多巴的23小时后定量了thp,在添加第2种生物生产剂(最初步骤的上清液)的18.5小时后定量了3hc、3hnmc及网脉番荔枝碱。此处,图中的误差条表述平均的标准误差(进行了n=3的独立测定)。
如图11所示,确认了通过上述2个阶段的细胞生产系统产生了thp、网脉番荔枝碱和2种中间体。
10.体内的thp的产生(dhpaas的3个突变体和tfncs的导入)
使用phe79tyr-tyr80phe-asn192his突变dhpaas表达载体ptrchis2b-tdhpaas、ncs表达载体pcdfduet-1-tfncs,将bl21(de3)共转化。在添加了氨苄青霉素、奇霉素、1mm抗坏血酸的lb培养基中以37℃进行培养。在od600达到0.4~0.6后,添加iptg以使最终浓度成为0.5mm,3小时后,通过4000×g的离心分离回收细胞,将颗粒再次悬浮于包含135μmplp、5.1mm抗坏血酸钠、1.97mml-多巴、1.94mmα-甲基多巴的lb培养基中。混合后,将培养物在25℃下维持16.5小时,以5000×g进行离心分离,使用lc-ms和mrm对上清液中的多巴胺和dhpaa、thp进行了定量。将结果示于图12。
如图12所示,通过导入tfncs,与仅内源性的ncs的情况相比,回收了更多的thp。
11.体内的由酪氨酸产生去甲乌药碱(罂粟(p.somniferum)的tydc和ncs的导入)
(1)导入了tydc1和tfncs的试验
制作了导入了罂粟(p.somniferum)的tydc1的野生型或突变体(tydc1-y98f-f99y-l205n)、以及tfncs(进行了密码子优化的碱基序列如序列号27所示,所对应的氨基酸序列如序列号28所示)的载体、各种pcdfduet-1-tfncs-pstydc1。需要说明的是,对于上述的突变,通过使用序列号17和18的引物,从而合成了导入了tyr98phe、phe99tyr的突变的核苷酸,通过使用序列号19和序列号20的引物,从而合成了导入了leu205asn的突变的核苷酸。使用该载体将bl21(de3)转化。在添加了奇霉素的lb中以37℃、200rpm进行了振荡培养。在od600超过0.3后,添加iptg以使最终浓度成为0.5mm,在28℃下以180rpm进行振荡培养。1小时后,将2mm抗坏血酸钠、0.5mm多巴胺(da)、1mm酪氨酸以各自的最终浓度如以下所示那样添加至培养液中。混合后,进行51小时振荡培养,使用lc-ms和mrm对上清液中的去甲乌药碱进行定量。将结果示于图13。
(2)导入了tydc3和tfncs的试验
制作了导入了罂粟(p.somniferum)的tydc3的野生型或突变体(tydc3-y100f-f101y-h203n)、以及tfncs(密码子优化的碱基序列如序列号27所示,所对应的氨基酸序列如序列号28所示)的载体、各种pcdfduet-1-tfncs-pstydc3。需要说明的是,对于上述的突变,通过使用序列号23和24的引物,从而合成了导入了phe101tyr、tyr100phe的突变的核苷酸,另外,通过使用序列号25和序列号26的引物,从而合成了导入了his203asn的突变的核苷酸。使用该载体将bl21(de3)转化。在添加了奇霉素的lb中以37℃、200rpm进行振荡培养。在od600超过0.3后,添加iptg以使最终浓度成为0.5mm,在28℃下以180rpm进行振荡培养。1小时后,将2mm抗坏血酸钠、0.5mm多巴胺(da)、1mm酪氨酸以各自的最终浓度如以下所示那样添加至培养液中。混合后,振荡培养51小时,使用lc-ms和mrm对上清液中的去甲乌药碱进行定量。将结果示于图13。
(3)导入了tydc1和psoncs3的试验
制作了导入了罂粟(p.somniferum)的tydc1的野生型或突变体(tydc1-y98f-f99y-l205n)、以及psoncs3(密码子优化的碱基序列如序列号29所示,所对应的氨基酸序列如序列号30所示)的载体、各种pcdfduet-1-psoncs3-pstydc1。需要说明的是,对于上述的突变,通过使用序列号17和18的引物,从而合成了导入了phe99tyr、tyr98phe的突变的核苷酸,另外,通过使用序列号19和序列号20的引物,从而合成了导入了leu205asn的突变的核苷酸。使用该载体将bl21(de3)转化。在添加了奇霉素的lb中以37℃、200rpm进行振荡培养。在od600超过0.3后,添加iptg以使最终浓度成为0.5mm,在28℃下以180rpm进行振荡培养。1小时后,将2mm抗坏血酸钠、0.5mm多巴胺(da)、1mm酪氨酸以各自的最终浓度如以下所示那样添加至培养液中。混合后,振荡培养51小时,使用lc-ms和mrm对上清液中的去甲乌药碱进行定量。将结果示于图14。
(4)导入了tydc3和psoncs3的试验
制作了导入了罂粟(p.somniferum)的tydc3的野生型或突变体(tydc3-y100f-f101y-h203n)、以及psoncs3(密码子优化的碱基序列如序列号29所示,所对应的氨基酸序列如序列号30所示)的载体、各种pcdfduet-1-psoncs3-pstydc3。需要说明的是,对于上述的突变,通过使用序列号17和18的引物,从而合成了导入了phe101tyr、tyr100phe的突变的核苷酸,另外,通过使用序列号19和序列号20的引物,从而合成了导入了his203asn的突变的核苷酸。使用该载体将bl21(de3)转化。在添加了奇霉素的lb中以37℃、200rpm进行振荡培养。在od600超过0.3后,添加iptg以使最终浓度成为0.5mm,在28℃下以180rpm进行振荡培养。1小时后,将2mm抗坏血酸钠、0.5mm多巴胺(da)、1mm酪氨酸以各自的最终浓度如以下所示那样添加至培养液中。混合后,振荡培养51小时,使用lc-ms和mrm对上清液中的去甲乌药碱进行定量。将结果示于图14。
如图13和图14所示,通过将罂粟(p.somniferum)的tydc1、或tydc3、及ncs(黄唐松草(thalictrumflavum)的tfncs、或罂粟(p.somniferum)的psoncs3)导入细胞内,从而成功地由酪氨酸产生4-hpaa和去甲乌药碱。进而,通过将上述的突变导入tydc1或tydc3中,从而能够显著增加去甲乌药碱的产生量。各tydc1中的第98位、第99位、第205位的氨基酸对应于具有共通结构的dhpaas中第79位、第80位、第192位的氨基酸。本试验中,可认为:将tydc1的第98位的氨基酸由tyr变为phe、将第99位的氨基酸由phe变为tyr、将第205位的氨基酸由his变为asn的突变是将有助于tydc1的脱羧酶活性的这些残基修饰为具有aas活性的突变。对于tydc3也可以说是同样的。
12.体内的由酪氨酸产生网脉番荔枝碱(罂粟(p.somniferum)的tydc和ncs的导入)
与上述11(1)的记载同样地,制作了导入了罂粟(p.somniferum)的tydc1的野生型或突变体(tydc1-y98f-f99y-l205n)、以及tfncs的载体、各种pcdfduet-1-tfncs-pstydc1。进而,使用了上述9中记载的表达山茶(c.japonica)来源的去甲乌药碱6-o-甲基转移酶(6’omt)、3’-羟基-n-甲基-(s)-乌药碱-4’-o-甲基转移酶(4’omt)、乌药碱-n-甲基转移酶(cnmt)、n-甲基乌药碱3-羟化酶(nmch)基因的pacyc184载体。使用这些载体将bl21(de3)转化。在添加了奇霉素、氯霉素、5mm抗坏血酸的m9培养基中以37℃、180rpm进行振荡培养。在od600达到0.2~0.3时,添加iptg以使最终浓度成为0.8mm,以25℃、180rpm进行30分钟振荡培养,将2.5mm多巴胺(da)、5mm酪氨酸以各自的最终浓度如以下所示那样添加至培养液中,混合后,将培养物以180rpm振荡培养93小时。使用lc-ms和mrm对上清液中的l-多巴、4hpaa、去甲乌药碱、thp、网脉番荔枝碱进行定量。将体内的反应路线图示于图15,将上清液中的l-多巴、4hpaa、去甲乌药碱、thp、网脉番荔枝碱的产生量示于图16。
如图16所示,通过将罂粟(p.somniferum)的tydc1、ncs、进而将6’omt、4’omt、cnmt、nmch导入细胞内,从而成功地由酪氨酸最终产生网脉番荔枝碱。
13.体内的由l-多巴产生thp和网脉番荔枝碱(修饰的恶臭假单胞菌(p.putida)的ddc的导入)
通过导入了恶臭假单胞菌(p.putida)的ddc的突变体(ddc-y79f-f80y-h181n)、山茶(c.japonica)来源的去甲乌药碱6-o-甲基转移酶(6’omt)、3’-羟基-n-甲基-(s)-乌药碱-4’-o-甲基转移酶(4’omt)、及乌药碱-n-甲基转移酶(cnmt)的pacyc184而将bl21(de3)转化。在添加了奇霉素和氯霉素的lb培养基中以28℃、180rpm进行振荡培养。在od600超过0.3时,添加iptg以使最终浓度成为0.74mm~1.48mm。以20℃、180rpm进行30分钟培养,将最终浓度约1.9mm的l-多巴和最终浓度约4.7mm的抗坏血酸钠添加至培养液中,混合后培养40小时。使用lc-ms和mrm对上清液中的thp和网脉番荔枝碱进行定量。将体内的反应路线图示于图17,及将上清液中的thp、3hnmc和网脉番荔枝碱的产生量示于图18。
如图17和18所示,通过恶臭假单胞菌(p.putida)的ddc的突变体(ddc-y79f-f80y-h181n)而能够由l-多巴产生thp、3hnmc和网脉番荔枝碱。该结果表明ddc的突变体(ddc-y79f-f80y-h181n)能够由l-多巴诱导多巴胺和dhpaa这两者。即,通过将与上述试验中导入到dhpaa中的突变(phe79tyr-tyr80phe-asn192his)相反的突变导入恶臭假单胞菌(p.putida)的ddc中(tyr79phe-phe80tyr-his181asn),从而成功地使ddc产生了dhpaas活性。
产业上的可利用性
根据本发明,通过使用表达作为双功能酶的芳香族醛合成酶(aas)、芳香族氨基酸脱羧酶(aaad)的野生型或突变体的重组宿主细胞,从而能够高效地且容易地生产苄基异喹啉生物碱(bia)。
序列表
<110>国立大学法人神户大学(kobeuniversity)
<120>用于产生苄基异喹啉生物碱(bia)的重组宿主细胞和苄基异喹啉生物碱(bia)的新的制造方法
<130>kp17076/fp4476pct
<160>30
<170>patentinversion3.5
<210>1
<211>1503
<212>dna
<213>家蚕(bombyxmori)
<400>1
atggacgcgaaccagtttcgggaattcggcagagcagtcatcgatatgttggcgagctac60
gctgaaaacataagggattatgatgttctgccgtctgttgaacctggttacttgttaaga120
gctctgcctgaaagtgcacccgaacaaccggaggattggaaagacataatgaaagatttc180
aatcaatcaataatgccgggtgtaacacactggcaatctccgcagttccatgcattctat240
ccttctggctcctcattcgcgagtattataggaaatatgcttagtgacggcttggcggtt300
gtgggatttagttggatggctagccctgcgtgtacagaactggaggtggtcacgatgaat360
tggcttggtaagctattagatttacctgaagaatttctaaattgctcttcgggtcccgga420
ggcggagttattcaaggatcagcgagcgaagccaccttagttggattgcttgtagccaaa480
gataaaactgttcgccgtttcatgaataataatccagatctcgatgaaaacgagataaaa540
gcaaaacttgtagcttatacatccgaccaatgtaactcgtcagtagaaaaagccggctta600
cttggttctatgaaaatgaaactgttgaaagcggatgctgatggatgcctacgcggagaa660
acattgaaaagggcaatcgaagaagataagtcgcaaggacttataccctgctatgtcgtc720
gctaatctgggaacaactggaacttgtgcattcgatcctttacatgaattagggccaata780
tgtagtgaggaagatatatggcttcacgtagatgcagcctatgcgggagcagcatttttg840
tgtcctgaatacagacacctaatgaaaggtattgaacattctcaatcgttcgtaacgaat900
gcacataagtggctaccggttaatttcgattgctctgctatgtgggttaaaaatggttat960
gatataacgagagcattcgatgtacaaagaatttatttggatgatgtaaaaacgacaatc1020
aagatcccagattacagacactggcaaatgccactaggccgtcgctttagagccttaaaa1080
ttgtggacagttatgagaatttatggtgctgaaggtttgaaaacgcatatcagacagcaa1140
atagaattagcacagtattttgcaaaactggtacgtgcggacgaacgttttgtgattggg1200
cccgaaccgactatggcattggtctgttttagactgaaagacggtgacacaattacgcga1260
caattgttggaaaatataacgcaaaaaaagaaagtgtttatggtggccggaacgcacagg1320
gatagatacgtcattagattcgtgatctgctcccgattgactaagaaggaagacgtcgat1380
tacagctggagccaaataaagaaagaaaccgacctcatctattcagataaaatacacaat1440
aaagcacaaataccagctcttgaacaattcacttcaagagaactatgcgaaaaatctaag1500
taa1503
<210>2
<211>1506
<212>dna
<213>家蚕(bombyxmori)
<400>2
atggacgcgaaccagtttcgggaattcggcagagcagtcatcgatatgttggcgagctac60
gctgaaaacataagggattatgatgttctgccgtctgttgaacctggttacttgttaaga120
gctctgcctgaaagtgcacccgaacaaccggaggattggaaagacataatgaaagatttc180
aatcaatcaataatgccgggtgtaacacactggcaatctccgcagttccatgcattctat240
ccttctggctcctcattcgcgagtattataggaaatatgcttagtgacggcttggcggtt300
gtgggatttagttggatggctagccctgcgtgtacagaactggaggtggtcacgatgaat360
tggcttggtaagctattagatttacctgaagaatttctaaattgctcttcgggtcccgga420
ggcggagttattcaaggatcagcgagcgaagccaccttagttggattgcttgtagccaaa480
gataaaactgttcgccgtttcatgaataataatccagatctcgatgaaaacgagataaaa540
gcaaaacttgtagcttatacatccgaccaatgtcactcgtcagtagaaaaagccggctta600
cttggttctatgaaaatgaaactgttgaaagcggatgctgatggatgcctacgcggagaa660
acattgaaaagggcaatcgaagaagataagtcgcaaggacttataccctgctatgtcgtc720
gctaatctgggaacaactggaacttgtgcattcgatcctttacatgaattagggccaata780
tgtagtgaggaagatatatggcttcacgtagatgcagcctatgcgggagcagcatttttg840
tgtcctgaatacagacacctaatgaaaggtattgaacattctcaatcgttcgtaacgaat900
gcacataagtggctaccggttaatttcgattgctctgctatgtgggttaaaaatggttat960
gatataacgagagcattcgatgtacaaagaatttatttggatgatgtaaaaacgacaatc1020
aagatcccagattacagacactggcaaatgccactaggccgtcgctttagagccttaaaa1080
ttgtggacagttatgagaatttatggtgctgaaggtttgaaaacgcatatcagacagcaa1140
atagaattagcacagtattttgcaaaactggtacgtgcggacgaacgttttgtgattggg1200
cccgaaccgactatggcattggtctgttttagactgaaagacggtgacacaattacgcga1260
caattgttggaaaatataacgcaaaaaaagaaagtgtttatggtggccggaacgcacagg1320
gatagatacgtcattagattcgtgatctgctcccgattgactaagaaggaagacgtcgat1380
tacagctggagccaaataaagaaagaaaccgacctcatctattcagataaaatacacaat1440
aaagcacaaataccagctcttgaacaattcacttcaagagaactatgcgaaaaatctaag1500
taataa1506
<210>3
<211>1506
<212>dna
<213>家蚕(bombyxmori)
<400>3
atggacgcgaaccagtttcgggaattcggcagagcagtcatcgatatgttggcgagctac60
gctgaaaacataagggattatgatgttctgccgtctgttgaacctggttacttgttaaga120
gctctgcctgaaagtgcacccgaacaaccggaggattggaaagacataatgaaagatttc180
aatcaatcaataatgccgggtgtaacacactggcaatctccgcagttccatgcatacttt240
ccttctggctcctcattcgcgagtattataggaaatatgcttagtgacggcttggcggtt300
gtgggatttagttggatggctagccctgcgtgtacagaactggaggtggtcacgatgaat360
tggcttggtaagctattagatttacctgaagaatttctaaattgctcttcgggtcccgga420
ggcggagttattcaaggatcagcgagcgaagccaccttagttggattgcttgtagccaaa480
gataaaactgttcgccgtttcatgaataataatccagatctcgatgaaaacgagataaaa540
gcaaaacttgtagcttatacatccgaccaatgtaactcgtcagtagaaaaagccggctta600
cttggttctatgaaaatgaaactgttgaaagcggatgctgatggatgcctacgcggagaa660
acattgaaaagggcaatcgaagaagataagtcgcaaggacttataccctgctatgtcgtc720
gctaatctgggaacaactggaacttgtgcattcgatcctttacatgaattagggccaata780
tgtagtgaggaagatatatggcttcacgtagatgcagcctatgcgggagcagcatttttg840
tgtcctgaatacagacacctaatgaaaggtattgaacattctcaatcgttcgtaacgaat900
gcacataagtggctaccggttaatttcgattgctctgctatgtgggttaaaaatggttat960
gatataacgagagcattcgatgtacaaagaatttatttggatgatgtaaaaacgacaatc1020
aagatcccagattacagacactggcaaatgccactaggccgtcgctttagagccttaaaa1080
ttgtggacagttatgagaatttatggtgctgaaggtttgaaaacgcatatcagacagcaa1140
atagaattagcacagtattttgcaaaactggtacgtgcggacgaacgttttgtgattggg1200
cccgaaccgactatggcattggtctgttttagactgaaagacggtgacacaattacgcga1260
caattgttggaaaatataacgcaaaaaaagaaagtgtttatggtggccggaacgcacagg1320
gatagatacgtcattagattcgtgatctgctcccgattgactaagaaggaagacgtcgat1380
tacagctggagccaaataaagaaagaaaccgacctcatctattcagataaaatacacaat1440
aaagcacaaataccagctcttgaacaattcacttcaagagaactatgcgaaaaatctaag1500
taataa1506
<210>4
<211>1506
<212>dna
<213>家蚕(bombyxmori)
<400>4
atggacgcgaaccagtttcgggaattcggcagagcagtcatcgatatgttggcgagctac60
gctgaaaacataagggattatgatgttctgccgtctgttgaacctggttacttgttaaga120
gctctgcctgaaagtgcacccgaacaaccggaggattggaaagacataatgaaagatttc180
aatcaatcaataatgccgggtgtaacacactggcaatctccgcagttccatgcatacttt240
ccttctggctcctcattcgcgagtattataggaaatatgcttagtgacggcttggcggtt300
gtgggatttagttggatggctagccctgcgtgtacagaactggaggtggtcacgatgaat360
tggcttggtaagctattagatttacctgaagaatttctaaattgctcttcgggtcccgga420
ggcggagttattcaaggatcagcgagcgaagccaccttagttggattgcttgtagccaaa480
gataaaactgttcgccgtttcatgaataataatccagatctcgatgaaaacgagataaaa540
gcaaaacttgtagcttatacatccgaccaatgtcactcgtcagtagaaaaagccggctta600
cttggttctatgaaaatgaaactgttgaaagcggatgctgatggatgcctacgcggagaa660
acattgaaaagggcaatcgaagaagataagtcgcaaggacttataccctgctatgtcgtc720
gctaatctgggaacaactggaacttgtgcattcgatcctttacatgaattagggccaata780
tgtagtgaggaagatatatggcttcacgtagatgcagcctatgcgggagcagcatttttg840
tgtcctgaatacagacacctaatgaaaggtattgaacattctcaatcgttcgtaacgaat900
gcacataagtggctaccggttaatttcgattgctctgctatgtgggttaaaaatggttat960
gatataacgagagcattcgatgtacaaagaatttatttggatgatgtaaaaacgacaatc1020
aagatcccagattacagacactggcaaatgccactaggccgtcgctttagagccttaaaa1080
ttgtggacagttatgagaatttatggtgctgaaggtttgaaaacgcatatcagacagcaa1140
atagaattagcacagtattttgcaaaactggtacgtgcggacgaacgttttgtgattggg1200
cccgaaccgactatggcattggtctgttttagactgaaagacggtgacacaattacgcga1260
caattgttggaaaatataacgcaaaaaaagaaagtgtttatggtggccggaacgcacagg1320
gatagatacgtcattagattcgtgatctgctcccgattgactaagaaggaagacgtcgat1380
tacagctggagccaaataaagaaagaaaccgacctcatctattcagataaaatacacaat1440
aaagcacaaataccagctcttgaacaattcacttcaagagaactatgcgaaaaatctaag1500
taataa1506
<210>5
<211>500
<212>prt
<213>家蚕(bombyxmori)
<400>5
metaspalaasnglnpheargglupheglyargalavalileaspmet
151015
leualasertyralagluasnileargasptyraspvalleuproser
202530
valgluproglytyrleuleuargalaleuprogluseralaproglu
354045
glnprogluasptrplysaspilemetlysasppheasnglnserile
505560
metproglyvalthrhistrpglnserproglnphehisalaphetyr
65707580
proserglyserserphealaserileileglyasnmetleuserasp
859095
glyleualavalvalglyphesertrpmetalaserproalacysthr
100105110
gluleugluvalvalthrmetasntrpleuglylysleuleuaspleu
115120125
progluglupheleuasncysserserglyproglyglyglyvalile
130135140
glnglyseralaserglualathrleuvalglyleuleuvalalalys
145150155160
asplysthrvalargargphemetasnasnasnproaspleuaspglu
165170175
asngluilelysalalysleuvalalatyrthrseraspglncysasn
180185190
serservalglulysalaglyleuleuglysermetlysmetlysleu
195200205
leulysalaaspalaaspglycysleuargglygluthrleulysarg
210215220
alaileglugluasplysserglnglyleuileprocystyrvalval
225230235240
alaasnleuglythrthrglythrcysalapheaspproleuhisglu
245250255
leuglyproilecysserglugluaspiletrpleuhisvalaspala
260265270
alatyralaglyalaalapheleucysproglutyrarghisleumet
275280285
lysglyilegluhisserglnserphevalthrasnalahislystrp
290295300
leuprovalasnpheaspcysseralamettrpvallysasnglytyr
305310315320
aspilethrargalapheaspvalglnargiletyrleuaspaspval
325330335
lysthrthrilelysileproasptyrarghistrpglnmetproleu
340345350
glyargargpheargalaleulysleutrpthrvalmetargiletyr
355360365
glyalagluglyleulysthrhisileargglnglnilegluleuala
370375380
glntyrphealalysleuvalargalaaspgluargphevalilegly
385390395400
progluprothrmetalaleuvalcyspheargleulysaspglyasp
405410415
thrilethrargglnleuleugluasnilethrglnlyslyslysval
420425430
phemetvalalaglythrhisargaspargtyrvalileargpheval
435440445
ilecysserargleuthrlyslysgluaspvalasptyrsertrpser
450455460
glnilelyslysgluthraspleuiletyrserasplysilehisasn
465470475480
lysalaglnileproalaleugluglnphethrserarggluleucys
485490495
glulysserlys
500
<210>6
<211>500
<212>prt
<213>家蚕(bombyxmori)
<400>6
metaspalaasnglnpheargglupheglyargalavalileaspmet
151015
leualasertyralagluasnileargasptyraspvalleuproser
202530
valgluproglytyrleuleuargalaleuprogluseralaproglu
354045
glnprogluasptrplysaspilemetlysasppheasnglnserile
505560
metproglyvalthrhistrpglnserproglnphehisalaphetyr
65707580
proserglyserserphealaserileileglyasnmetleuserasp
859095
glyleualavalvalglyphesertrpmetalaserproalacysthr
100105110
gluleugluvalvalthrmetasntrpleuglylysleuleuaspleu
115120125
progluglupheleuasncysserserglyproglyglyglyvalile
130135140
glnglyseralaserglualathrleuvalglyleuleuvalalalys
145150155160
asplysthrvalargargphemetasnasnasnproaspleuaspglu
165170175
asngluilelysalalysleuvalalatyrthrseraspglncyshis
180185190
serservalglulysalaglyleuleuglysermetlysmetlysleu
195200205
leulysalaaspalaaspglycysleuargglygluthrleulysarg
210215220
alaileglugluasplysserglnglyleuileprocystyrvalval
225230235240
alaasnleuglythrthrglythrcysalapheaspproleuhisglu
245250255
leuglyproilecysserglugluaspiletrpleuhisvalaspala
260265270
alatyralaglyalaalapheleucysproglutyrarghisleumet
275280285
lysglyilegluhisserglnserphevalthrasnalahislystrp
290295300
leuprovalasnpheaspcysseralamettrpvallysasnglytyr
305310315320
aspilethrargalapheaspvalglnargiletyrleuaspaspval
325330335
lysthrthrilelysileproasptyrarghistrpglnmetproleu
340345350
glyargargpheargalaleulysleutrpthrvalmetargiletyr
355360365
glyalagluglyleulysthrhisileargglnglnilegluleuala
370375380
glntyrphealalysleuvalargalaaspgluargphevalilegly
385390395400
progluprothrmetalaleuvalcyspheargleulysaspglyasp
405410415
thrilethrargglnleuleugluasnilethrglnlyslyslysval
420425430
phemetvalalaglythrhisargaspargtyrvalileargpheval
435440445
ilecysserargleuthrlyslysgluaspvalasptyrsertrpser
450455460
glnilelyslysgluthraspleuiletyrserasplysilehisasn
465470475480
lysalaglnileproalaleugluglnphethrserarggluleucys
485490495
glulysserlys
500
<210>7
<211>500
<212>prt
<213>家蚕(bombyxmori)
<400>7
metaspalaasnglnpheargglupheglyargalavalileaspmet
151015
leualasertyralagluasnileargasptyraspvalleuproser
202530
valgluproglytyrleuleuargalaleuprogluseralaproglu
354045
glnprogluasptrplysaspilemetlysasppheasnglnserile
505560
metproglyvalthrhistrpglnserproglnphehisalatyrphe
65707580
proserglyserserphealaserileileglyasnmetleuserasp
859095
glyleualavalvalglyphesertrpmetalaserproalacysthr
100105110
gluleugluvalvalthrmetasntrpleuglylysleuleuaspleu
115120125
progluglupheleuasncysserserglyproglyglyglyvalile
130135140
glnglyseralaserglualathrleuvalglyleuleuvalalalys
145150155160
asplysthrvalargargphemetasnasnasnproaspleuaspglu
165170175
asngluilelysalalysleuvalalatyrthrseraspglncysasn
180185190
serservalglulysalaglyleuleuglysermetlysmetlysleu
195200205
leulysalaaspalaaspglycysleuargglygluthrleulysarg
210215220
alaileglugluasplysserglnglyleuileprocystyrvalval
225230235240
alaasnleuglythrthrglythrcysalapheaspproleuhisglu
245250255
leuglyproilecysserglugluaspiletrpleuhisvalaspala
260265270
alatyralaglyalaalapheleucysproglutyrarghisleumet
275280285
lysglyilegluhisserglnserphevalthrasnalahislystrp
290295300
leuprovalasnpheaspcysseralamettrpvallysasnglytyr
305310315320
aspilethrargalapheaspvalglnargiletyrleuaspaspval
325330335
lysthrthrilelysileproasptyrarghistrpglnmetproleu
340345350
glyargargpheargalaleulysleutrpthrvalmetargiletyr
355360365
glyalagluglyleulysthrhisileargglnglnilegluleuala
370375380
glntyrphealalysleuvalargalaaspgluargphevalilegly
385390395400
progluprothrmetalaleuvalcyspheargleulysaspglyasp
405410415
thrilethrargglnleuleugluasnilethrglnlyslyslysval
420425430
phemetvalalaglythrhisargaspargtyrvalileargpheval
435440445
ilecysserargleuthrlyslysgluaspvalasptyrsertrpser
450455460
glnilelyslysgluthraspleuiletyrserasplysilehisasn
465470475480
lysalaglnileproalaleugluglnphethrserarggluleucys
485490495
glulysserlys
500
<210>8
<211>500
<212>prt
<213>家蚕(bombyxmori)
<400>8
metaspalaasnglnpheargglupheglyargalavalileaspmet
151015
leualasertyralagluasnileargasptyraspvalleuproser
202530
valgluproglytyrleuleuargalaleuprogluseralaproglu
354045
glnprogluasptrplysaspilemetlysasppheasnglnserile
505560
metproglyvalthrhistrpglnserproglnphehisalatyrphe
65707580
proserglyserserphealaserileileglyasnmetleuserasp
859095
glyleualavalvalglyphesertrpmetalaserproalacysthr
100105110
gluleugluvalvalthrmetasntrpleuglylysleuleuaspleu
115120125
progluglupheleuasncysserserglyproglyglyglyvalile
130135140
glnglyseralaserglualathrleuvalglyleuleuvalalalys
145150155160
asplysthrvalargargphemetasnasnasnproaspleuaspglu
165170175
asngluilelysalalysleuvalalatyrthrseraspglncyshis
180185190
serservalglulysalaglyleuleuglysermetlysmetlysleu
195200205
leulysalaaspalaaspglycysleuargglygluthrleulysarg
210215220
alaileglugluasplysserglnglyleuileprocystyrvalval
225230235240
alaasnleuglythrthrglythrcysalapheaspproleuhisglu
245250255
leuglyproilecysserglugluaspiletrpleuhisvalaspala
260265270
alatyralaglyalaalapheleucysproglutyrarghisleumet
275280285
lysglyilegluhisserglnserphevalthrasnalahislystrp
290295300
leuprovalasnpheaspcysseralamettrpvallysasnglytyr
305310315320
aspilethrargalapheaspvalglnargiletyrleuaspaspval
325330335
lysthrthrilelysileproasptyrarghistrpglnmetproleu
340345350
glyargargpheargalaleulysleutrpthrvalmetargiletyr
355360365
glyalagluglyleulysthrhisileargglnglnilegluleuala
370375380
glntyrphealalysleuvalargalaaspgluargphevalilegly
385390395400
progluprothrmetalaleuvalcyspheargleulysaspglyasp
405410415
thrilethrargglnleuleugluasnilethrglnlyslyslysval
420425430
phemetvalalaglythrhisargaspargtyrvalileargpheval
435440445
ilecysserargleuthrlyslysgluaspvalasptyrsertrpser
450455460
glnilelyslysgluthraspleuiletyrserasplysilehisasn
465470475480
lysalaglnileproalaleugluglnphethrserarggluleucys
485490495
glulysserlys
500
<210>9
<211>608
<212>prt
<213>家蚕(bombyxmori)
<400>9
metglyhishishishishishisglyserleuglnaspsergluval
151015
asnglnglualalysprogluvallysprogluvallysprogluthr
202530
hisileasnleulysvalseraspglysersergluilephephelys
354045
ilelyslysthrthrproleuargargleumetglualaphealalys
505560
argglnglylysglumetaspserleuargpheleutyraspglyile
65707580
argileglnalaaspglnalaprogluaspleuaspmetgluaspasn
859095
aspileileglualahisarggluglnileglyglymetaspalaasn
100105110
glnpheargglupheglyargalavalileaspmetleualasertyr
115120125
alagluasnileargasptyraspvalleuproservalgluprogly
130135140
tyrleuleuargalaleuprogluseralaprogluglnprogluasp
145150155160
trplysaspilemetlysasppheasnglnserilemetproglyval
165170175
thrhistrpglnserproglnphehisalaphetyrproserglyser
180185190
serphealaserileileglyasnmetleuseraspglyleualaval
195200205
valglyphesertrpmetalaserproalacysthrgluleugluval
210215220
valthrmetasntrpleuglylysleuleuaspleuproglugluphe
225230235240
leuasncysserserglyproglyglyglyvalileglnglyserala
245250255
serglualathrleuvalglyleuleuvalalalysasplysthrval
260265270
argargphemetasnasnasnproaspleuaspgluasngluilelys
275280285
alalysleuvalalatyrthrseraspglncysasnserservalglu
290295300
lysalaglyleuleuglysermetlysmetlysleuleulysalaasp
305310315320
alaaspglycysleuargglygluthrleulysargalailegluglu
325330335
asplysserglnglyleuileprocystyrvalvalalaasnleugly
340345350
thrthrglythrcysalapheaspproleuhisgluleuglyproile
355360365
cysserglugluaspiletrpleuhisvalaspalaalatyralagly
370375380
alaalapheleucysproglutyrarghisleumetlysglyileglu
385390395400
hisserglnserphevalthrasnalahislystrpleuprovalasn
405410415
pheaspcysseralamettrpvallysasnglytyraspilethrarg
420425430
alapheaspvalglnargiletyrleuaspaspvallysthrthrile
435440445
lysileproasptyrarghistrpglnmetproleuglyargargphe
450455460
argalaleulysleutrpthrvalmetargiletyrglyalaglugly
465470475480
leulysthrhisileargglnglnilegluleualaglntyrpheala
485490495
lysleuvalargalaaspgluargphevalileglyprogluprothr
500505510
metalaleuvalcyspheargleulysaspglyaspthrilethrarg
515520525
glnleuleugluasnilethrglnlyslyslysvalphemetvalala
530535540
glythrhisargaspargtyrvalileargphevalilecysserarg
545550555560
leuthrlyslysgluaspvalasptyrsertrpserglnilelyslys
565570575
gluthraspleuiletyrserasplysilehisasnlysalaglnile
580585590
proalaleugluglnphethrserarggluleucysglulysserlys
595600605
<210>10
<211>608
<212>prt
<213>家蚕(bombyxmori)
<400>10
metglyhishishishishishisglyserleuglnaspsergluval
151015
asnglnglualalysprogluvallysprogluvallysprogluthr
202530
hisileasnleulysvalseraspglysersergluilephephelys
354045
ilelyslysthrthrproleuargargleumetglualaphealalys
505560
argglnglylysglumetaspserleuargpheleutyraspglyile
65707580
argileglnalaaspglnalaprogluaspleuaspmetgluaspasn
859095
aspileileglualahisarggluglnileglyglymetaspalaasn
100105110
glnpheargglupheglyargalavalileaspmetleualasertyr
115120125
alagluasnileargasptyraspvalleuproservalgluprogly
130135140
tyrleuleuargalaleuprogluseralaprogluglnprogluasp
145150155160
trplysaspilemetlysasppheasnglnserilemetproglyval
165170175
thrhistrpglnserproglnphehisalaphetyrproserglyser
180185190
serphealaserileileglyasnmetleuseraspglyleualaval
195200205
valglyphesertrpmetalaserproalacysthrgluleugluval
210215220
valthrmetasntrpleuglylysleuleuaspleuproglugluphe
225230235240
leuasncysserserglyproglyglyglyvalileglnglyserala
245250255
serglualathrleuvalglyleuleuvalalalysasplysthrval
260265270
argargphemetasnasnasnproaspleuaspgluasngluilelys
275280285
alalysleuvalalatyrthrseraspglncyshisserservalglu
290295300
lysalaglyleuleuglysermetlysmetlysleuleulysalaasp
305310315320
alaaspglycysleuargglygluthrleulysargalailegluglu
325330335
asplysserglnglyleuileprocystyrvalvalalaasnleugly
340345350
thrthrglythrcysalapheaspproleuhisgluleuglyproile
355360365
cysserglugluaspiletrpleuhisvalaspalaalatyralagly
370375380
alaalapheleucysproglutyrarghisleumetlysglyileglu
385390395400
hisserglnserphevalthrasnalahislystrpleuprovalasn
405410415
pheaspcysseralamettrpvallysasnglytyraspilethrarg
420425430
alapheaspvalglnargiletyrleuaspaspvallysthrthrile
435440445
lysileproasptyrarghistrpglnmetproleuglyargargphe
450455460
argalaleulysleutrpthrvalmetargiletyrglyalaglugly
465470475480
leulysthrhisileargglnglnilegluleualaglntyrpheala
485490495
lysleuvalargalaaspgluargphevalileglyprogluprothr
500505510
metalaleuvalcyspheargleulysaspglyaspthrilethrarg
515520525
glnleuleugluasnilethrglnlyslyslysvalphemetvalala
530535540
glythrhisargaspargtyrvalileargphevalilecysserarg
545550555560
leuthrlyslysgluaspvalasptyrsertrpserglnilelyslys
565570575
gluthraspleuiletyrserasplysilehisasnlysalaglnile
580585590
proalaleugluglnphethrserarggluleucysglulysserlys
595600605
<210>11
<211>608
<212>prt
<213>家蚕(bombyxmori)
<400>11
metglyhishishishishishisglyserleuglnaspsergluval
151015
asnglnglualalysprogluvallysprogluvallysprogluthr
202530
hisileasnleulysvalseraspglysersergluilephephelys
354045
ilelyslysthrthrproleuargargleumetglualaphealalys
505560
argglnglylysglumetaspserleuargpheleutyraspglyile
65707580
argileglnalaaspglnalaprogluaspleuaspmetgluaspasn
859095
aspileileglualahisarggluglnileglyglymetaspalaasn
100105110
glnpheargglupheglyargalavalileaspmetleualasertyr
115120125
alagluasnileargasptyraspvalleuproservalgluprogly
130135140
tyrleuleuargalaleuprogluseralaprogluglnprogluasp
145150155160
trplysaspilemetlysasppheasnglnserilemetproglyval
165170175
thrhistrpglnserproglnphehisalatyrpheproserglyser
180185190
serphealaserileileglyasnmetleuseraspglyleualaval
195200205
valglyphesertrpmetalaserproalacysthrgluleugluval
210215220
valthrmetasntrpleuglylysleuleuaspleuproglugluphe
225230235240
leuasncysserserglyproglyglyglyvalileglnglyserala
245250255
serglualathrleuvalglyleuleuvalalalysasplysthrval
260265270
argargphemetasnasnasnproaspleuaspgluasngluilelys
275280285
alalysleuvalalatyrthrseraspglncysasnserservalglu
290295300
lysalaglyleuleuglysermetlysmetlysleuleulysalaasp
305310315320
alaaspglycysleuargglygluthrleulysargalailegluglu
325330335
asplysserglnglyleuileprocystyrvalvalalaasnleugly
340345350
thrthrglythrcysalapheaspproleuhisgluleuglyproile
355360365
cysserglugluaspiletrpleuhisvalaspalaalatyralagly
370375380
alaalapheleucysproglutyrarghisleumetlysglyileglu
385390395400
hisserglnserphevalthrasnalahislystrpleuprovalasn
405410415
pheaspcysseralamettrpvallysasnglytyraspilethrarg
420425430
alapheaspvalglnargiletyrleuaspaspvallysthrthrile
435440445
lysileproasptyrarghistrpglnmetproleuglyargargphe
450455460
argalaleulysleutrpthrvalmetargiletyrglyalaglugly
465470475480
leulysthrhisileargglnglnilegluleualaglntyrpheala
485490495
lysleuvalargalaaspgluargphevalileglyprogluprothr
500505510
metalaleuvalcyspheargleulysaspglyaspthrilethrarg
515520525
glnleuleugluasnilethrglnlyslyslysvalphemetvalala
530535540
glythrhisargaspargtyrvalileargphevalilecysserarg
545550555560
leuthrlyslysgluaspvalasptyrsertrpserglnilelyslys
565570575
gluthraspleuiletyrserasplysilehisasnlysalaglnile
580585590
proalaleugluglnphethrserarggluleucysglulysserlys
595600605
<210>12
<211>608
<212>prt
<213>家蚕(bombyxmori)
<400>12
metglyhishishishishishisglyserleuglnaspsergluval
151015
asnglnglualalysprogluvallysprogluvallysprogluthr
202530
hisileasnleulysvalseraspglysersergluilephephelys
354045
ilelyslysthrthrproleuargargleumetglualaphealalys
505560
argglnglylysglumetaspserleuargpheleutyraspglyile
65707580
argileglnalaaspglnalaprogluaspleuaspmetgluaspasn
859095
aspileileglualahisarggluglnileglyglymetaspalaasn
100105110
glnpheargglupheglyargalavalileaspmetleualasertyr
115120125
alagluasnileargasptyraspvalleuproservalgluprogly
130135140
tyrleuleuargalaleuprogluseralaprogluglnprogluasp
145150155160
trplysaspilemetlysasppheasnglnserilemetproglyval
165170175
thrhistrpglnserproglnphehisalatyrpheproserglyser
180185190
serphealaserileileglyasnmetleuseraspglyleualaval
195200205
valglyphesertrpmetalaserproalacysthrgluleugluval
210215220
valthrmetasntrpleuglylysleuleuaspleuproglugluphe
225230235240
leuasncysserserglyproglyglyglyvalileglnglyserala
245250255
serglualathrleuvalglyleuleuvalalalysasplysthrval
260265270
argargphemetasnasnasnproaspleuaspgluasngluilelys
275280285
alalysleuvalalatyrthrseraspglncyshisserservalglu
290295300
lysalaglyleuleuglysermetlysmetlysleuleulysalaasp
305310315320
alaaspglycysleuargglygluthrleulysargalailegluglu
325330335
asplysserglnglyleuileprocystyrvalvalalaasnleugly
340345350
thrthrglythrcysalapheaspproleuhisgluleuglyproile
355360365
cysserglugluaspiletrpleuhisvalaspalaalatyralagly
370375380
alaalapheleucysproglutyrarghisleumetlysglyileglu
385390395400
hisserglnserphevalthrasnalahislystrpleuprovalasn
405410415
pheaspcysseralamettrpvallysasnglytyraspilethrarg
420425430
alapheaspvalglnargiletyrleuaspaspvallysthrthrile
435440445
lysileproasptyrarghistrpglnmetproleuglyargargphe
450455460
argalaleulysleutrpthrvalmetargiletyrglyalaglugly
465470475480
leulysthrhisileargglnglnilegluleualaglntyrpheala
485490495
lysleuvalargalaaspgluargphevalileglyprogluprothr
500505510
metalaleuvalcyspheargleulysaspglyaspthrilethrarg
515520525
glnleuleugluasnilethrglnlyslyslysvalphemetvalala
530535540
glythrhisargaspargtyrvalileargphevalilecysserarg
545550555560
leuthrlyslysgluaspvalasptyrsertrpserglnilelyslys
565570575
gluthraspleuiletyrserasplysilehisasnlysalaglnile
580585590
proalaleugluglnphethrserarggluleucysglulysserlys
595600605
<210>13
<211>7422
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>6omt-4'omt-cnmt_pacyc184
<400>13
aactttcaccataatgaaataagatcactaccgggcgtattttttgagttatcgagattt60
tcaggagctaaggaagctaaaatggagaaaaaaatcactggatataccaccgttgatata120
tcccaatggcatcgtaaagaacattttgaggcatttcagtcagttgctcaatgtacctat180
aaccagaccgttcagctggatattacggcctttttaaagaccgtaaagaaaaataagcac240
aagttttatccggcctttattcacattcttgcccgcctgatgaatgctcatccggaattc300
cgtatggcaatgaaagacggtgagctggtgatatgggatagtgttcacccttgttacacc360
gttttccatgagcaaactgaaacgttttcatcgctctggagtgaataccacgacgatttc420
cggcagtttctacacatatattcgcaagatgtggcgtgttacggtgaaaacctggcctat480
ttccctaaagggtttattgagaatatgtttttcgtctcagccaatccctgggtgagtttc540
accagttttgatttaaacgtggccaatatggacaacttcttcgcccccgttttcaccatg600
ggcaaatattatacgcaaggcgacaaggtgctgatgccgctggcgattcaggttcatcat660
gccgtctgtgatggcttccatgtcggcagaatgcttaatgaattacaacagtactgcgat720
gagtggcagggcggggcgtaatttttttaaggcagttattggtgcccttaaacgcctggt780
gctacgcctgaataagtgataataagcggatgaatggcagaaattcgaaagcaaattcga840
cccggtcgtcggttcagggcagggtcgttaaatagccgcttatgtctattgctggtttac900
cggtttattgactaccggaagcagtgtgaccgtgtgcttctcaaatgcctgaggccagtt960
tgctcaggctctccccgtggaggtaataattgacgatatgatcatttattctgcctccca1020
gagcctgataaaaacggttagcgcttcgttaatacagatgtaggtgttccacagggtagc1080
cagcagcatcctgcgatgcagatccggaacataatggtgcagggcgcttgtttcggcgtg1140
ggtatggtggcaggccccgtggccgggggactgttgggcgctgccggcacctgtcctacg1200
agttgcatgataaagaagacagtcataagtgcggcgacgatagtcatgccccgcgcccac1260
cggaaggagctaccggacagcggtgcggactgttgtaactcagaataagaaatgaggccg1320
ctcatggcgttgactctcagtcatagtatcgtggtatcaccggttggttccactctctgt1380
tgcgggcaacttcagcagcacgtaggggacttccgcgtttccagactttacgaaacacgg1440
aaaccgaagaccattcatgttgttgctcaggtcgcagacgttttgcagcagcagtcgctt1500
cacgttcgctcgcgtatcggtgattcattctgctaaccagtaaggcaaccccgccagcct1560
agccgggtcctcaacgacaggagcacgatcatgcgcacccgtggccaggacccaacgctg1620
cccgagatgcgccgcgtgcggctgctggagatggcggacgcgatggatatgttctgccaa1680
gggttggtttgcgcattcacagttctccgcaagaattgattggctccaattcttggagtg1740
gtgaatccgttagcgaggtgccgccggcttccattcaggtcgaggtggcccggctccatg1800
caccgcgacgcaacgcggggaggcagacaaggtatagggcggcgcctacaatccatgcca1860
acccgttccatgtgctcgccgaggcggcataaatcgccgtgacgatcagcggtccagtga1920
tcgaagttaggctggtaagagccgcgagcgatccttgaagctgtccctgatggtcgtcat1980
ctacctgcctggacagcatggcctgcaacgcgggcatcccgatgccgccggaagcgagaa2040
gaatcataatggggaaggccatccagcctcgcgtcgctggcacgacaggtttcccgactg2100
gaaagcgggcagtgagcgcaacgcaattaatgtgagttagctcactcattaggcacccca2160
ggctttacactttatgcttccggctcgtatgttgtgtggaattgtgagcggataacaatt2220
tcacacaggaaacagctatgaccatgattacgaattcgagctcggtaccgatcccgcgaa2280
attaatacgactcactataggggaattgtgagcggataacaattcccctctagaaataat2340
tttgtttaactttaagaaggagatataccatgttagtgaagaagaaggacaatctctcat2400
ctcaagctaaactgtggaacttcatttatggttttgctgaatcactagtcctcaaatgtg2460
cagtgcaacttgatctagccaacataattcacaacagtggcacgtccatgactctttccg2520
agttatcttcgcgtcttccaagtcaacctgtcaatgaagacgccttgtatcgagtcatgc2580
gttacttggttcacatgaagctattcacaaaagcatcaatagatggagaactaagatatg2640
gacttgcaccaccagctaagtatcttgttaaaggttgggataaatgtatggttggctcaa2700
ttttagcaatcactgataaagatttcatggcaccatggcattaccttaaggatggattat2760
caggcgaaagtggtacagcgtttgagaaggccttggggacgaatatatgggggtacatgg2820
cagagcaccctgagaaaaaccagctatttaatgaagcaatggctaatgattcaaggctta2880
ttatgtctgcattggtgaaagaatgtggaaatatttttaatggtataactacacttgtgg2940
atgttggtggtggtactggaactgctgtgaggaatattgccaatgcatttccacatataa3000
agtgtactgtttatgatcttcctcatgtcattgctgattctcctgggtactccgaagttc3060
attgcgtggcaggtgatatgttcaagttcataccaaaggctgatgctatcatgatgaagt3120
gcatccttcacgactgggatgacaaagaatgcattgaaattctaaagcgatgcaaggagg3180
cagtaccagtcaaaggcgggaaagtgattatagtcgacattgtcttaaatgtgcaatcag3240
aacatccttataccaagatgagactgactttggatttggacatgatgctcaacactggag3300
gaaaagagaggactgaagaggaatggaagaagctcatccatgatgcagggtacaaagggc3360
ataagataacacaaattactgctgtacaatctgtgattgaggcttatccatattagatct3420
cgatcccgcgaaattaatacgactcactataggggaattgtgagcggataacaattcccc3480
tctagaaataattttgtttaactttaagaaggagatataccatgtctttccatgggaaag3540
atgatgttctggacatcaaagctcaagctcatgtgtggaaaatcatctatggttttgcag3600
attccctagtcctccgatgtgcagtggaacttggaatcgtcgacatcattgataacaaca3660
accaacccatggcacttgccgatctggcatctaagcttcctgtttccgatgtgaattgcg3720
ataatttgtatcggatattacgatacttggtgaaaatggaaatactgagagtggaaaaat3780
ctgatgatggtcagaagaagtacgcgcttgaacctattgcaacattgctttcaaggaatg3840
cgaagaggagtatggttccaatgattcttggaatgactcaaaaagattttatgactcctt3900
ggcattcaatgaaggatggcttaagtgacaatggtactgcttttgagaaggccatgggaa3960
tgactatatgggagtacttggaaggacaccctgatcaaagccaattattcaatgaaggca4020
tggccggtgaaacaaggcttctcacttcttcactcatatctggaagtagagatatgtttc4080
aaggtattgactcacttgttgatgttggtggaggaaatggtactactgtcaaggccattt4140
ctgacgcatttccacatatcaagtgcaccctctttgatctccctcatgtcattgccaatt4200
cctatgaccttcctaatattgaacgaattggtggcgacatgtttaaatccgtgcccagtg4260
cccaagctatcatactcaagctaattttgcacgattggaatgacgaagactcgatcaaga4320
ttttaaagcaatgcagaaatgcagtgccaaaagatggaggaaaagtgattatagtggatg4380
tggcattagatgaggagtcagaccatgagcttagcagcacacgattgatccttgatatcg4440
atatgttggtgaacactggtggtaaagagcggactaaagaggtttgggagaaaattgtga4500
aaagtgcaggatttagtggttgcaaaatcaggcacatagcggctatacaatcagtcattg4560
aggtttttccataggatccgatcccgcgaaattaatacgactcactataggggaattgtg4620
agcggataacaattcccctctagaaataattttgtttaactttaagaaggagatatacca4680
tggctgtggaagcaaagcaaacaaagaaggcagccatagtagagttgttaaaacagttgg4740
agctgggcttggttccatatgatgatattaagcagctcataaggagggaactggcaaggc4800
gcctgcaatggggttataaacctacttatgaagaacaaatagctgaaatccaaaacttaa4860
ctcattctctgcgacaaatgaaaattgcaacagaggttgagaccttggattcacaattgt4920
acgagattcctattgagtttctaaagattatgaatggaagtaacttaaaaggaagttgtt4980
gctacttcaaagaagattcaacaacattagatgaagctgagatagcgatgctggatttat5040
actgcgagagagctcaaatccaagatggacagagtgttcttgatcttggatgtgggcaag5100
gagctcttacattacatgttgcacagaaatataaaaactgtcgcgtaacagcagtaacaa5160
attcagtttcacaaaaagagtacattgaagaagaatcaaggagacgtaatttgttgaatg5220
tggaagtcaaattggcagacataaccacacatgagatggctgagacatacgatcgtattt5280
tggtaatagagttgtttgagcacatgaagaactatgaacttctcctgaggaaaatctcag5340
agtggatatcgaaagatgggcttctctttctagagcacatatgccacaagacctttgctt5400
accactatgagcctctagacgacgacgattggtttacagagtacgtgtttcctgctggga5460
ctatgatcataccatctgcatcgttctttttgtatttccaggatgacgtttcggttgtga5520
accattggactcttagtgggaagcacttttcgcgtaccaatgaggaatggttgaagagat5580
tggacgcaaaccttgatgttattaaaccaatgtttgagactttaatgggaaatgaggaag5640
aggcagtgaagttgattaactattggagaggattttgtttatctggaatggaaatgtttg5700
gatataacaatggtgaagaatggatggcaagtcatgttctgttcaagaaaaaatgagaat5760
tcgagctccgtcgacaagcttgcggccgcactcgagcaccaccaccaccaccactgagat5820
ccggctgctaacaaagcccgaaaggaagctgagttggctgctgccaccgctgagcaataa5880
ctagcataaccccttggggcctctaaacgggtcttgaggggttttttgctgcctgcaggc5940
atgcaagcttggcactggccgtcgttttacaacgtcgtgactgggaaaaccctggcgtta6000
cccaacttaatcgccttgcagcacatccccctttcgccagatcccgcaagaggcccggca6060
gtaccggcataaccaagcctatgcctacagcatccagggtgacggtgccgaggatgacga6120
tgagcgcattgttagatttcatacacggtgcctgactgcgttagcaatttaactgtgata6180
aactaccgcattaaagcttatcgatgataagctgtcaaacatgagaattacaacttatat6240
cgtatggggctgacttcaggtgctacatttgaagagataaattgcactgaaatctagaaa6300
tattttatctgattaataagatgatcttcttgagatcgttttggtctgcgcgtaatctct6360
tgctctgaaaacgaaaaaaccgccttgcagggcggtttttcgaaggttctctgagctacc6420
aactctttgaaccgaggtaactggcttggaggagcgcagtcaccaaaacttgtcctttca6480
gtttagccttaaccggcgcatgacttcaagactaactcctctaaatcaattaccagtggc6540
tgctgccagtggtgcttttgcatgtctttccgggttggactcaagacgatagttaccgga6600
taaggcgcagcggtcggactgaacggggggttcgtgcatacagtccagcttggagcgaac6660
tgcctacccggaactgagtgtcaggcgtggaatgagacaaacgcggccataacagcggaa6720
tgacaccggtaaaccgaaaggcaggaacaggagagcgcacgagggagccgccagggggaa6780
acgcctggtatctttatagtcctgtcgggtttcgccaccactgatttgagcgtcagattt6840
cgtgatgcttgtcaggggggcggagcctatggaaaaacggctttgccgcggccctctcac6900
ttccctgttaagtatcttcctggcatcttccaggaaatctccgccccgttcgtaagccat6960
ttccgctcgccgcagtcgaacgaccgagcgtagcgagtcagtgagcgaggaagcggaata7020
tatcctgtatcacatattctgctgacgcaccggtgcagccttttttctcctgccacatga7080
agcacttcactgacaccctcatcagtgccaacatagtaagccagtatacactccgctagc7140
gctgatgtccggcggtgcttttgccgttacgcaccaccccgtcagtagctgaacaggagg7200
gacagctgatagaaacagaagccactggagcacctcaaaaacaccatcatacactaaatc7260
agtaagttggcagcatcacccgacgcactttgcgccgaataaatacctgtgacggaagat7320
cacttcgcagaataaataaatcctggtgtccctgttgataccgggaagccctgggccaac7380
ttttggcgaaaatgagacgttgatcggcacgtaagaggttcc7422
<210>14
<211>5903
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>dhpaas-ptrchis2b
<400>14
gtttgacagcttatcatcgactgcacggtgcaccaatgcttctggcgtcaggcagccatc60
ggaagctgtggtatggctgtgcaggtcgtaaatcactgcataattcgtgtcgctcaaggc120
gcactcccgttctggataatgttttttgcgccgacatcataacggttctggcaaatattc180
tgaaatgagctgttgacaattaatcatccggctcgtataatgtgtggaattgtgagcgga240
taacaatttcacacaggaaacagcgccgctgagaaaaagcgaagcggcactgctctttaa300
caatttatcagacaatctgtgtgggcactcgaccggaattatcgattaactttattatta360
aaaattaaagaggtatatattaatgtatcgattaaataaggaggaataaaccatggatgg420
acgcgaaccagtttcgggaattcggcagagcagtcatcgatatgttggcgagctacgctg480
aaaacataagggattatgatgttctgccgtctgttgaacctggttacttgttaagagctc540
tgcctgaaagtgcacccgaacaaccggaggattggaaagacataatgaaagatttcaatc600
aatcaataatgccgggtgtaacacactggcaatctccgcagttccatgcatactttcctt660
ctggctcctcattcgcgagtattataggaaatatgcttagtgacggcttggcggttgtgg720
gatttagttggatggctagccctgcgtgtacagaactggaggtggtcacgatgaattggc780
ttggtaagctattagatttacctgaagaatttctaaattgctcttcgggtcccggaggcg840
gagttattcaaggatcagcgagcgaagccaccttagttggattgcttgtagccaaagata900
aaactgttcgccgtttcatgaataataatccagatctcgatgaaaacgagataaaagcaa960
aacttgtagcttatacatccgaccaatgtcactcgtcagtagaaaaagccggcttacttg1020
gttctatgaaaatgaaactgttgaaagcggatgctgatggatgcctacgcggagaaacat1080
tgaaaagggcaatcgaagaagataagtcgcaaggacttataccctgctatgtcgtcgcta1140
atctgggaacaactggaacttgtgcattcgatcctttacatgaattagggccaatatgta1200
gtgaggaagatatatggcttcacgtagatgcagcctatgcgggagcagcatttttgtgtc1260
ctgaatacagacacctaatgaaaggtattgaacattctcaatcgttcgtaacgaatgcac1320
ataagtggctaccggttaatttcgattgctctgctatgtgggttaaaaatggttatgata1380
taacgagagcattcgatgtacaaagaatttatttggatgatgtaaaaacgacaatcaaga1440
tcccagattacagacactggcaaatgccactaggccgtcgctttagagccttaaaattgt1500
ggacagttatgagaatttatggtgctgaaggtttgaaaacgcatatcagacagcaaatag1560
aattagcacagtattttgcaaaactggtacgtgcggacgaacgttttgtgattgggcccg1620
aaccgactatggcattggtctgttttagactgaaagacggtgacacaattacgcgacaat1680
tgttggaaaatataacgcaaaaaaagaaagtgtttatggtggccggaacgcacagggata1740
gatacgtcattagattcgtgatctgctcccgattgactaagaaggaagacgtcgattaca1800
gctggagccaaataaagaaagaaaccgacctcatctattcagataaaatacacaataaag1860
cacaaataccagctcttgaacaattcacttcaagagaactatgcgaaaaatctaagtaat1920
aactcgagatctgcagctggtaccatatgggaattcgaagctttctagaacaaaaactca1980
tctcagaagaggatctgaatagcgccgtcgaccatcatcatcatcatcattgagtttaaa2040
cggtctccagcttggctgttttggcggatgagagaagattttcagcctgatacagattaa2100
atcagaacgcagaagcggtctgataaaacagaatttgcctggcggcagtagcgcggtggt2160
cccacctgaccccatgccgaactcagaagtgaaacgccgtagcgccgatggtagtgtggg2220
gtctccccatgcgagagtagggaactgccaggcatcaaataaaacgaaaggctcagtcga2280
aagactgggcctttcgttttatctgttgtttgtcggtgaacgctctcctgagtaggacaa2340
atccgccgggagcggatttgaacgttgcgaagcaacggcccggagggtggcgggcaggac2400
gcccgccataaactgccaggcatcaaattaagcagaaggccatcctgacggatggccttt2460
ttgcgtttctacaaactctttttgtttatttttctaaatacattcaaatatgtatccgct2520
catgagacaataaccctgataaatgcttcaataatattgaaaaaggaagagtatgagtat2580
tcaacatttccgtgtcgcccttattcccttttttgcggcattttgccttcctgtttttgc2640
tcacccagaaacgctggtgaaagtaaaagatgctgaagatcagttgggtgcacgagtggg2700
ttacatcgaactggatctcaacagcggtaagatccttgagagttttcgccccgaagaacg2760
ttttccaatgatgagcacttttaaagttctgctatgtggcgcggtattatcccgtgttga2820
cgccgggcaagagcaactcggtcgccgcatacactattctcagaatgacttggttgagta2880
ctcaccagtcacagaaaagcatcttacggatggcatgacagtaagagaattatgcagtgc2940
tgccataaccatgagtgataacactgcggccaacttacttctgacaacgatcggaggacc3000
gaaggagctaaccgcttttttgcacaacatgggggatcatgtaactcgccttgatcgttg3060
ggaaccggagctgaatgaagccataccaaacgacgagcgtgacaccacgatgcctgtagc3120
aatggcaacaacgttgcgcaaactattaactggcgaactacttactctagcttcccggca3180
acaattaatagactggatggaggcggataaagttgcaggaccacttctgcgctcggccct3240
tccggctggctggtttattgctgataaatctggagccggtgagcgtgggtctcgcggtat3300
cattgcagcactggggccagatggtaagccctcccgtatcgtagttatctacacgacggg3360
gagtcaggcaactatggatgaacgaaatagacagatcgctgagataggtgcctcactgat3420
taagcattggtaactgtcagaccaagtttactcatatatactttagattgatttaaaact3480
tcatttttaatttaaaaggatctaggtgaagatcctttttgataatctcatgaccaaaat3540
cccttaacgtgagttttcgttccactgagcgtcagaccccgtagaaaagatcaaaggatc3600
ttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccgct3660
accagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaactgg3720
cttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggccacca3780
cttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtggc3840
tgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccgga3900
taaggcgcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaac3960
gacctacaccgaactgagatacctacagcgtgagctatgagaaagcgccacgcttcccga4020
agggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgcacgag4080
ggagcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccacctctg4140
acttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccag4200
caacgcggcctttttacggttcctggccttttgctggccttttgctcacatgttctttcc4260
tgcgttatcccctgattctgtggataaccgtattaccgcctttgagtgagctgataccgc4320
tcgccgcagccgaacgaccgagcgcagcgagtcagtgagcgaggaagcggaagagcgcct4380
gatgcggtattttctccttacgcatctgtgcggtatttcacaccgcatatggtgcactct4440
cagtacaatctgctctgatgccgcatagttaagccagtatacactccgctatcgctacgt4500
gactgggtcatggctgcgccccgacacccgccaacacccgctgacgcgccctgacgggct4560
tgtctgctcccggcatccgcttacagacaagctgtgaccgtctccgggagctgcatgtgt4620
cagaggttttcaccgtcatcaccgaaacgcgcgaggcagcagatcaattcgcgcgcgaag4680
gcgaagcggcatgcatttacgttgacaccatcgaatggtgcaaaacctttcgcggtatgg4740
catgatagcgcccggaagagagtcaattcagggtggtgaatgtgaaaccagtaacgttat4800
acgatgtcgcagagtatgccggtgtctcttatcagaccgtttcccgcgtggtgaaccagg4860
ccagccacgtttctgcgaaaacgcgggaaaaagtggaagcggcgatggcggagctgaatt4920
acattcccaaccgcgtggcacaacaactggcgggcaaacagtcgttgctgattggcgttg4980
ccacctccagtctggccctgcacgcgccgtcgcaaattgtcgcggcgattaaatctcgcg5040
ccgatcaactgggtgccagcgtggtggtgtcgatggtagaacgaagcggcgtcgaagcct5100
gtaaagcggcggtgcacaatcttctcgcgcaacgcgtcagtgggctgatcattaactatc5160
cgctggatgaccaggatgccattgctgtggaagctgcctgcactaatgttccggcgttat5220
ttcttgatgtctctgaccagacacccatcaacagtattattttctcccatgaagacggta5280
cgcgactgggcgtggagcatctggtcgcattgggtcaccagcaaatcgcgctgttagcgg5340
gcccattaagttctgtctcggcgcgtctgcgtctggctggctggcataaatatctcactc5400
gcaatcaaattcagccgatagcggaacgggaaggcgactggagtgccatgtccggttttc5460
aacaaaccatgcaaatgctgaatgagggcatcgttcccactgcgatgctggttgccaacg5520
atcagatggcgctgggcgcaatgcgcgccattaccgagtccgggctgcgcgttggtgcgg5580
atatctcggtagtgggatacgacgataccgaagacagctcatgttatatcccgccgtcaa5640
ccaccatcaaacaggattttcgcctgctggggcaaaccagcgtggaccgcttgctgcaac5700
tctctcagggccaggcggtgaagggcaatcagctgttgcccgtctcactggtgaaaagaa5760
aaaccaccctggcgcccaatacgcaaaccgcctctccccgcgcgttggccgattcattaa5820
tgcagctggcacgacaggtttcccgactggaaagcgggcagtgagcgcaacgcaattaat5880
gtgagttagcgcgaattgatctg5903
<210>15
<211>518
<212>prt
<213>罂粟(papaversomniferum)
<400>15
metglyserleuproalaasnasnpheglusermetserleucysser
151015
glnasnproleuaspproaspglupheargargglnglyhismetile
202530
ileasppheleualaasptyrtyrlysasnvalglulystyrproval
354045
argthrglnvalaspproglytyrleulyslysargleuprogluser
505560
alaprotyrasnprogluserilegluthrileleugluaspvalthr
65707580
asnaspileileproglyleuthrhistrpglnserproasntyrphe
859095
alatyrpheproserserglyserilealaglypheleuglyglumet
100105110
leuserthrglypheasnvalvalglypheasntrpmetserserpro
115120125
alaalathrgluleugluserilevalmetasntrpleuglyglnmet
130135140
leuthrleuprolysserpheleupheserseraspglysersergly
145150155160
glyglyglyvalleuglnglythrthrcysglualaileleucysthr
165170175
leuthralaalaargasplysmetleuasnlysileglyarggluasn
180185190
ileasnlysleuvalvaltyralaseraspglnthrleuseralaleu
195200205
glnlysalaalaglnilealaglyileasnprolysasnpheleuala
210215220
ilealathrserlysalathrasnpheglyleuserproasnserleu
225230235240
glnserthrileleualaaspilegluserglyleuvalproleuphe
245250255
leucysalathrvalglythrthrserserthralavalaspproile
260265270
glyproleucysalavalalalysleuhisglyiletrpvalhisile
275280285
aspalaalatyralaglyseralacysilecysprogluphearghis
290295300
pheileaspglyvalgluaspalaaspserpheserleuasnalahis
305310315320
lystrpphephethrthrleuaspcyscyscysleutrpvallysasp
325330335
seraspserleuvallysalaleuserthrserproglutyrleulys
340345350
asnlysalathraspserlysglnvalileasptyrlysasptrpgln
355360365
ilealaleuserargargpheargsermetlysleutrpleuvalleu
370375380
argsertyrglyilealaasnleuargthrpheleuargserhisval
385390395400
lysmetalalyshispheglnglyleuileglymetaspasnargphe
405410415
gluilevalvalproargthrphealametvalcyspheargleulys
420425430
proalaalailephearglyslysilevalgluaspasphisileglu
435440445
alaglnthrasngluvalasnalalysleuleugluservalasnala
450455460
serglylysiletyrmetthrhisalavalvalglyglyvaltyrmet
465470475480
ileargphealavalglyalathrleuthrglugluarghisvalthr
485490495
glyalatrplysvalvalglngluhisthraspalaileleuglyala
500505510
leuglygluaspvalcys
515
<210>16
<211>1554
<212>dna
<213>罂粟(papaversomniferum)
<400>16
atgggaagtcttccagctaataactttgaaagcatgtcgctgtgttcgcaaaatccactt60
gatccagatgaattcagaaggcaaggtcacatgattattgatttccttgctgattactac120
aaaaatgttgagaaatatccagttagaacccaagtcgatcccggttatttgaagaaaagg180
ttacccgaatcagctccgtacaatcctgaatccattgaaaccattcttgaagatgtgaca240
aatgatatcatccctggtctaactcactggcaaagtccaaattactttgctttttatcct300
tctagtggttctatcgctggtttcctaggggaaatgctaagtaccggatttaatgttgtc360
gggtttaattggatgtcatctccggccgcaactgagttggagagtattgttatgaattgg420
cttggccagatgcttacgcttcccaaatcatttctcttttcatcagacggaagttcggga480
ggtggaggagttttgcaagggactacttgtgaagccattttatgtactctaactgcggca540
agagataaaatgctgaacaaaattggtagagaaaatattaacaagttggttgtttatgct600
tctgatcaaaccctaagtgcactacagaaagctgctcaaattgctgggattaatcctaag660
aatttccttgctatcgcaacctccaaggctacaaattttggtctctctccaaattcactt720
caatcgacaattcttgctgatatcgaatccgggttagttccattgtttctctgtgccact780
gtcggaacaacttcttcaacagccgtagatcctattggcccactttgcgcggtggcaaaa840
ttgcacggtatttgggttcacattgatgctgcatacgctggaagtgcatgtatctgccca900
gagttcaggcacttcatcgatggtgtggaagatgcagactcatttagtctaaatgcacac960
aagtggttctttactactttggattgttgctgtttatgggtgaaagactctgattcactg1020
gtcaaggcattatcaacaagtccagaatatttgaagaacaaagcaactgattccaaacaa1080
gttatcgattacaaagattggcaaatagcgctcagcagaagattccgatccatgaaactc1140
tggttagtacttcgcagctatggaattgctaacttaagaaccttccttaggagccatgtt1200
aaaatggctaagcactttcaggggctcattggtatggacaacaggtttgagattgtagtt1260
cctagaacatttgccatggtgtgctttcgccttaaaccagctgccatttttaggaaaaaa1320
atagttgaagatgatcacattgaagctcaaacaaatgaggtaaatgcgaaattgcttgaa1380
tcagtcaatgcgtccgggaagatatacatgactcatgctgttgttggaggggtgtacatg1440
attcggtttgccgtcggggcaacactgacagaggaaagacatgtcactggggcttggaag1500
gtggtacaggagcatacagatgccatacttggtgcactaggtgaggatgtttgt1554
<210>17
<211>41
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>正向引物
<400>17
ctttgctttttatccttctagtggttctatcgctggtttcc41
<210>18
<211>41
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>反向引物
<400>18
ggaaaccagcgatagaaccactagaaggataaaaagcaaag41
<210>19
<211>40
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>正向引物
<400>19
gcttctgatcaaaccaacagtgcactacagaaagctgctc40
<210>20
<211>40
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>反向引物
<400>20
gagcagctttctgtagtgcactgttggtttgatcagaagc40
<210>21
<211>531
<212>prt
<213>罂粟(papaversomniferum)
<400>21
metglyserleuasnthrgluaspvalleugluhisserseralaphe
151015
glyalathrasnproleuaspprogluglupheargargglnglyhis
202530
metileileasppheleualaasptyrtyrargaspvalglulystyr
354045
provalargserglnvalgluproglytyrleuarglysargleupro
505560
gluthralaprotyrasnprogluserilegluthrileleuglnasp
65707580
valthrsergluileileproglyleuthrhistrpglnserproasn
859095
tyrtyralatyrpheproserserglyservalalaglypheleugly
100105110
glumetleuserthrglypheasnvalvalglypheasntrpmetser
115120125
serproalaalathrgluleugluglyilevalmetasptrpphegly
130135140
lysmetleuasnleuprolyssertyrleupheserglythrglygly
145150155160
glyvalleuglnglythrthrcysglualaileleucysthrleuthr
165170175
alaalaargasparglysleuasnlysileglyarggluhisilegly
180185190
argleuvalvaltyrglyseraspglnthrhiscysalaleuglnlys
195200205
alaalaglnilealaglyileasnprolysasnpheargalavallys
210215220
thrphelysalaasnserpheglyleualaalaserthrleuargglu
225230235240
valileleugluaspileglualaglyleuileproleuphevalcys
245250255
prothrvalglythrthrserserthralavalaspproileglypro
260265270
ilecysgluvalalalysglutyrglumettrpvalhisileaspala
275280285
alatyralaglyseralacysilecysprogluphearghispheile
290295300
aspglyvalgluglualaaspserpheserleuasnalahislystrp
305310315320
phephethrthrleuaspcyscyscysleutrpvallysaspproser
325330335
serleuvallysalaleuserthrasnproglutyrleuargasnlys
340345350
alathrgluserargglnvalvalasptyrlysasptrpglnileala
355360365
leuileargargpheargsermetlysleutrpmetvalleuargser
370375380
tyrglyvalthrasnleuargasnpheleuargserhisvalargmet
385390395400
alalysthrphegluglyleuvalglyalaaspargargphegluile
405410415
thrvalproargthrphealametvalcyspheargleuleupropro
420425430
thrthrvallysvalcysglygluasnglyvalhisglnasnglyasn
435440445
glyvalilealavalleuargasngluasnglugluleuvalleuala
450455460
asnlysleuasnglnvaltyrleuargglnvallysalathrglyser
465470475480
valtyrmetthrhisalavalvalglyglyvaltyrmetileargphe
485490495
alavalglyserthrleuthrglugluarghisvalilehisalatrp
500505510
gluvalleuglngluhisalaaspleuileleuserlyspheaspglu
515520525
alaasnphe
530
<210>22
<211>1593
<212>dna
<213>罂粟(papaversomniferum)
<400>22
atgggtagtcttaacacagaagatgttcttgaacacagttcagctttcggtgcaacaaac60
ccattagacccagaagaattcagaagacaaggtcacatgataatcgacttcttagctgat120
tattacagagatgtcgagaaatatccagttcgaagtcaagtagaacccggttatctacgt180
aaaagattaccagaaacagctccatacaatccagaatctatcgaaacgattcttcaagat240
gtgacgagtgagattattccagggttaacacattggcaaagtcctaattactatgcttat300
ttcccttccagtggttccgttgctggattcctcggtgaaatgcttagtactggttttaat360
gtcgttggttttaactggatgtcttcacctgctgctacagaactcgagggtattgttatg420
gattggttcggcaaaatgcttaaccttccaaaatcatacttgttctctggtaccggtggt480
ggagttttacagggaactacttgtgaagctatcttatgtacattaacagctgcaagagac540
agaaagttgaacaaaatcggtcgtgaacatatcggaagattagttgtttatggatctgat600
cagactcactgtgcactacagaaagctgctcagattgcaggaatcaaccccaagaacttc660
cgtgctgttaagacgtttaaagctaattcattcggattagcagcttcaactctaagagaa720
gttattcttgaagatattgaagccgggttgatccctctgtttgtatgtccaacggtcgga780
actacatcatcgactgcagtggatccaatcggtcctatctgtgaagtggcgaaagaatac840
gaaatgtgggttcacatcgacgcagcttacgctggaagtgcatgtatctgtcccgagttt900
agacactttatcgacggagtggaggaagcagattcattcagtctcaatgcgcataaatgg960
tttttcacaactttggattgttgttgtctttgggttaaagatccaagttccctggttaaa1020
gctctttccacaaatcctgagtacttgagaaacaaagctacagagtcaagacaggtcgtt1080
gactacaaagactggcagatcgcactcattcgccgattccgatccatgaagctttggatg1140
gttttacgtagctatggtgtgactaatctgagaaatttcttgaggagtcatgttagaatg1200
gcaaagacatttgagggtctcgttggtgcggataggagattcgaaattactgtgcctagg1260
acgtttgctatggtctgcttccgccttttacccccaacaaccgtaaaggtatgcggtgaa1320
aatggagtacaccagaatggaaacggggtcattgcagtactacgcaatgaaaatgaagaa1380
ttagtccttgctaataagctgaatcaagtgtatttgagacaggtcaaggcaacaggtagt1440
gtttatatgacacatgcggtcgtcggaggtgtctacatgattcggttcgcagtcggttcg1500
accttgacagaggaacgccatgttattcatgcttgggaggttttgcaagagcatgcagat1560
ctgattcttagtaagttcgatgaagcaaatttt1593
<210>23
<211>40
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>正向引物
<400>23
gtcctaattactatgctttttacccttccagtggttccgt40
<210>24
<211>40
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>反向引物
<400>24
acggaaccactggaagggtaaaaagcatagtaattaggac40
<210>25
<211>40
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>正向引物
<400>25
tatggatctgatcagactaactgtgcactacagaaagctg40
<210>26
<211>40
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>反向引物
<400>26
cagctttctgtagtgcacagttagtctgatcagatccata40
<210>27
<211>576
<212>dna
<213>黄唐松草(thalictrumflavum)
<400>27
atgaaattaatcctgaccggtcgcccgtttttacatcatcagggcatcatcaaccaggtg60
agcaccgtcaccaaagtcattcaccacgaactggaagtggcggccagcgccgatgatatc120
tggacggtttacagctggccgggtctggcgaaacatctgccggatctgctgcctggcgcg180
tttgaaaagctggaaattatcggcgatggcggcgttggcaccatcctcgatatgaccttt240
gttccgggcgaattcccgcatgaatacaaagaaaaatttattctggttgataacgaacat300
cgcctgaaaaaagtgcagatgattgaaggcggttatctggatctcggcgtgacctattat360
atggataccattcatgtggtgccgacgggtaaagatagctgcgtgattaaatcgtccacc420
gaatatcacgttaaaccggaatttgtgaaaatcgttgaaccgctgatcaccaccggcccg480
ctggcagcgatggcggatgccatcagcaaactggtgctggagcataaaagtaaaagcaac540
agcgatgaaatcgaagcggcgattattaccgtttaa576
<210>28
<211>191
<212>prt
<213>黄唐松草(thalictrumflavum)
<400>28
metlysleuileleuthrglyargpropheleuhishisglnglyile
151015
ileasnglnvalserthrvalthrlysvalilehishisgluleuglu
202530
valalaalaseralaaspaspiletrpthrvaltyrsertrpprogly
354045
leualalyshisleuproaspleuleuproglyalapheglulysleu
505560
gluileileglyaspglyglyvalglythrileleuaspmetthrphe
65707580
valproglyglupheprohisglutyrlysglulyspheileleuval
859095
aspasngluhisargleulyslysvalglnmetilegluglyglytyr
100105110
leuaspleuglyvalthrtyrtyrmetaspthrilehisvalvalpro
115120125
thrglylysaspsercysvalilelysserserthrglutyrhisval
130135140
lysprogluphevallysilevalgluproleuilethrthrglypro
145150155160
leualaalametalaaspalaileserlysleuvalleugluhislys
165170175
serlysserasnseraspgluileglualaalaileilethrval
180185190
<210>29
<211>641
<212>prt
<213>罂粟(papaversomniferum)
<400>29
metarglysvalilelystyraspmetgluvalalavalseralaasp
151015
servaltrpalavaltyrserserproaspileproargleuleuarg
202530
aspvalleuleuproglyvalpheglulysleuaspvalileglugly
354045
asnglyglyvalglythrvalleuaspilevalpheproproglyala
505560
valproargsertyrlysglulysphevalasnileaspargglulys
65707580
argleulysgluvalilemetilegluglyglytyrleuaspmetgly
859095
cysthrphetyrleuaspargilehisvalvalglulysthrlysser
100105110
sercysvalilegluserserilevaltyraspalalysgluglucys
115120125
alaaspalametserlysleuilethrthrgluproleulyssermet
130135140
alagluvalileserasntyrvalileglnlysgluserpheserala
145150155160
argasnileleuserlysglnservalvallyslysgluileargtyr
165170175
aspleugluvalproileseralaaspseriletrpservaltyrser
180185190
cysproaspileproargleuleuargaspvalleuleuproglyval
195200205
pheglulysleuaspvalilegluglyaspglyglyvalglythrval
210215220
leuaspilevalpheproproglyalavalproargsertyrlysglu
225230235240
lysphevalasnileaspargglulysargleulysgluvalilemet
245250255
ilegluglyglytyrleuaspmetglycysthrphetyrleuasparg
260265270
ilehisvalvalglulysserleusersercysvalilegluserser
275280285
ilevaltyrgluvallysgluglutyrvalaspalametserlysleu
290295300
ilethrthrgluproleulyssermetalagluvalileserasntyr
305310315320
valileglnarggluserpheseralaargasnileleuasnlysasn
325330335
serleuvallyslysgluileargtyraspleugluvalprothrser
340345350
alaaspseriletrpservaltyrsercysproaspileproargleu
355360365
leuargaspvalleuleuproglyvalpheglnlysleuaspvalile
370375380
gluglyasnglyglyvalglythrvalleuaspilevalphepropro
385390395400
glyalavalproargsertyrlysglulysphevalasnileasnhis
405410415
glulysargleulysgluvalilemetilegluglyglytyrleuasp
420425430
metglycysthrsertyrleuaspargilehisvalvalglulysthr
435440445
serlyssercysileilelysserservalvaltyrgluvallysgln
450455460
glucysvalglualametserlysleuilethrthrgluproleulys
465470475480
sermetalagluvalileserasntyralametlysglnglnserval
485490495
sergluargasnileprolyslysglnserleuleuarglysgluile
500505510
thrtyrgluthrgluvalglnthrseralaaspseriletrpasnval
515520525
tyrserserproaspileproargleuleuargaspvalleuleupro
530535540
glyvalpheglulysleuaspvalilealaglyasnglyglyvalgly
545550555560
thrvalleuaspilealapheproleuglyalavalargargargtyr
565570575
lysglulysphevallysileasnhisglulysargleulysgluval
580585590
valmetilegluglyglytyrleuaspmetglycysthrphetyrmet
595600605
aspargilehisvalpheglulysthrproasnsercysvalileglu
610615620
serserileilethrlysleulyslyssermetleuvallystrpleu
625630635640
ser
<210>30
<211>1926
<212>dna
<213>罂粟(papaversomniferum)
<400>30
atgcgcaaagtgattaaatacgatatggaagtggcggtttctgctgacagtgtctgggcg60
gtctattcatcgccagatattccacgcttgttgcgtgacgtcttactgcccggcgtgttt120
gaaaaattagacgtgatcgaaggcaatggcggcgtcggcaccgtgctggatattgttttc180
ccgccaggcgcggttccgcgttcttataaagagaagttcgtgaatattgatcgcgaaaaa240
cgcctgaaagaggtcattatgattgaagggggttatcttgatatgggctgcaccttctat300
ctggaccgtattcacgttgtcgaaaaaaccaaaagcagttgcgtcattgaatccagtatt360
gtgtacgacgcgaaagaagaatgcgccgatgcgatgagtaaactgattactaccgagccg420
ttgaaatcgatggcagaagtgattagcaattatgtcattcaaaaagagagttttagcgcg480
cgtaatattctgtcaaaacaaagcgtggtaaaaaaggaaattcgctatgacctggaggtg540
ccgatttctgcggattctatctggagcgtatatagctgtccggacatcccgcgtctgtta600
cgtgatgtgctgttgcctggcgtgttcgaaaagctggatgtgatcgagggcgatggcggc660
gttggtaccgttctggacattgtcttcccgccgggggcggtgccgcgcagctacaaagag720
aagtttgtcaatatcgaccgcgagaaacgtctcaaagaagttatcatgatcgaaggtggc780
tacctggatatgggctgtaccttttacctcgaccgcattcatgtggttgaaaaatctctg840
tcttcctgcgtgattgagagctccatcgtttatgaagtgaaagaagagtatgttgatgcc900
atgtcgaaactgatcaccacggaaccgctgaaaagcatggctgaagttatcagcaactat960
gtgattcagcgtgaaagcttcagcgcccgcaatatcctgaataaaaatagcctggttaag1020
aaagagattcgttatgatctggaagtcccgacctctgccgacagcatctggtcggtttac1080
agctgcccggatatcccacgtttattacgcgacgttttgctgccgggcgtattccagaaa1140
ctcgatgttatagaaggcaacggcggtgtcggtacggtactggatatcgtctttccaccg1200
ggtgcggtaccgcgcagttataaagagaaattcgttaacatcaaccatgaaaagcgtctg1260
aaggaggttattatgattgaaggcggttaccttgatatggggtgtaccagctatctcgat1320
cgcatccatgtcgttgagaaaacaagcaaatcctgtattattaagagcagcgttgtttat1380
gaggtgaagcaggaatgtgtggaagcgatgagcaaattgattaccaccgaaccactgaaa1440
tcaatggcggaagtcatcagtaactacgcaatgaaacagcagagcgtcagcgaacgtaac1500
attccgaaaaaacagtcgctgctgcgtaaagaaatcacctacgaaaccgaagtgcagacc1560
agtgctgatagcatttggaacgtgtattccagcccggatattcctcgcctgctgcgcgat1620
gtcctgcttcccggtgtttttgaaaaactggatgtaattgccggtaacggtggtgtgggc1680
acggtgttggatatcgcctttccgctgggcgcagtgcgtcgccgctacaaagaaaaattt1740
gttaaaattaaccacgaaaagcgcttaaaagaagtggtgatgatcgaaggtggctatctg1800
gacatgggttgtacgttttatatggatcgtatccacgtatttgagaaaacgccgaacagc1860
tgcgttattgaaagttcgattatcactaaacttaaaaaaagtatgctggtgaaatggctg1920
agttaa1926
- 还没有人留言评论。精彩留言会获得点赞!