GRAMC:顺式调节模块的基因组规模报道子测定方法
相关申请的交叉参考
本申请要求于2018年10月31日提交的美国临时申请号62/753,608的权益,其全部内容并入本文作参考。
发明领域
本申请提供了报道子核酸的文库,例如功能性调节元件,以及用于构建和使用这种文库的方法和试剂盒。
发明背景
顺式调节模块(crm),如增强子、启动子和阻抑物是基因组中的功能元件。据估计,有几十万的crm散布于人基因组中(niu,etal.nucleicacidsresearch46.11(2018):5395-5409;visel,etal.nature461.7261(2009):199;encodeprojectconsortium.nature489.7414(2012):57)。由于crm调节基因表达的时间、地点和水平,因此crm几乎参与了每个生物过程。各个crm直接与多种转录因子相互作用,多种crm组合起作用以介导基因调节活性(davidson.theregulatorygenome,elsevier(2006);levine,etal.cell157.1(2014):13-25;delaat,etal.nature502.7472(2013):499)。对这些元件进行综合实验鉴别是一个挑战。
鉴别crm的标准报道子测定是在基础启动子和报道基因上游克隆候选crm,及检验其驱动报道基因表达的能力(rosenthal,methodsinenzymology152(1987):704-720;arnone,etal.methodsincellbiology74.(2004):621-652;banerji,etal.cell27.2(1981):299-308)。相同的报道子构建体可以监测crm如何响应基因扰动(nam,etal.plosone7.4(2012):e35934)和转录结合位点中的突变(damle,etal.developmentalbiology357.2(2011):505-517;de-leon,etal.pnasusa107.22(2010):10103-10108;cui,etal.cellreports19.2(2017):364-374;emison,etal.nature434.7035(2005):857;guerreiro,etal.pnasusa110.26(2013):10682-10686)。然而,这种常规的逐个报道子测定不适于分析基因组中包含的数百万种潜在crm(例如高通量分析)。已经尝试了一些高通量分析,但是可能会有偏差。
发明概述
本发明公开了构建核酸分子报道子文库的方法,以及使用本文公开的方法产生的核酸分子报道子文库。与在标准报道子测定的情况一样,所公开的基因组规模报道子测定方法对于增强子和启动子都是有效的。所述测定还可以容纳长dna插入物,从而可以筛选完整crm而不是部分crm。过量基因组覆盖和dna条形码增加实验成本,而不足的基因组覆盖和dna条形码导致数据可靠性降低。然而,在本文公开的文库和方法中,文库中的基因组覆盖和dna条形码的数目是可调的。最后,与现有方法相比,本发明的测定方法使用可比或更少的输入材料即可生成可再现的数据。
在一些实施方案中,构建核酸分子报道子文库的方法包括分离选择的大小范围(例如大小范围为100-3000个碱基对长、例如约750-850个碱基对长)的多个核酸分子(例如基因组dna或合成dna),将所述多个分离的核酸分子与至少一个线性衔接子序列(如包括至少两个连续核糖核苷酸的衔接子,两侧是在3’末端的至少一个脱氧核糖核苷酸和在5’末端的至少一个脱氧核糖核苷酸)连接,以形成包含插入物(分离的核酸分子)和衔接子的多个环状核酸分子,将所述多个环状核酸分子与酶在足以产生多个线性核酸分子的条件下接触,并将所述多个线性核酸分子与至少一个报道子核酸融合以产生多个报道子构建体,形成核酸分子报道子文库。
可以使用任何核酸分子,包括基因组dna(例如基因组dna片段)或合成dna。在一些实例中,所述核酸是得自感兴趣的细胞或细胞群的基因组dna。基因组dna可以来自任何感兴趣的生物,包括但不限于动物(例如哺乳动物)、植物、细菌、真菌或古细菌。在一些实例中,所述方法包括使用凝胶电泳或基于珠的大小选择法以选择分离的核酸分子的大小范围。在一些实例中,所述方法包括使用连接酶将所述多个分离的核酸分子与至少一个线性衔接子序列连接。在一些实例中,所述连接酶包括dna连接酶,例如t4dna连接酶。所述线性衔接子序列可以包括至少两个连续核糖核苷酸,两侧是在3’末端的至少一个脱氧核糖核苷酸和在5'末端的至少一个脱氧核糖核苷酸(例如seqidno:1和/或seqidno:2所示核酸)。因此,连接产生包括插入物和衔接子的多个环状核酸分子。
在一些实例中,所述方法进一步包括在线性化所述环状核酸之前,在足以从所述多个环状核酸分子中去除线性核酸分子的条件下,使所述多个环状核酸分子与核酸外切酶(例如核酸外切酶i、核酸外切酶iii和/或λ核酸外切酶)接触。在一些实例中,所述方法然后包括在足以产生多个线性核酸分子的条件下,将所述多个环状核酸分子与核糖核酸内切酶(例如特异于dna双链体中核糖核苷酸的核糖核酸内切酶,例如rnasehii或尿嘧啶-dna糖基化酶)接触,每个所述线性核酸分子包含在插入物两侧的所述在3’末端的至少一个脱氧核糖核苷酸和所述在5’末端的至少一个脱氧核糖核苷酸。在一些实例中,所述方法包括将所述多个线性核酸分子与至少一个报道子核酸(例如编码荧光蛋白的核酸和/或包括条形码的核酸)融合以产生多个报道子构建体。
在一些实例中,所述方法进一步包括确定所述多个线性核酸分子的基因组覆盖。例如,确定基因组覆盖可以包括选择至少一个感兴趣的基因组区域,扩增所述多个线性核酸分子,以及确定选择的基因组区域是否存在于所述多个线性核酸分子中,在所述多个线性核酸分子中选择的基因组区域的拷贝数目和/或基因组覆盖。在一些实例中,通过选择一个或多个单拷贝靶进行分析以确定基因组覆盖。示例的单拷贝靶包括acta1、adm、adam12、axl、cfb、dlx5、kiss1、ncoa6、notch2、rpp30和top1。依据文库起始材料的来源可以选择其它或另外的单拷贝靶。
在一些实例中,所述方法包括将所述多个核酸分子与线性载体核酸(例如包括基础启动子的线性载体核酸)融合。因此,所述方法可用于产生包含核酸分子的多个线性载体。
在一些实例中,所述至少一个报道子核酸包括编码荧光蛋白的核酸,将所述多个线性核酸分子与至少一个报道子核酸的融合包括将所述多个线性载体与荧光报道子核酸融合。因此,所述方法可用于产生多个荧光报道子构建体。在其它实例中,所述至少一个报道子核酸包括编码条形码的核酸,将所述多个线性核酸分子与至少一个报道子核酸的融合包括将所述多个报道子线性载体与条形码核酸融合。因此,所述方法可用于产生多个条形码报道子构建体。在一些实例中,所述至少一个报道子核酸包括编码条形码的核酸和编码荧光蛋白的核酸,将所述多个线性载体与至少一个报道子核酸融合包括将所述多个报道子构建体与条形码核酸和编码荧光蛋白的核酸融合。因此,所述方法可用于产生多个荧光和条形码报道子构建体。
在一些实例中,所述方法进一步包括使所述多个线性载体的每一个与包括条形码报道子构建体的引物核酸接触。在一些实例中,所述方法随后包括进行聚合酶链反应(pcr)。因此,本文的方法可用于产生包括条形码报道子构建体的多个扩增的载体。在一些实例中,所述方法然后包括使包括条形码报道子构建体的扩增的载体自连接以产生环状载体。因此,本文的方法可用于产生条形码报道子构建体。在一些实例中,本文的方法进一步包括在足以从包含条形码报道子构建体的多个环状载体中去除线性核酸分子的条件下,使包括条形码报道子构建体的多个环状载体与核酸外切酶(例如核酸外切酶i、核酸外切酶iii和/或λ核酸外切酶)接触。
在构建核酸分子报道子文库的方法的具体实例中,所述方法包括分离多个选择的大小范围的核酸分子;使用连接酶连接所述多个分离的核酸分子与至少一个线性衔接子序列,其中所述线性衔接子序列包括至少两个连续核糖核苷酸,两侧是在3’末端的至少一个脱氧核糖核苷酸和在5’末端的至少一个脱氧核糖核苷酸,从而产生包括插入物和衔接子的多个环状核酸分子;在足以从所述多个环状核酸分子中去除线性核酸分子的条件下,使所述多个环状核酸分子与核酸外切酶接触;在足以产生多个线性核酸分子的条件下,使所述多个环状核酸分子与核糖核酸内切酶接触,每个所述线性核酸分子包括在插入物两侧的所述在3’末端的至少一个脱氧核糖核苷酸和所述在5’末端的至少一个脱氧核糖核苷酸;并且将所述多个线性核酸分子与至少一个报道子核酸融合以产生多个报道子构建体,例如通过(a)将所述多个核酸分子与线性载体核酸融合,从而产生多个包括所述核酸分子的线性载体;(b)使每个包括所述核酸分子的多个线性载体与包括条形码核酸的引物接触;及(c)进行聚合酶链反应(pcr)和连接反应,产生多个包括条形码报道子构建体的环状载体;并且在足以从包括条形码报道子构建体的多个环状载体中去除线性核酸分子的条件下,使包括条形码报道子构建体的多个环状载体与核酸外切酶接触。在一些实例中,所述方法进一步包括在将所述多个线性核酸分子与所述至少一个报道子核酸融合之前,确定插入物的基因组覆盖。
本文进一步公开了检测功能性核酸调节元件的方法(例如高通量方法)。在一些实例中,所述方法包括用本文公开的任何文库转染或转化至少一种感兴趣的细胞。示例的细胞包括动物(例如哺乳动物)细胞、细菌细胞、植物细胞、真菌细胞和古细菌细胞。例如,哺乳动物细胞可以包括心肌细胞、神经元、肝细胞、内皮细胞、胚胎干细胞、类器官衍生的细胞和诱导的干细胞。在一些实例中,所述方法包括从至少两个对象收集所述至少一种感兴趣的细胞,其中所述至少两个对象包括至少一个患有疾病或状况的对象和至少一个没有疾病或状况的对象。在一些实例中,所述法包括从至少一个对象收集所述至少一种感兴趣的细胞,其中在不同条件下从所述对象收集多个细胞。
在一些实例中,所述方法还包括测量所述至少一个报道子。例如,一些方法可以包括鉴别和/或量化所述至少一个报道子。在一些实例中,所述方法包括从感兴趣的细胞中分离rna以产生分离的rna。在一些实例中,鉴别所述报道子包括逆转录所述分离的rna以产生cdna,例如使用重组莫洛尼鼠白血病病毒(rmomulv)逆转录酶或禽成髓细胞瘤病毒(amv)逆转录酶。在特定的实例中,rna和dna依赖性dna聚合酶也用于逆转录所述分离的rna。
在一些实例中,所述方法然后包括检测所述cdna。在一些实例中,检测包括扩增所述cdna。例如,在至少一个报道子是至少一个独特的条形码核酸的情况下,扩增所述cdna可以包括选择特异于包括至少一个独特的核酸条形码的核苷酸的引物,使所述引物与所述cdna接触,并使用所述引物和cdna进行pcr以产生扩增的dna。
在一些实例中,所述方法进一步包括鉴别至少一个独特的核酸条形码。在一些实例中,通过对扩增的dna进行测序鉴别至少一个独特的核酸条形码。在一些实例中,所述方法还包括对至少一个独特的核酸条形码进行定量。
在本文所述方法的一些实例中,所述多个核酸分子例如使用本文所述方法产生的文库中的所述多个核酸分子,包括选择的感兴趣的基因组的至少80%。在本文所述方法的一些实例中,所述多个核酸分子包括选择的感兴趣的基因组中所述顺式调节元件的至少80%。
本文还公开了用于构建核酸分子报道子文库的试剂盒。在一些实例中,所述试剂盒包括本文所述的任何报道子核酸中的至少一个。在一些实例中,所述报道子核酸包括seqidno:1和/或seqidno:2所示线性衔接子序列。示例的试剂盒还可包括至少一个连接酶、核酸外切酶、核糖核酸内切酶和/或聚合酶。
本文进一步公开了用于高通量鉴别和/或定量功能性核酸调节元件的试剂盒。在一些实例中,所述试剂盒包括本文公开的任何文库,例如覆盖至少80%感兴趣基因组的文库。所述试剂盒的其它实例包括至少一种逆转录酶和/或pcr引物和高保真dna聚合酶。
根据以下参考附图进行的详细描述,本公开的前述和其它特征将变得更明显。
附图简述
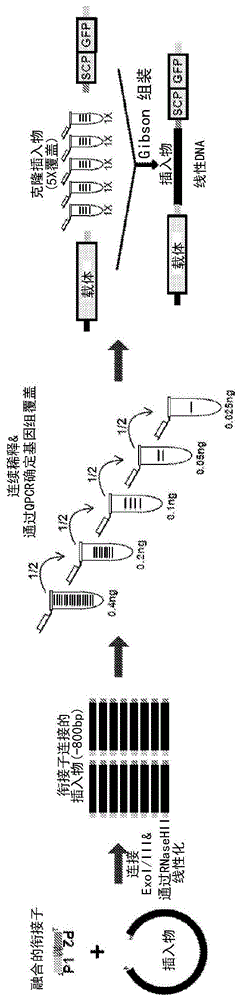
图1a-1d:gramc库建立。图1a示出了控制文库的基因组覆盖的示例性方法。通过与融合的衔接子连接,将经大小选择和末端修复的随机基因组dna片段环化。通过核酸外切酶处理去除线性dna及随后进行rnasehii消化以线性化连接产物和dice衔接子串联体。然后将衔接子连接的产物连续稀释,以通过qpcr确定每个稀释度的基因组覆盖。使用gibson
图2a-2e示出gramc的再现性和准确性。图2a示出gramc结果的再现性。在两批200mhepg2细胞中测试人gramc文库。crm活性针对输入质粒的拷贝数和背景活性(bg)被双重标准化。在一批细胞中驱动报道子表达≥5×bg及在另一批细胞中驱动报道子表达≥4.5×bg的插入物被认为是crm(有活性),crm调用是80%可再现的。在一批细胞中未达到截断值但仍≥3×bg及在另一批细胞中≥2.7×bg的插入物被认为是边缘活性的,具有62%的较低再现性。图2b示出通过单独报道子测定法对gramc结果的验证。通过qpcr在4批单独报道子测定中测试了一组11个crm(有活性)、5个边缘活性插入物和4个无活性插入物。将来自4批单独报道子测定的平均活性(实线)与gramc数据进行比较(r2=0.83)。图2c示出染色体1上crm(上方)和表达的基因(中间)的相关基因组分布。输入文库的基因组分布在下方示出。除去着丝粒的插入物。图2d示出具有多达100kb的表达基因(黑点)和未表达基因(灰点)侧翼区的2kb窗口中crm的富集。基因组平均值以虚线表示。基因区域位于位置0,包括外显子和内含子。基因上游区域在左半部分,下游区域在右半部分示出。图2e示出crm中encode染色质注释(g5,大于5×bg)相对于无活性插入物(l1,小于1×bg)的相对富集。encode注释基于其相对富集进行排序。
图3a-3g示出在chromhmm预测的强增强子中顺式调节活性和tfbs基序富集。图3a示出crm中预测的增强子的富集(黑色条)相对于通过gramc测量的crm活性(灰色条)。在两批gramc数据中根据插入物的平均活性将其分类:g5,大于5×bg;g3l5,等于或大于3×bg且小于5×bg;g2l3,等于或大于2×bg且小于3×bg;g1l2,等于或大于1×bg且小于2×bg;及l1,小于1×bg。图3b-3g示出活性逐渐减弱的预测的增强子相对于gramc鉴别的crm(g5)的相对基序富集(log2量表)。每个点表示tfbs基序,线表示两个数据集之间的2倍差异。每个图的左上角示出预测的增强子中每个仓(bin)的百分比比例。
图4a-4e示出crm驱动的基因调节程序预测。图4a示出crm中tfbs基序的丰度和富集。丰度是含给定tfbs基序的crm(g5组)或无活性组(l1组)的比例,相对富集是g5组和l1组之间基序富集的比率。垂直线表示基序相对富集的边界。标记了几个高度富集和丰富基序。图4b示出在g5组中预测的tfbs基序和encodechip-seq注释的富集的比较。图4c示出在其它细胞(细胞x)中pitx2或ikzf1对hepg2-crm的作用的两个供选假说。图4d-4e示出通过人pitx2(图4d)和人ikzf1(图4e)相对于cmv::gfp对照的异位表达,测试关于hepg2中未表达的转录因子的富集tfbs基序的假说。属于g5组的插入物以红点(基序+)或黑点(基序-)示出。两条黑色对角线表示扰动组相对于对照组之间的2倍差异。插入物箱线图示出使用两样本t检验在基序+相对于基序-插入物p值之间的差异。
图5a-5b示出gramc数据中重复元件的富集。如图3a-3g所示,插入物通过其在两批gramc数据中的平均活性而分类。图5a示出gramc数据中的重复元件的代表性家族。图中示出具有不同活性的基因组区域内重复元件的富集。g5组中的基因组区域被认为是crm。图5b示出gramc数据中alu元件的三个主要亚家族的富集。
图6a-6b示出融合的衔接子及衔接子连接的插入物的产生。图6a示出融合的衔接子。通过将两个5’-磷酸化寡聚物退火(上方是seqidno:1;下方是seqidno:2)以制备融合的衔接子。融合的衔接子含两个引物位点,p1(黄色箭头)和p2(洋红色箭头),用于扩增衔接子连接的基因组插入物。方框表示用于rnasehii切割的两个核糖核苷酸。图6b示出用于制备纯衔接子连接的插入物群的示例方法。插入物与融合的衔接子的连接产生对核酸外切酶处理具有抗性的环状dna。通过核酸外切酶i/iii去除所有不希望的线性dna。由于环状dna难以使用pcr扩增,因此环状连接产物可通过rnasehii线性化。然后将线性化的衔接子连接的插入物准备用于使用p1和p2引物的pcr扩增。
图7是示出制备用于gibson
图8示出用于为illuminanextseq500构建双末端测序文库的示例方法。用在插入物和n25条形码两侧的衔接子序列的2对引物(p2/np3和p1/p4)进行gramc文库的pcr,然后自连接,生成2个亚文库,其中n25与插入物的5’末端配对(hs800_14)或与插入物的3’末端配对(hs800_23)。核酸外切酶处理可确保在随后用另一组引物(对于hs800_23使用p1/p4,对于hs800_14使用p2/np3)进行第二轮插入物::n25盒扩增期间仅配对的环状连接物存活,生成2个测序文库hs800_2314和hs800_1423。pcr为illumina双末端测序加入pe1和pe2位点。每个测序文库使用七个异相(outofphase)引物加入pe1位点,以弥补侧翼衔接子序列多样性的缺乏。成相引物(phasedprimer)在pe1位点与相应的np3或p4位点之间掺入0n、2n、4n、6n、8n、10n和12n随机序列。在illuminanextseq500平台上对14个成相文库(phasedlibrary)进行测序。
图9示出从总rna中制备gramc测序文库的示例示意图。在第一个qc步骤(qc1)期间,通过qpcr测量gfpdna以监测rna样品中污染dna的去除。经dnase处理12小时后,如果gfpdna的ct值保持≤30,则继续进行dna消化。每6小时观察一次ct值,并重复这个程序直至ct值>30。作为逆转录(rt)的质量控制(qc)标准,将dnasei/exoi/exoiii消化的1000ng总rna用于标准rt反应。在第二个qc(qc2)步骤期间,监测基因组规模rt反应并根据需要补充试剂,直到gfpcdna的ct值在qc标准中ct值的1个周期内。
图10a-10f示出人基因组38的crm、表达的基因和输入的密度。图10a-10b示出人基因组38的gramccrm密度。图10c-10d示出人基因组38表达的基因密度。图10e-10f示出了人基因组38的gramc输入密度。
图11示出异位转录因子表达的western印迹确认。对用来自gramc文库的80k构建体及带有flag标记的egfp(对照)或flag标记的转录因子pitx2或ikzf1共转染的细胞样品进行抗-flag检测蛋白质表达。用抗gapdh对照印迹确认等价样品上样。
图12示出gramc的示例示意图,包括文库构建和鉴定以及文库在报道子测定中的应用以及数据去卷积。
图13示出从短随机寡聚物中逐步合成长随机dna序列的示例。从头合成大量的长随机dna序列仍然具有挑战性;因此,本发明示出一种简单的方法,从可商购的短随机单链dna(ssdna)产生长随机dna序列集(pool)。首先,使用多核苷酸激酶将2μg的ssdna磷酸化,然后将其通过随机六聚体、dntp和klenow酶转化为双链dna(dsdna)。同时,使用随机六聚体、dntp和klenow酶将1μg未磷酸化的ssdna转化为dsdna。其次,用在1×t4dna连接酶缓冲液中的200ng未磷酸化的dsdna和t4dna连接酶制备反应管。未磷酸化的dsdna与磷酸化的dsdna连接。第三,为了开始连接,将50ng磷酸化的dsdna(或部分未磷酸化的dna,例如约1/4)加入到连接反应管中。由于反应中存在过量的未磷酸化dna,因此大多数磷酸化dna与未磷酸化dna连接。每个未磷酸化的dna分子最多可以接受两个磷酸化dna分子(一个末端一个分子)。连接产物包括未磷酸化的5’-末端。重复所述连接程序至少一个循环(例如至少约1、2、3、4、5、6、7、8、9、10、12、15、18、20、25、30、45、50、60、75、90或100个循环,或约1-5、1-10、1-15、1-20、5-20、10-25、25-50或50-100个循环,或约16个循环)。循环数(x)预计为≥2xl/i,其中l和i分别是随机dna的期望长度和起始寡聚物的长度。例如,为了用长度为100bp的寡聚物合成长度约800bp的dna分子集,x应约≥16。第四,用dna修复酶(nebprecrrepairmix,cat#m0309s)修复连接产物中的缺口。第五,使用基于凝胶或基于珠的大小选择法富集期望长度的dna。然后,洗脱的dna准备用于文库构建(例如crm文库),例如具有至少约10、25、50、100、250、500、103、104、105、106、107、108或109个报道子构建体如约10-100、100-103、103-104、104-106、106-107、107-108、108-109或106-109报道子构建体或者约107个报道子构建体的文库(例如具有插入物),例如具有至少约50、100、200、300、400、500、750、800、900、1000、1200、1500、2000、2500或3000个碱基对长,如约50-3000或100-3000个碱基对长,例如约50-200、100-200、100-300、300-500、100-1500、500-1200、700-1000或750-850个碱基对长或者约800个碱基对长的插入物。长的随机dna序列的逐步合成也可以用于其它应用。
图14示出扰动实验的再现性。对于每个扰动实验,比较两个独立批次的80000个随机选择的报道子构建体。所有三个实验均高度可再现(pearson’sr≥0.97)。
序列表
如37c.f.r.1.822定义,使用核苷酸碱基的标准字母缩写和氨基酸的3个字母代码,显示在所附序列表中列出的核酸和氨基酸序列。每个核酸序列仅显示一条链,但应理解提及所展示链包括了互补链。序列表以ascii文本文件提交,创建于2019年10月30日,30kb,在此引用作为参考。在随附的序列表中:
seqidno:1和2是示例的线性衔接子核酸序列。
seqidno:3-116是示例的引物序列。
seqidno:117-124是示例的修剪衔接子序列。
发明详述
除非另有说明,否则根据常规用法使用技术术语。分子生物学中常用术语的定义可见于benjaminlewin,genesvii,publishedbyoxforduniversitypress,2000(isbn019879276x);kendrewetal.(eds.),theencyclopediaofmolecularbiology,publishedbyblackwellpublishers,1994(isbn0632021829);roberta.meyers(ed.),molecularbiologyandbiotechnology:acomprehensivedeskreference,publishedbywiley,john&sons,inc.,1995(isbn0471186341);及georgep.rédei,encyclopedicdictionaryofgenetics,genomics,andproteomics,2ndedition,2003(isbn:0-471-26821-6)。
除非上下文另外明确指出,否则单数形式“a”、“an”和“the”是指一个或多个。除非上下文另外明确指出,否则术语“或”是指所陈述的可替换元件中的一个元件或两个或更多个元件的组合。如本文所用,“包含”是指“包括”。因此,“包含a或b”是指“包括a、b或a和b”,而不排除其它元件。
还应理解,针对核酸或多肽给出的所有碱基大小或氨基酸大小以及所有分子量或分子质量值均为近似值,且为描述所提供。尽管与本文描述的那些类似或等价的方法和材料可以用于本公开的实施或测试中,但是在下文描述了合适的方法和材料。本文提及的所有出版物、专利申请、专利和其它参考文献以及
为了便于回顾本公开的各个实施方案,提供了对特定术语的以下解释。
衔接子(或衔接子序列或接头):单链或双链核酸(例如dna、rna或二者组合),其可以连接于其它核酸分子(例如dna和/或rna)的末端。可以合成双链衔接子,使其具有平端、粘端、或粘端和平端。在特定实例中,衔接子序列包括至少一个核糖核苷酸或至少两个连续核糖核苷酸(例如至少约2、3、4、5、6、7、8、9、10、25、50或100个核糖核苷酸,如约2-5、2-10、2-25、25-50或50-100个核糖核苷酸,或约2个核糖核苷酸),例如两侧是在3’末端的至少一个脱氧核糖核苷酸和在5’末端的至少一个脱氧核糖核苷酸(例如在3’末端和/或5’末端的至少约1、2、5、10、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、40、45、50、100、250、500或1000个脱氧核糖核苷酸,或者约5-45、10-40、15-35、20-30、1-50、1-100、1-250、1-500或1-1000个脱氧核糖核苷酸,或约21、28或29或约15-35或20-30个脱氧核糖核苷酸)。特别地,衔接子序列的非限制性实例包括seqidno:1和seqidno:2。
条形码(barcode):任何核酸或遗传标记。条形码可以是随机的(例如对于报道子应用,例如高通量应用)、半随机或非随机的(例如在分类应用中,例如特异于分类群的独特条形码以进行鉴别)。在特定实例中,条形码是随机条形码。在一些实例中,条形码来自条形码文库(例如预先存在或算法生成的条形码文库),如至少10、25、50、100、250、500、103、104、105、106、107、108或109个条形码如约10-100、100-103、103-104、104-106、106-107、107-108、108-109或106-109个条形码或者约107-2×107个条形码或者约2×107个条形码的文库。在特定实例中,条形码来自约2×107个条形码的随机文库。在一些实例中,条形码是短条形码,例如至少约5、10、15、20、25、30、35、40、45、50、75、100、250、500、1000、2000、3000或5000个核苷酸长或约5-10、10-20、15-40、20-30、10-50、10-75、10-100、100-250、250-500、500-1000、1000-3000或1000-5000个核苷酸长,或约20、25、30、15-40或20-30个核苷酸长。
互补:一个核酸分子被称为与另一个核酸分子互补是如果这两个核酸分子共享足够数目的互补核苷酸(例如a-t,a-u或g-c)以在所述链例如通过形成watson-crick、hoogsteen或反向hoogsteen碱基对而彼此结合(杂交)时形成稳定的双链体或三链体。当在所需条件下由于核酸分子中互补核苷酸之间的碱基配对导致核酸分子保持可检测地结合另一核酸时,发生稳定或特异性结合。
足以…的条件:任何允许所需活性的环境,例如允许两个分子之间(例如核酸与蛋白质之间或两个核酸之间)特异性结合或允许酶促活性(例如连接酶活性或核酸酶活性)的环境。
接触:置于直接物理相关中;包括固体和液体形式二者。例如,可以在体外或细胞中与核酸、蛋白质和/或酶(例如连接酶或核酸酶)发生接触。
检测:确定是否存在物质(如核酸分子和/或报道子分子)。在一些实例中,这可进一步包括鉴别和/或定量。例如,在特定实例中使用所公开的方法和检测探针可以确定核酸或报道子分子(例如报道子核酸)的存在、量和/或特性(identity)。
杂交:互补单链dna、rna或dna/rna杂交体形成双链体分子(也称为杂交复合物)的能力。
连接:通过一个核酸分子的3’羟基与另一个核酸分子的5’磷酸基团之间的磷酸二酯键将两个核酸分子连接在一起。催化核酸的并列的5’磷酸与3’羟基末端之间磷酸二酯键形成的酶称为连接酶。示例的连接酶包括dna连接酶(包括t4dna连接酶,t3dna连接酶,t7dna连接酶,taqdna连接酶(例如taqdna连接酶或高保真taqdna连接酶如hifitaqdna连接酶)),热稳定dna连接酶(例如催化无缺口地与互补dna链杂交和精确配对的两条相邻dna链的5’-磷酸与3’-羟基之间磷酸二酯键形成的热稳定连接酶,如9°
核酸酶:切割磷酸二酯键的酶。核酸内切酶是切割核苷酸链内的内部磷酸二酯键的酶(与在核苷酸链的末端切割磷酸二酯键的核酸外切酶相比)。核酸内切酶包括限制性核酸内切酶或其它位点特异性核酸内切酶,如核糖核酸内切酶(其在序列特异性位点切割rna),例如rnasehii(例如以去除任何核糖核苷酸)或尿嘧啶-dna糖基化酶。核酸酶的其它实例包括dnasei,s1核酸酶,celi核酸酶,绿豆核酸酶,核糖核酸酶a(rnasea),核糖核酸酶t1(rnaset1),核糖核酸酶h(rnaseh),rnasei,rnasephym,rnaseu2,rnaseclb,微球菌核酸酶和无嘌呤/无嘧啶核酸内切酶。核酸外切酶包括核酸外切酶i,核酸外切酶iii,λ核酸外切酶,核酸外切酶vii和bal31核酸酶。在本文的特定实例中,核酸酶是rna特异性核酸酶,如rnasehii(例如以去除任何核糖核苷酸)或尿嘧啶-dna糖基化酶,或核酸外切酶,如核酸外切酶i,核酸外切酶iii或λ核酸外切酶。
调节元件:核酸分子节段,其能增加或降低特定基因的表达。示例的调节元件包括激活物,例如启动子(例如启动基因转录的dna区域)和增强子(例如可以与其它分子如蛋白质相互作用以增加特定基因转录可能性的转录因子或dna区域),或阻抑物,如沉默子(例如与阻抑蛋白或转录因子结合时抑制dna序列转录为rna的dna区域)。
对象:任何多细胞脊椎动物生物体,如人和非人哺乳动物(例如兽医对象)。
载体:被用作人工将外来遗传材料携带至另一细胞中的运载体的核酸(例如dna或rna)。载体的示例类型包括质粒、病毒载体、粘粒和人工染色体。载体中包括的示例元件是复制起点、调节元件(例如启动子或增强子)、多克隆位点、标记和/或报道子。在具体实例中,载体可至少包括多克隆位点;调节元件;例如启动子(例如基础启动子和/或合成启动子,如超级核心启动子),增强子或阻抑物;及聚(a)尾部。
构建核酸分子报道子文库的方法
本文描述了构建核酸分子报道子文库的方法。因此,提供了可以确定在较大核酸序列如基因组(例如动物或人基因组)内感兴趣的核酸序列的存在与否和/或感兴趣的核酸序列的表达的方法,所述感兴趣的核酸序列例如是特异性和/或功能性序列。本文的方法可以与任何感兴趣的核酸序列一起使用,例如功能性核酸序列,例如调节基因表达的核酸序列(例如调节元件或模块,如顺式调节元件或模块)。在一些实例中,所述公开的方法允许鉴别或定量感兴趣的核酸序列。在一些实例中,所述方法包括分离多个核酸序列,如包括感兴趣的核酸序列的多个核酸序列,及将所述多个核酸序列与报道子核酸融合,产生多个报道子构建体。
在一些实施方案中,所述方法包括分离经选择的大小范围的多个核酸分子。可以使用任何核酸分子,包括基因组dna(例如基因组dna片段)或合成dna。在一些实例中,所述核酸是得自感兴趣的细胞或细胞群的基因组dna。可以使用任何细胞或细胞群,如动物细胞(例如哺乳动物细胞)、植物细胞、细菌细胞、真菌细胞或古细菌细胞。在一些实例中,哺乳动物细胞包括干细胞、神经细胞、心血管细胞、肝细胞、内皮细胞、上皮细胞、口腔细胞、生殖系统细胞、内分泌细胞、晶状体细胞、脂肪细胞、分泌细胞、肾细胞、细胞外基质细胞、收缩细胞、免疫细胞、血细胞或生殖细胞中的至少一种。在具体的非限制性实例中,哺乳动物细胞是心肌细胞、神经元、肝细胞、内皮细胞(例如人脐静脉内皮细胞,huvec,如在血管生成模型中)、胚胎干细胞、诱导的多能干细胞、hepg2细胞、lncap细胞、hela细胞、hct116细胞或k562细胞中的至少一种。在一些实例中,植物细胞包括分生组织细胞(包括分生组织衍生细胞)、薄壁组织细胞(例如叶肉细胞、转移细胞或绿色组织细胞)、厚角组织细胞、厚壁组织细胞(如厚壁组织硬化细胞或厚壁组织纤维)、管胞、管状分子(vesselelement)、韧皮部细胞(如筛管、伴细胞、韧皮纤维或韧皮硬化细胞)或表皮细胞(如气孔保卫细胞)中的至少一种。在具体的非限制性实例中,植物细胞是拟南芥属(arabidopsis)、大麻、玉米、水稻、大麦、小麦、柳枝稷、番茄、马铃薯、衣藻属(chlamydomonas)、水网藻属(hydrodictyon)、水绵属(spirogyra)和actebularia的至少一种。在一些实例中,细菌细胞包括革兰氏阴性或革兰氏阳性细菌细胞的至少一种,例如酸杆菌属(acidobacteria)、放线菌属(actinobacteria)、产水菌属(aquificae)、拟杆菌属(bacteroidetes)、嗜热丝菌属(caldiserica)、衣原体属(chlamydiae)、绿菌属(chlorobi)、绿弯菌属(chloroflexi)、产金菌属(chrysiogenetes)、蓝细菌属(cyanobacteria)、脱铁杆菌属(deferribacteres)、恐球菌-栖热菌属(deinococcus-thermus)、网团菌属(dictyoglomi)、埃希氏菌属(escherichia)、迷踪菌属(elusimicrobia)、纤维杆菌属(fibrobacteres)、厚壁菌属(firmicutes)、梭杆菌属(fusobacteria)、芽单胞菌属(gemmatimonadetes)、黏胶球形菌属(lentisphaerae)、硝化螺旋菌属(nitrospira)、浮霉菌属(planctomycetes)、变形菌属(proteobacteria)、螺旋体属(spirochaetes)、互养菌属(synergistetes)、软壁菌属(tenericutes)、热脱硫杆菌属(thermodesulfobacteria)、热袍菌属(thermotogae)或疣微菌属(verrucomicrobia)细胞。在一些实例中,真菌细胞包括木霉属(trichoderma)、链孢霉属(neurospora)、曲霉菌属(aspergillus)、红曲霉属(monascus)、毛霉菌属(mucor)、酵母菌属(saccharomyces)、毕赤酵母属(pichia)或根霉菌属(rhizopus)的至少一种。在一些实例中,古细菌细胞包括餐古菌属(cenarchaeum)、caldococcus、ignisphaera、酸叶菌属(acidilobus)、acidococcus、气火菌属(aeropyrum)、除硫球菌属(desulfurococcus)、燃球菌属(ignicoccus)、葡萄嗜热菌属(staphylothermus)、stetteria、厌硫球菌属(sulfophobococcus)、热盘菌属(thermodiscus)、热球形菌属(thermosphaera)、geogemma、超热菌属(hyperthermus)、热网菌属(pyrodictium)、火叶菌属(pyrolobus)、氨氧化古菌(nitrosopumilus(candidatus))、酸菌属(acidianus)、生金球形菌属(metallosphaera)、憎叶菌属(stygiolobus)、硫化叶菌属(sulfolobus)、硫磺球形菌属(sulfurisphaera)、热丝菌属(thermofilum)、暖枝菌属(caldivirga)、热棒菌属(pyrobaculum)、热分支菌属(thermocladium)、热变形菌属(thermoproteus)、火山鬃菌属(vulcanisaeta)、aciduliprofundum、古球菌属(archaeoglobus)、铁球状菌属(ferroglobus)、地球状菌属(geoglobus)、适盐菌属(haladaptatus)、盐碱球菌(halalkalicoccus)、haloalcalophilium、盐盒菌属(haloarcula)、盐杆菌属(halobacterium)、盐棒菌属(halobaculum)、盐二型菌属(halobiforma)、盐球菌属(halococcus)、极嗜盐菌属(haloferax)、盐几何菌属(halogeometricum)、盐微菌属(halomicrobium)、盐惰菌属(halopiger)、盐盘菌属(haloplanus)、haloquadra、盐棍菌属(halorhabdus)、盐红菌属(halorubrum)、盐八叠球菌(halosarcina)、盐简菌属(halosimplex)、盐陆生菌属(haloterrigena)、halovivax、钠白菌属(natrialba)、钠线菌属(natrinema)、钠杆菌属(natronobacterium)、钠球菌属(natronococcus)、盐碱湖菌属(natronolimnobius)、盐碱红菌属(natronorubrum)、methanoregula(candidatus)、甲烷砾菌属(methanocalculus)、甲烷杆菌属(methanobacterium)、甲烷短杆菌属(methanobrevibacter)、甲烷球形菌属(methanosphaera)、甲烷嗜热杆菌属(methanothermobacter)、甲烷热菌属(methanothermus)、甲烷暖球菌属(methanocaldococcus)、甲烷炎菌属(methanotorris)、产甲烷球菌属(methanococcus)、methanothermococcus、甲烷粒菌属(methanocorpusculum)、甲烷囊菌属(methanoculleus)、甲烷泡菌属(methanofollis)、产甲烷菌属(methanogenium)、甲烷裂叶菌属(methanolacinia)、甲烷微菌属(methanomicrobium)、甲烷盘菌属(methanoplanus)、甲烷螺菌科(methanospirillaceae)、甲烷螺旋菌属(methanospirillum)、甲烷鬃菌属(methanosaeta)、甲烷微球菌属(methanimicrococcus)、拟甲烷球菌属(methanococcoides)、甲烷盐菌属(methanohalobium)、甲烷嗜盐菌属(methanohalophilus)、甲烷叶菌属(methanolobus)、甲烷食甲基菌属(methanomethylovorans)、甲烷咸菌属(methanosalsum)、甲烷八叠球菌属(methanosarcina)、甲烷火菌属(methanopyrus)、古老球菌属(palaeococcus)、焦球菌属(pyrococcus)、热球菌属(thermococcus)、铁原体属(ferroplasma)、嗜酸菌属(picrophilus)、热原体属(thermoplasma)、初古菌门(korarchaeota)、纳古菌门(nanoarchaeota)或纳古菌属(nanoarchaeum)细胞中的至少一种。
经选择的大小范围的多个核酸分子可以来自任何来源,例如来自细胞的基因组或部分基因组,包括染色体dna和线粒体dna。因此,在一些实例中,所述分离的核酸是从选择的细胞类型或细胞类型群体分离的。将所述dna(例如基因组dna)例如通过消化、剪切、超声或其组合进行片段化。在一些实例中,所述核酸是合成dna,如选定长度或长度范围的随机双链dna序列。任何dna合成方法都可用于产生合成dna。在特定实例中,合成dna(例如选定大小范围的dna)可以通过连接小于选定大小范围的dna的两个或更多个dna分子产生(例如对于选定大小范围为约750-850个碱基对或约800个碱基对的dna而言,较小dna可以至少是约25、50、100、200、300或400个碱基对,或约25-50、25-100、25-200、25-400或100-400个碱基对,或约100个碱基对)。产生选定大小范围的合成dna核酸分子的示例方法示于图13。
在一些实例中,所述分离的核酸的大小范围为至少约50、100、200、300、400、500、750、800、900、1000、1200、1500、2000、2500或3000个碱基对长,如约50-3000或100-3000个碱基对长,如约50-200、100-200、100-300、300-500、100-1500、500-1200、700-1000、700-900或750-850个碱基对长或约800个碱基对长。可以使用任何方法选择期望大小范围的多个核酸分子。在一些实例中,使用凝胶电泳(例如使用琼脂糖凝胶,如人工制备的琼脂糖凝胶或琼脂糖凝胶盒,如使用恒定电压或变化电压,如至少1%、1.2%、1.5%、2%、3%或5%琼脂糖凝胶,如1-5%、1-2%、2-3%或3-5%琼脂糖凝胶或1.2%琼脂糖凝胶)或基于珠的大小选择法(例如固相可逆固定化spri,如使用顺磁珠,例如带有羧基涂层的顺磁性珠)选择所述多个核酸分子。
在一些实例中,所述方法包括将核酸分子(例如选定大小的多个分离的核酸分子,在本文中也称为“插入物”)连接于衔接子序列(例如至少一个衔接子序列,如至少一个线性衔接子序列)。可以使用任何衔接子序列,如能例如通过与多个分离的核酸分子连接而形成环状核酸分子(例如多个环状核酸分子)的线性衔接子序列。在一些实例中,衔接子序列包括核糖核苷酸和脱氧核糖核苷酸。在特定实例中,衔接子序列包括一个核糖核苷酸或至少两个连续核糖核苷酸(例如至少约2、3、4、5、6、7、8、9、10、25、50或100个核糖核苷酸,如约2-5、2-10、2-25、25-50或50-100个核糖核苷酸,或约2个核糖核苷酸)。在一些实例中,衔接子序列包括一个核糖核苷酸或至少两个连续核糖核苷酸,两侧是在3’末端的至少一个脱氧核糖核苷酸(例如在3’末端的至少约1、2、5、10、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、40、45、50、100、250、500或1000个脱氧核糖核苷酸,或约5-45、10-40、15-35、20-30、1-50、1-100、1-250、1-500或1-1000个脱氧核糖核苷酸,或约21、28或29或约15-35或20-30个脱氧核糖核苷酸)及在5’末端的至少一个脱氧核糖核苷酸(例如在5’末端的至少约1、2、5、10、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、40、45、50、100、250、500或1000个脱氧核糖核苷酸,或约5-45、10-40、15-35、20-30、1-50、1-100、1-250、1-500或1-1000个脱氧核糖核苷酸,或约21、28或29个或约15-35或20-30个脱氧核糖核苷酸)。在特定实例中,线性衔接子序列可包括以下序列:ctgctgaatcactagtgaattattacccrurucaagacactactctccagcagt(seqidno::1)或者ctgctggagagtagtgtcttgraragggtaataattcactagtgattcagcagt(seqidno:2),其中“ru”和“ra”表示核糖核苷酸。在特定实例中,衔接子是通过seqidno:1和2的核酸杂交制备的双链线性衔接子。
使用任何连接方法(例如连接酶介导的连接或化学连接),将所述多个分离的核酸分子(例如多个插入物)连接于衔接子序列(例如至少一个衔接子序列,如至少一个线性衔接子序列,例如seqidno:1和/或seqidno:2)。在一些实例中,至少一种连接酶用于连接。可以使用本文描述的任何核酸或衔接子序列。在一些实例中,连接方法足以形成包括“插入物”核酸分子和衔接子序列(例如包括seqidno:1和seqidno:2的双链衔接子)的环状核酸分子(例如多个环状核酸分子)。因此,在特定实例中,所述方法可用于产生均具有插入物和衔接子序列的多个环状核酸分子。在一些实例中,使用dna连接酶。可以使用足以连接核酸的任何连接酶(例如t4dna连接酶)。可以使用的连接酶的实例包括dna连接酶(包括t4dna连接酶,t3dna连接酶,t7dna连接酶,taqdna连接酶(例如taqdna连接酶或高保真taqdna连接酶,如hifitaqdna连接酶),热稳定dna连接酶(例如,热稳定连接酶,其催化两条与互补dna链杂交和精确配对的无缺口的相邻dna链的5’-磷酸和3’-羟基之间磷酸二酯键的形成,如9°
在一些实施方案中,所述方法进一步包括在足以从环状核酸分子(例如本文所述的任何环状核酸分子,例如多个环状核酸分子)中去除线性核酸的条件下,将所述多个环状核酸分子与至少一种酶(例如至少约1、2、5或10种酶,或约1-2、1-5或1-10种酶或约1或2种酶)接触,所述酶特异于从多核苷酸分子末端去除连续核苷酸(例如至少一种核酸外切酶,如至少约1、2、5或10种核酸外切酶,或约1-2、1-5或1-10种核酸外切酶,或约1或2种核酸外切酶)。在一些实例中,所述至少一种核酸外切酶包括核酸外切酶i、核酸外切酶iii和/或λ核酸外切酶。在特定实例中,所述至少一种核酸外切酶是核酸外切酶i和核酸外切酶iii。
在一些实施方案中,所述方法包括将包括插入物和衔接子序列的所述多个环状核酸分子与特异于分离多核苷酸链内核苷酸的酶(例如除了5’或3’末端的那些核苷酸之外的核苷酸,如核酸内切酶)在足以从包括插入物和衔接子的所述多个环状核酸分子中产生线性核酸分子(例如多个线性核酸分子)的条件下接触。在一些实例中,产生的线性核酸分子均包括在5’末端的至少一个脱氧核糖核苷酸和在3’末端的至少一个脱氧核糖核苷酸,例如在插入物两侧(例如本文所述的任何插入物)。在一些实例中,产生的线性核酸分子包括插入物,其两侧是在5’末端的至少一个脱氧核糖核苷酸和在3’末端的至少一个脱氧核糖核苷酸。例如,所述在5’末端或3’末端的至少一个脱氧核糖核苷酸可以包括至少一个脱氧核糖核苷酸,如约至少约1、2、5、10、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、40、45、50、100、250、500或1000个脱氧核糖核苷酸,或约5-45、10-40、15-35、20-30、1-50、1-100、1-250、1-500或1-1000个脱氧核糖核苷酸,或约21、28或29个或约15-35或20-30个脱氧核糖核苷酸。在特定实例中,所述酶特异于去除双链核酸内的核糖核苷酸(例如核糖核酸内切酶)。例如,所述酶可以从环状核酸(例如本文所述的任何环状核酸分子,如多个环状核酸分子)去除至少一个核糖核苷酸,如约至少约2、3、4、5、6、7、8、9、10、25、50或100个核糖核苷酸,如约2-5、2-10、2-25、25-50或50-100个核糖核苷酸,或约2个核糖核苷酸。在特定实例中,所述酶(例如核糖核酸内切酶)可以包括rnasehii(例如以去除任何核糖核苷酸)或尿嘧啶-dna糖基化酶(例如以去除尿嘧啶)。线性化所述环状核酸产生包括所述插入物核酸及3’末端的至少一个脱氧核糖核苷酸和5’末端的至少一个脱氧核糖核苷酸的多个线性核酸分子。
在一些实施方案中,所述方法包括将通过线性化包括插入物和在3’末端的至少一个脱氧核糖核苷酸和在5’末端的至少一个脱氧核糖核苷酸的环状核酸而获得的多个线性核酸分子融合至至少一个报道子核酸(例如产生多个报道子构建体,如核酸分子报道子文库)。可以使用任何报道子核酸,例如荧光或条形码报道子核酸,如编码荧光蛋白的核酸和/或包括条形码的核酸。在一些实例中,至少一个报道子是编码荧光蛋白的核酸。可以编码任何荧光蛋白,如蓝色、紫色、绿色、黄色、橙色或红色荧光蛋白,或具有这种荧光的任何组合或变化的蛋白。在特定实例中,至少一个报道子核酸是编码绿色荧光蛋白(gfp)的核酸。在其它实例中,至少一个报道子核酸是包括条形码的核酸(例如核酸或遗传标记)。任何核酸或遗传标记都可以用作条形码。在一些实例中,条形码是短核酸或遗传标记,例如至少约5、10、15、20、25、30、35、40、45、50、75、100、250、500、1000、2000、3000或5000个核苷酸长、或约5-10、10-20、15-40、20-30、10-50、10-75、10-100、100-250、250-500、500-1000、1000-3000或1000-5000个核苷酸长、或约20、25、30、15-40或20-30个核苷酸长的核酸或遗传标记。在进一步实例中,所述报道子包括至少一个编码荧光蛋白的核酸和至少一个条形码核酸。
在特定实例中,至少一个报道子核酸是条形码核酸。可以使用任何核酸条形码;例如可以使用随机的、半随机的或非随机的条形码,如来自条形码文库的。在特定实例中,所述条形码是随机条形码。在一些实例中,所述条形码来自条形码文库(例如预先存在或算法生成的条形码文库),如至少10、25、50、100、250、500、103、104、105、106、107、108或109个条形码、如约10-100、100-103、103-104、104-106、106-107、107-108、108-109或106-109个条形码或约107-2×107个条形码或约2×107个条形码的文库。在特定实例中,所述条形码来自约2×107个条形码的随机文库。
在一些实施方案中,所述方法包括将包括插入物核酸和在3’末端的至少一个脱氧核糖核苷酸和在5’末端的至少一个脱氧核糖核苷酸的线性核酸分子以及报道子融合于线性载体核酸以产生多个线性载体。可以使用任何线性载体核酸。例如,线性载体核酸可包括核酸酶切割位点和转录或翻译调节元件(如启动子、增强子、阻抑物和/或聚(a)尾部)。在一些实例中,所述线性载体核酸可包括至少一个启动子,如基础启动子和/或合成启动子。例如,所述线性载体核酸可包括至少约1、2、3、4、5、6、8或10个启动子,或约1-4、5-10或1-10个启动子。在一些实例中,至少一个启动子如基础和/或合成启动子可包括至少一个启动子基序,如至少约1、2、3、4、5、6、8或10个启动子基序,或约1-4、5-10或1-10个启动子基序或约4个启动子基序,例如合成启动子可包括tata盒、起始子(inr)、十基序元件(mte)、下游启动子元件(dpe)、b识别元件(bre)、e-box、ccaat盒、nrf-1、gabpa、yy1、actacanntccc和/或十聚体启动子基序。在特定实例中,至少一个启动子是合成启动子,其包括tata盒、inr、mte和dpe基序(例如超级核心启动子);其它示例的启动子可见于morgan,addgeneblog:“plasmids101:thepromoterregion–let'sgo!”,2014,在此以其全部内容并入本文作参考。
包括在3’末端具有至少一个脱氧核糖核苷酸和在5’末端具有至少一个脱氧核糖核苷酸的插入物核酸的线性核酸分子可以在任何时间与线性载体核酸融合,例如线性核酸分子与至少一个报道子核酸融合时、之前或之后。在一些实例中,所述线性载体核酸包括至少一个报道子核酸(例如编码荧光蛋白如绿色荧光蛋白的至少一个报道子核酸,或包括至少一个条形码的至少一个报道子核酸),因此将线性核酸分子与线性载体核酸的融合包括与至少一个报道子核酸的融合。在一些实例中,所述方法包括在线性核酸分子与至少一个报道子核酸融合之前(例如编码荧光蛋白的核酸或包括条形码的核酸),将线性核酸分子与线性载体核酸融合。例如,将多个线性核酸分子与至少一个报道子核酸的融合可以包括将多个线性载体与编码荧光蛋白的报道子核酸(例如荧光报道子核酸)融合以产生多个荧光报道子构建体。在一些实例中,将多个线性核酸分子与至少一个报道子核酸融合可包括将多个线性载体与包括条形码的报道子核酸(例如条形码报道子核酸)融合以产生多个条形码报道子构建体。在其它实例中,在与线性载体核酸融合之前,所述线性核酸包括具有在3’末端的至少一个脱氧核糖核苷酸和在5’末端的至少一个脱氧核糖核苷酸的插入核酸和报道子核酸。
所述方法包括将任意数目的报道子核酸与多个线性核酸分子或包括核酸分子的多个线性载体融合,例如至少约1、2、3、4、5、10、15、20或25个、或约1-2、1-5、1-10、10-20、15-25或1-25个或约2个报道子核酸。在一些实例中,所述方法包括将多个线性核酸分子或包括核酸分子的多个线性载体与荧光报道子核酸(例如编码gfp的报道子核酸)融合以产生多个荧光报道子构建体。在一些实例中,所述方法包括将多个线性核酸分子或包括核酸分子的多个线性载体与条形码报道子核酸(例如包括短条形码例如约25个核苷酸长的条形码的报道子核酸)融合以产生多个条形码报道子构建体。在一些实例中,所述方法包括将多个线性核酸分子或包括核酸分子的多个线性载体与荧光报道子核酸和条形码报道子核酸(例如编码gfp的报道子核酸和包括短条形码如约25个核苷酸长的条形码的报道子核酸)融合以产生多个荧光和条形码报道子构建体。在特定实例中,所述方法包括将包括核酸分子的多个线性载体与荧光报道子核酸和/或条形码报道子核酸(例如编码gfp的报道子核酸和/或包括短条形码例如约25个核苷酸长的条形码的报道子核酸)融合以产生多个荧光和条形码报道子构建体。
在一些实施方案中,将多个线性核酸分子或包括核酸分子的多个线性载体与条形码报道子核酸融合包括将多个线性核酸分子或多个线性载体与引物核酸接触,所述线性核酸分子包括具有在3’末端的至少一个脱氧核糖核苷酸和在5’末端的至少一个脱氧核糖核苷酸的插入物核酸,所述线性载体包括具有在3’末端的至少一个脱氧核糖核苷酸和在5’末端的至少一个脱氧核糖核苷酸的插入物核酸,所述引物核酸包括条形码报道子核酸(例如包括短条形码如约25个核苷酸长的条形码的报道子核酸)。在一些实例中,使用多个线性核酸分子或包括线性核酸分子的多个线性载体及包括条形码报道子核酸的至少一个引物核酸进行聚合酶链反应(pcr),如用于延伸线性核酸分子或多个线性载体以产生多个条形码报道子构建体或包括条形码报道子构建体的多个线性载体。在特定实例中,使用包括核酸分子的多个线性载体和包括条形码报道子核酸的引物核酸进行聚合酶链反应(pcr),以产生包括条形码报道子构建体的多个线性载体。
在一些实例中,所述方法包括使用连接酶将包括报道子构建体(例如荧光和/或条形码报道子构建体)的多个线性载体的末端连接以产生包括报道子构建体(例如荧光和/或条形码报道子构建体)的多个环状载体。在特定实例中,所述方法包括使用连接酶将包括条形码报道子构建体的多个线性载体的末端连接以产生包括条形码报道子构建体的多个环状载体。可以使用本文所述的任何连接酶(例如dna连接酶,如t4dna连接酶)。在一些实例中,所述连接酶足以连接双链核酸的平端(例如t4dna连接酶或t3dna连接酶)。在特定实例中,所述连接酶是t4dna连接酶。在一些实例中,所述方法还包括将包括条形码报道子构建体的多个环状载体与至少一个核酸外切酶接触,以从所述多个环状载体中去除线性核酸分子。可以使用本文所述的任何核酸外切酶(例如核酸外切酶i,核酸外切酶iii和/或λ核酸外切酶)。在特定实例中,所述至少一个核酸外切酶是核酸外切酶i和核酸外切酶iii。
在一些实施方案中,所述方法还包括确定多个线性核酸分子的基因组覆盖,例如,其中所述多个线性核酸分子包括基因组dna时。可以随时确定基因组覆盖。在一些实例中,在将多个线性核酸分子与报道子核酸融合之前确定基因组覆盖,所述线性核酸分子包括具有在3’末端的至少一个脱氧核糖核苷酸和在5’末端的至少一个脱氧核糖核苷酸的插入物核酸。在特定实例中,可以使用多个线性核酸分子(例如包括核酸分子和衔接子序列的线性核酸分子)确定覆盖。基因组覆盖可以使用任何方法确定。在特定实例中,通过选择至少一个感兴趣的基因组区域(例如整个基因组或部分基因组)、扩增所述多个线性核酸分子(例如使用pcr,如定量pcr,即qpcr)并确定在所述多个线性核酸分子中是否存在选择的基因组区域,由此确定基因组覆盖。在一些实例中,如在线性核酸分子包括核酸分子和衔接子序列的情况下,使用与衔接子序列互补的引物(例如与全部或部分衔接子序列互补的引物,如位于核酸分子5’的全部或部分衔接子序列)进行pcr。
在构建核酸分子报道子文库的方法的特定实例中,所述方法包括分离选定大小范围(例如至少约50、100、200、300、400、500、750、800、900、1000、1200、1500、2000、2500或3000个碱基对长、如约50-3000或100-3000个碱基对长、如约50-200、100-200、100-300、300-500、100-1500、500-1200、700-1000或750-850个碱基对长或约800个碱基对长)的多个核酸分子;使用连接酶(例如t4连接酶)将所述多个核酸分子与至少一个线性衔接子序列连接,其中所述线性衔接子序列包括至少两个连续核糖核苷酸,两侧是在3’末端的至少一个脱氧核糖核苷酸和在5’末端的至少一个脱氧核糖核苷酸(例如在3’末端或5’末端的至少约21、28或29或约15-35或20-30个脱氧核糖核苷酸),如seqidno:1或seqidno:2,从而产生包括插入物和衔接子的多个环状核酸分子;在足以从所述多个环状核酸分子中去除线性核酸分子的条件下,使所述多个环状核酸分子与核酸外切酶(例如核酸外切酶i和/或核酸外切酶iii)接触;在足以产生多个线性核酸分子的条件下使所述多个环状核酸分子与核糖核酸内切酶(例如rnasehii)接触,所述多个线性核酸分子每一个均包括在插入物两侧的所述在3’末端的至少一个脱氧核糖核苷酸和所述在5’末端的至少一个脱氧核糖核苷酸;将所述多个线性核酸分子与至少一个报道子核酸融合以产生多个报道子构建体,如通过(a)将所述多个核酸分子与线性载体核酸融合,从而产生包括所述核酸分子的多个线性载体;(b)使所述包括所述核酸分子的多个线性载体每一个与包括条形码核酸的引物接触;以及(c)进行聚合酶链反应(pcr),产生包括条形码报道子构建体的多个环状载体;和在足以从包括条形码报道子构建体的所述多个环状载体中去除线性核酸分子的条件下,使包括所述条形码报道子构建体的多个环状载体与核酸外切酶(例如核酸外切酶i和/或核酸外切酶iii)接触。
构建核酸分子报道子文库的组合物和试剂盒
本文预期的是使用本文描述的任何方法产生的核酸分子报道子文库。所述报道子文库可以包括任意数目的报道子构建体。在一些实例中,报道子构建体的数目可以取决于一或多个感兴趣的核酸序列。例如,当核酸分子报道子文库包括来自较大序列例如基因组(例如动物或人基因组、植物基因组、细菌基因组、真菌基因组或古细菌基因组)的核酸分子时,报道子构建体的数目可取决于所述较大序列的大小和/或所述文库的覆盖水平。在一些实例中,报道子构建体的数目为至少约10、25、50、100、250、500、103、104、105、106、107、108或109、如约10-100、100-103、103-104、104-106、106-107、107-108、108-109或106-109或约107-2×107或约2×107(例如1.91×107)。
本文预期的是包括报道子分子和核酸分子(例如插入物)的报道子构建体的文库。使用本文的方法产生的核酸分子报道子文库中的报道子构建体的元件也可以根据预期的鉴别和/或定量方法而变化。例如,使用本文的方法产生的文库可以在体内或体外使用,以及鉴别和/或定量的范围可以从使用基于目测的报道子(例如荧光报道子,例如编码蓝色、紫色、绿色、黄色、橙色或红色荧光蛋白的核酸,如用于基于目测和/或光谱测定的鉴别和/或定量)至基于序列的报道子(例如条形码报道子,例如随机、半随机或非随机条形码,包括至少约5、10、15、20、25、30、35、40、45、50、75、100、250、500、1000、2000、3000或5000个核苷酸长、或约5-10、10-20、15-40、20-30、10-50、10-75、10-100、100-250、250-500、500-1000、1000-3000或1000-5000个核苷酸长、或约20、25、30、15-40或20-30个核苷酸长的核酸或遗传标记,如用于基于阵列和/或基于测序的鉴别和/或定量)。本文预期的是包括多于一个的报道子或报道子类型的文库。在一些实例中,所述文库可以包括基于目测和基于序列的报道子,如包括荧光和条形码报道子的文库。在特定实例中,所述文库包括具有编码gfp的核酸及包括短条形码(例如约25个核苷酸长的条形码)的核酸二者的报道子构建体。报道子构建体的预期插入物的大小也可以根据预期的鉴别和/或定量方法而变化。例如,插入物大小范围是至少约50、100、200、300、400、500、750、800、900、1000、1200、1500、2000、2500或3000个碱基对长,如约50-3000或100-3000个碱基对长,如约50-200、100-200、100-300、300-500、100-1500、500-1200、700-1000或750-850个碱基对长或约800个碱基对长。
本文进一步预期的是包括除报道子分子以外的其它元件的报道子构建体的文库。例如,可以包括报道子核酸的线性衔接子序列或其部分(例如seqidno:1和/或seqidno:2或其部分)。例如,所述报道子构建体还可包括本文所述的任何载体和/或载体元件,如核酸酶切割位点和转录或翻译调节元件,例如启动子(例如基础启动子和/或合成启动子,如超级核心启动子)、增强子、阻抑物和/或聚(a)尾部。
本文还预期的是用于构建核酸分子报道子文库的试剂盒。在一些实例中,所述试剂盒包括一个或多个线性衔接子,例如seqidno:1和/或seqidno:2。在一些实例中,所述试剂盒包括本文所述的任何报道子核酸。例如,可以包括基于目测的核酸报道子(例如荧光报道子,如编码蓝色、紫色、绿色、黄色、橙色或红色荧光蛋白的核酸,如用于基于目测和/或基于光谱测定的鉴别和/或定量)和/或基于序列的报道子(例如条形码报道子,如随机、半随机或非随机条形码,包括至少约5、10、15、20、25、30、35、40、45、50、75、100、250、500、1000、2000、3000或5000个核苷酸长、或约5-10、10-20、15-40、20-30、10-50、10-75、10-100、100-250、250-500、500-1000、1000-3000或1000-5000个核苷酸长、或约20、25、30、15-40或20-30个核苷酸长的核酸或遗传标记,如用于基于阵列和/或基于测序的鉴别和/或定量)。可以考虑一个以上的报道子或报道子类型。例如,所述试剂盒可包括基于目测和基于序列的报道子,如荧光和条形码报道子。在特定实例中,所述试剂盒包括编码gfp的核酸和包括短条形码(例如约25个核苷酸长的条形码)的核酸二者的核酸报道子。
本文进一步预期的是具有报道子构建体的试剂盒,所述报道子构建体包括报道子分子之外的其它元件。例如,可以包括报道子核酸的线性衔接子序列(例如seqidno:1和/或seqidno:2)。所述试剂盒还可包括本文所述的任何载体和/或载体元件,如核酸酶切割位点和转录或翻译调节元件,例如启动子(例如基础启动子和/或合成启动子,如超级核心启动子)、增强子、阻抑物和/或聚(a)尾部。本文还预期的是用于实施本文所述方法的任何酶。例如,所述试剂盒可以包括至少一种连接酶,例如dna连接酶(包括t4dna连接酶,t3dna连接酶,t7dna连接酶,taqdna连接酶(例如taqdna连接酶或高保真taqdna连接酶如hifitaqdna连接酶),热稳定dna连接酶(例如热稳定连接酶,其可催化与互补dna链杂交且无缺口精确配对的两条相邻dna链的5’-磷酸和3’-羟基之间磷酸二酯键的形成,如9°
检测功能性核酸调节元件的方法及所用试剂盒
本文公开的文库可用于多种目的,包括鉴别感兴趣的基因组中顺式调节元件。在一些实例中,本公开的文库可以用于直接测量来自相同物种的不同个体的crm中的功能差异。本公开的文库和方法可以在基于细胞的方法(例如心肌细胞、神经元、肝细胞)中直接测量序列变异的功能结果。在其它实例中,本公开的文库和方法可用于鉴别生物标记crm,如介导药物细胞毒性的crm,维持细胞病理状态的crm和/或维持健康细胞状态的crm。
例如,本公开的文库法可以鉴别应答药物细胞毒性的crm。可以产生检测多种不同细胞毒性作用的生物标记crm的集合,这个生物标记集合可用于在一次筛选中检测药物毒性。本公开的文库和方法还可以鉴别特异于患者衍生细胞(例如ipsc衍生心肌病细胞)中的病理细胞状态特异的crm。本公开的文库和方法还用于鉴别特异于对照细胞(例如ipsc衍生对照心肌细胞)中的健康细胞状态的crm。此外,通过合并所有三种类型生物标记crm,可以在一次筛选中筛选出可将病理细胞状态转变为正常状态而不会引起细胞毒性作用的药物。
在另一个实施方案中,本公开的文库和方法可以筛选具有任何期望活性的人工crm。这些crm可以包括任何细胞类型中选择标记的强大驱动子(例如精确控制工程化细胞(细菌、真菌、植物、古细菌和哺乳动物细胞)中基因表达的驱动子(例如酶))。
在其它实施方案中,本公开的文库和方法可以筛选宿主细胞类型中未表达转录因子的富集基序,例如用以检测各种细胞类型(例如互斥细胞类型,例如从干细胞如胚胎干细胞或诱导型干细胞形成的)中的基因调节相互作用。示例性的应用包括组织工程化,例如以产生特定细胞类型。例如,一种细胞类型可以被抑制而另一种细胞类型可以被促进(例如对于其中一种细胞类型可以转变为另一种细胞类型的应用,例如希望的细胞类型或感兴趣的细胞类型可以转变为不希望的细胞类型或不感兴趣的细胞类型的情况)。
本文公开了检测功能性核酸调节元件(例如crm,如启动子、增强子和/或阻抑物)的方法。在一些实例中,所述方法可以包括用本文公开的核酸分子报道子文库转染至少一种感兴趣的细胞。在一些实例中,所述方法包括选择感兴趣的细胞。可以使用和/或选择任何感兴趣的细胞,如动物细胞(例如哺乳动物细胞)、植物细胞、真菌细胞、细菌细胞或古细菌细胞。在一些实例中,哺乳动物细胞包括干细胞、神经细胞、心血管细胞、肝细胞、内皮细胞、上皮细胞、口腔细胞、生殖系统细胞、内分泌细胞、晶状体细胞、脂肪细胞、分泌细胞、肾细胞、细胞外基质细胞、收缩细胞、免疫细胞、血细胞或生殖细胞中的至少一种。在特定的非限制性实例中,哺乳动物细胞是心肌细胞、神经元、肝细胞、内皮细胞(例如人脐静脉内皮细胞、huvec如在血管生成模型中)、胚胎干细胞、诱导的多能干细胞、hepg2细胞、lncap细胞、hela细胞、hct116细胞或k562细胞中的至少一种。在一些实例中,植物细胞包括分生组织细胞(包括分生组织衍生细胞)、薄壁组织细胞(如叶肉细胞、转移细胞或绿皮组织细胞)、厚角组织细胞、厚壁组织细胞(例如厚壁组织硬化细胞或厚壁组织纤维)、管胞、管状分子、韧皮部细胞(例如筛管、伴细胞、韧皮纤维或韧皮硬化细胞)或表皮细胞(如气孔保卫细胞)中的至少一种。在特定的非限制性实例中,植物细胞是拟南芥属、大麻、玉米、水稻、大麦、小麦、柳枝稷、番茄、马铃薯、衣藻属、水网藻属、水绵属和actebularia中的至少一种。在一些实例中,细菌细胞包括革兰氏阴性或革兰氏阳性细菌细胞中的至少一种,例如酸杆菌属、放线菌属、产水菌属、拟杆菌属、嗜热丝菌属、衣原体属、绿菌属、绿弯菌属、产金菌属、蓝细菌属、脱铁杆菌属、恐球菌-栖热菌属、网团菌属、迷踪菌属、埃希氏菌属、纤维杆菌属、厚壁菌属、梭杆菌属、芽单胞菌属、黏胶球形菌属、硝化螺旋菌属、浮霉菌属、变形菌属、螺旋体属、互养菌属、软壁菌属、热脱硫杆菌属、热袍菌属或疣微菌属细胞。在一些实例中,真菌细胞包括木霉属、链孢霉属、曲霉菌属、红曲霉属、毛霉菌属、酵母菌属、毕赤酵母属或根霉菌属的至少一种。在一些实例中,古细菌细胞包括餐古菌属、caldococcus、ignisphaera、酸叶菌属、acidococcus、气火菌属、除硫球菌属、燃球菌属、葡萄嗜热菌属、stetteria、厌硫球菌属、热盘菌属、热球形菌属、geogemma、超热菌属、热网菌属、火叶菌属、氨氧化古菌(nitrosopumilus(candidatus))、酸菌属、生金球形菌属、憎叶菌属、硫化叶菌属、硫磺球形菌属、热丝菌属、暖枝菌属、热棒菌属、热分支菌属、热变形菌属、火山鬃菌属、aciduliprofundum、古球菌属、铁球状菌属、地球状菌属、适盐菌属、盐碱球菌、haloalcalophilium、盐盒菌属、盐杆菌属、盐棒菌属、盐二型菌属、盐球菌属、极嗜盐菌属、盐几何菌属、盐微菌属、盐惰菌属、盐盘菌属、haloquadra、盐棍菌属、盐红菌属、盐八叠球菌、盐简菌属、盐陆生菌属、halovivax、钠白菌属、钠线菌属、钠杆菌属、钠球菌属、盐碱湖菌属、盐碱红菌属、methanoregula(candidatus)、甲烷砾菌属、甲烷杆菌属、甲烷短杆菌属、甲烷球形菌属、甲烷嗜热杆菌属、甲烷热菌属、甲烷暖球菌属、甲烷炎菌属、产甲烷球菌属、methanothermococcus、甲烷粒菌属、甲烷囊菌属、甲烷泡菌属、产甲烷菌属、甲烷裂叶菌属、甲烷微菌属、甲烷盘菌属、甲烷螺菌科、甲烷螺旋菌属、甲烷鬃菌属、甲烷微球菌属、拟甲烷球菌属、甲烷盐菌属、甲烷嗜盐菌属、甲烷叶菌属、甲烷食甲基菌属、甲烷咸菌属、甲烷八叠球菌属、甲烷火菌属、古老球菌属、焦球菌属、热球菌属、铁原体属、嗜酸菌属、热原体属、初古菌门、纳古菌门或纳古菌属细胞中的至少一种。
在一些实例中,所述方法包括收集至少一种感兴趣的细胞(例如来自至少一个对象)。在一些实例中,从至少两个对象收集细胞,例如至少一个患有疾病或状况的对象和至少一个没有疾病或状况的对象。在其它实例中,在不同条件下(例如在施用试剂或方案如药物或治疗方案之前或之后)从细胞或对象收集细胞。可以使用本文描述的任何文库。所述方法还可以包括测量所述至少一个报道子。在一些实施方案中,所述方法还包括鉴别和/或定量至少一个报道子。在特定实施方案中,鉴别和/或定量至少一个报道子表示存在与该报道子关联的一或多种crm。可以例如通过分离与报道子关联的核酸并对该核酸进行测序来进一步鉴定crm。分离的核酸可以进一步测试以鉴别核酸中包括的crm。
在一些实施方案中,所述方法包括从已经用核酸报道子文库转染的感兴趣的细胞中分离rna,从而产生分离的rna。可以使用任何方法分离rna,包括提取和沉淀方法(例如tanetal.journalofbiomedicine&biotechnology(2009):574398-574398,以其全部内容并入本文作参考)。在一些实例中,可以包括其它步骤,如用以增强分离的rna的纯度。可以包括任何其它rna分离步骤,如将rna与特异于dna的酶接触,例如dnase(如dnasei)和/或核酸外切酶(例如核酸外切酶i和/或核酸外切酶iii)。
在一些实施方案中,鉴别报道子包括合成cdna。在一些实例中,合成cdna包括逆转录分离的rna(例如使用本文所述的任何方法分离的rna),从而产生cdna。可以使用任何逆转录方法。在一些实例中,所述方法包括将分离的rna与至少一种逆转录酶接触。可以使用任何逆转录酶。在一些实例中,可以使用重组莫洛尼鼠白血病病毒(rmomulv)逆转录酶和/或禽成髓细胞瘤病毒(amv)逆转录酶。可以包括任何其它cdna合成步骤。在特定实例中,其它cdna合成步骤包括将rna和至少一种逆转录酶与rna依赖性和dna依赖性的dna聚合酶进一步接触。在一些实例中,其它cdna合成步骤包括加入rnase(例如特异于单链rna的rnase,如rnaseif)。
在一些实施方案中,所述方法包括检测和/或鉴别cdna(例如使用本文所述的任何方法合成的cdna)。可以使用任何检测和/或鉴别cdna的方法(例如基于测序、基于微阵列和/或基于pcr的方法,如下一代测序方法、微阵列和杂交和/或定量pcr)。在一些实例中,cdna包括至少一个独特的条形码报道子。在一些实例中,检测cdna包括扩增cdna(例如使用pcr,如高保真pcr,例如通过使cdna与高保真聚合酶和/或至少一个引物如一对通用引物接触),如条形码报道子cdna(例如条形码报道子cdna)。在特定实例中,扩增cdna包括选择特异于包括至少一个独特核酸条形码的核苷酸的引物(例如至少一个引物,如一对引物,例如一对通用引物)。在一些实例中,所述引物包括一对通用引物,其扩增cdna中的条形码集。在一些实例中,扩增cdna进一步包括使引物与cdna接触并进行pcr(例如使用所述引物和所述cdna)。因此,在一些实例中,所述方法可用于产生扩增的dna(例如cdna),如扩增的条形码dna。在一些实例中,所述方法包括鉴别cdna,如通过鉴别报道子(例如核酸条形码)。在一些实例中,所述方法包括使用基于测序、基于微阵列和/或基于pcr的方法例如下一代测序、微阵列和杂交和/或定量pcr来鉴别核酸条形码。在特定实例中,通过对核酸条形码进行测序(例如使用下一代测序)来鉴别cdna。示例的方法可以进一步包括定量步骤(例如定量所述至少一个独特的核酸条形码)。
在一些实例中,本文描述的方法是高通量方法。在一些实例中,本文所述文库中的多个核酸分子覆盖选择的感兴趣的基因组(例如动物或人基因组)的至少约10%、20%、30%、40%、50%、60%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、98%或100%,或约10-20%、20-40%、25-50%、50-75%、75-85%、80-90%、85-90%、85-100%或90-100%,或约93%、93.4%或94%。在其它实例中,所述文库中的多个核酸可提供大于1×的基因组覆盖(例如1×、1.5×、2×、2.5×、3×、3.5×、4×、4.5×、5×、8×、10×或更大的覆盖)。在一些实例中,所述多个核酸分子包括选择的的感兴趣基因组中顺式调节元件的至少约10%、20%、30%、40%、50%、60%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、98%或100%,或约10-20%、20-40%、25-50%、50-75%、75-85%、80-90%、85-90%、85-100%或90-100%或约85%、90%或95%。
本文进一步预期的是检测功能性核酸调节元件的试剂盒。在一些实例中,所述试剂盒可用于鉴别和/或定量功能性核酸调节元件。在一些实例中,所述试剂盒可用于功能性核酸调节元件的高通量检测、鉴别和/或定量。在一些实例中,所述试剂盒可包括本文所述的任何核酸报道子文库。在某些实例中,所述文库覆盖选择的感兴趣的基因组(例如动物或人基因组)的至少约10%、20%、30%、40%、50%、60%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、98%或100%,或约10-20%、20-40%、25-50%、50-75%、75-85%、80-90%、85-90%、85-100%或90-100%,或约93%、93.4%或94%。在一些实例中,所述文库包括选择的感兴趣的基因组(例如动物或人基因组)中顺式调控元件的至少约10%、20%、30%、40%、50%、60%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、98%或100%,或约10-20%、20-40%、25-50%、50-75%、75-85%、80-90%、85-90%、85-100%或90-100%,或约85%、90%或95%。
在一些实例中,所述试剂盒进一步包括至少一种逆转录酶(例如重组莫洛尼鼠白血病病毒(rmomulv)逆转录酶,禽成髓细胞瘤病毒(amv)逆转录酶)。可以包括其它cdna合成元件,如rna依赖性和dna依赖性dna聚合酶和/或rnase(例如特异于单链rna的rnase,例如rnaseif)。在一些实例中,所述试剂盒包括用于扩增如通过pcr扩增(例如cdna如包括至少一个独特条形码的cdna)的元件。在特定实例中,所述试剂盒包括pcr引物和dna聚合酶(例如高保真dna聚合酶)。
实施例
提供以下实施例以说明某些特定特征和/或实施方案。这些实施例不应被解释为将本公开限制为所描述的特定特征或实施方案。这些实施例描述了用于顺式调节模块(crm)的基因组规模报道子测定方法。gramc可以可靠地测量带有约800bp随机片段化插入物的2亿个hepg2细胞中人基因组近90%的顺式调节活性。生成了一个报道子构建体文库,其覆盖了人基因组约4倍(4×覆盖),具有≥15m的约800bp的随机片段化插入物。
实施例1
这个实施例描述了用于实施例1-7的方法和材料。
gramc文库构建
融合的衔接子制备:gramc制备包括定制设计的融合衔接子,以最小化形成不需要的串联体(图6)。通过integrateddnatechnologies(idt)合成两个互补杂合寡聚物:p-ad4_f(5'-/p/ctgctgaatcactagtgaattattacccrurucaagacactactctccagcagt-3’;seqidno:1)和p-ad4_r(5'-/p/ctgctggagagtagtgtcttgraragggtaataattcactagtgattcagcagt-3’;seqidno:2))。核糖核苷酸位点标记为“ru”和“ra”。通过在1×t4dna连接酶缓冲液(
gramc载体制备:通过用基于pgem-teasy载体
基因组插入物的制备:将20μg的ng16408基因组dna(coriellinstitute)在具有
从短随机寡聚物逐步合成长随机dna序列:由于从头合成大量长随机dna序列仍具有挑战性,因此在某些实例中,从可商购的短随机单链dna(ssdna;图13)产生长随机dna序列集。首先,使用多核苷酸激酶将2μgssdna磷酸化,然后通过随机六聚体、dntps和klenow酶转化为双链dna(dsdna)。平行地,使用随机六聚体、dntps和klenow酶将1μg未磷酸化的ssdna转化为dsdna。其次,用在1×t4dna连接酶缓冲液中的200ng未磷酸化的dsdna和t4dna连接酶制备反应管。将未磷酸化的dsdna与磷酸化的dsdna连接。第三,为起始连接,将50ng磷酸化的dsdna(或未磷酸化的dna的一部分,例如约1/4)加入连接反应管中。由于反应中存在过量的未磷酸化的dna,因此大多数磷酸化的dna与未磷酸化的dna连接。每个未磷酸化的dna分子最多可以接受两个分子的磷酸化的dna(一个末端一个分子)。连接产物包括未磷酸化的5’-末端。重复这个连接程序至少一个循环(例如至少约1、2、3、4、5、6、7、8、9、10、12、15、18、20、25、30、45、50、60、75、90或100个循环,或约1-5、1-10、1-15、1-20、5-20、10-25、25-50或50-100个循环,或约16个循环)。循环数(x)预计为≥2xl/i,其中l和i分别是希望的产生的随机dna的长度和起始核酸的长度。例如,要用100bp长的核酸合成约800bp长的dna分子集,x应约≥16。第四,用dna修复酶(
基因组覆盖估算:为确定代表1×基因组覆盖的衔接子连接的插入物的量,制备0.5ng/μl、0.25ng/μl、0.1ng/μl、0.05ng/μl和0.025ng/μl的插入物稀释液。用两种衔接子特异性引物nj-213和nj-214扩增每种稀释液,在61℃退火及延伸1分钟,通过循环测试确定。使用
文库中存在的基因组区域的泊松概率(p)表示为p=1-(1-p)xn,其中p=(插入物大小)/(基因组大小),n=对于指定插入物大小的基因组分区数,x=预期的基因组覆盖。将通过qpcr鉴别存在的靶比例与p值进行比较。基于此模型,对于约1×基因组覆盖的样品的p约为0.6。测试0.1ng/μl稀释液对11个靶中的6个呈阳性或比例为0.545,表示0.5×-1×覆盖。因此,确定0.2ng插入物代表约1×基因组覆盖。混合等摩尔量的独立扩增的重复样品以获得5×基因组覆盖的插入物集合。
gramc文库的插入物克隆和n25条形码编码:将30ng的5×基因组插入物克隆进在16μl
为在gfporf下游加入n25条形码,在50μl的q5高保真dna聚合酶反应中使用150ng文库用于用nj-127的单次pcr循环,其包含随机25bp条形码序列,核心聚(a)信号(nag,etal.rna12.8(2006):1534-1544)和5’生物素酰化,退火温度为60℃,持续40秒,延伸时间为15分钟。nj-126在pcr中用作竞争剂,以通过占据和延伸相反链来降低模板转换的可能性。如前所述,通过使用50μl珠进行
分离后,将c1珠在20μl水中洗涤,然后重悬在50μl水中。将一半的条形码编码的文库在24×20μl重复
然后将条形码编码的gramc文库进行自连接。为减少分子间连接,将125ng条形码编码的文库在600μl的1×t4连接酶缓冲液(
gramc文库的转化和大小估算:为确定电穿孔的规模,将1μl连接产物电穿孔到25μl的
为产生具有200m集落靶的完整gramc文库,每2×25μl
作为质量控制步骤,从平板中挑选12个集落,并提取质粒以使用sanger测序检查插入物大小和条形码。每个集落的质粒应含有一个插入物(约800bp)和一个条形码。在连接产物包括高条形码多样性的情况中,从集落中鉴别的条形码序列不应出现在最终文库中。表3中提供了使用的gramc载体和寡聚物的示例序列。
表3:引物和衔接子修剪序列示例
通过
测序文库:为鉴别单个报道子构建体中的插入物和相关条形码,使用nextseq500平台进行双末端(paired-end)测序。在
在这个实施例中,构建测序文库从使用针对载体主链或gfporf的sgrna用cas9(
为从环化的第一轮pcr产物中扩增插入物::n25盒,使用nj-209和nj141扩增4个包含2ng的hs800_14连接物的重复样品(以下称为hs800_1423),及使用nj-208和nj142扩增4个包含2ng的hs800_23连接物的重复样品(以下称为hs800_2314),退火温度60℃,延伸时间90秒,共8个循环。将产物进行柱清洗,凝胶分离和珠清洁,以用于随后的pcr扩增,以加入用于
为增加在
从插入物和条形码修剪衔接子序列:从每对序列读取提取插入物的5’和3’末端及其相关的n25条形码。使用trimmomatic(bolger,etal.bioinformatics30.15(2014):2114-2120)去除衔接子序列,及使用seqtk(github.com)以反向互补序列。为提取插入物的5’-末端和3’-末端,分别修剪了p1和p2衔接子。为提取n25条形码,根据序列读取方向,首先修剪p3或p4衔接子,对修剪的序列进行反向互补,再修剪p4或p3衔接子。未能修剪任何衔接子序列的双末端读取被放弃。注意,对于n25条形码序列,每个衔接子保留1bp,获得27bp读取。表3中提供了用于修剪的衔接子序列。
人基因组中插入物的序列读取作图和鉴别:为鉴别插入物,将提取的插入物的5’和3’末端作图于grch38/hg38组件上(从genome.ucsc.edu下载)。使用burrows-wheeler比对工具(bwa)(li,etal.bioinformatics25.14(2009):1754-1760)通过以下命令对序列作图:“bwamem-w1500”。跨越>1,500bp或<300bp的作图的成对读取被放弃。当两个作图的插入物重叠时,其中点在20bp范围内,两端在50bp范围内,将其合并为一个插入物,并采用最大化其长度的坐标。
聚类n25条形码:为鉴别同一条形码的读取,基于以下步骤对提取的条形码读取聚类:i)使用khmer软件包(crusoe,etal.f1000research4(2015))及如下命令通过过滤冗余读取生成代表性读取:“normalize-by-median.py-c1-k25-n5-x2.5e9”;及ii)使用bwa软件(li,etal.bioinformatics25.14(2009):1754-1760)及如下命令将整组条形码读取与代表性读取进行匹配:“bwaaln-n2-o2-e-1-m3-o11-e8-k1-l6”。将与任何代表性读取均不匹配的条形码读取加入代表性读取文件中,并重复进行bwa检索。通过单连锁聚类法鉴别相同条形码的读取,并且为每个聚类指派独特的条形码聚类(bcl)编号。生成具有bcl编号的代表性读取新文件,用于将来使用(见下文,hepg2中的gramc测定:将条形码读取与条形码聚类匹配)。
将基因组插入物与条形码聚类(bcls)相关联:尽管在双末端读取中每个条形码读取均固有地与来自插入物的读取相关联,但一小部分bcl与一个以上已鉴别的基因组插入物相关联。这种歧义的主要原因是基因组中高度相似的重复区域所致。bcl的指派被强制用于具有最大bcl读取的插入物。如果≥2个插入物对bcl具有相同读取编号,则不会将bcl指派给任何插入物。
hepg2中gramc测定
细胞培养:将hepg2细胞(atcchb-8065)在供应商推荐条件下在补加不含抗生素的10%胎牛血清的emem中生长。自所有实验接收之日起,使用不超过16代内的hepg2细胞。所有实验均在融化后经历最低5代传代的细胞中进行,因为在<5代传代的细胞中与在≥5代传代的细胞中的报道子表达不同。
基因组规模转染和裂解物收集:对于每批基因组规模转染批次,将107个细胞接种于在10×150mm培养皿中的30ml培养基中(100m细胞),使其附着30小时。根据制造商的方案,用100μg的hs800_gramc文库转染细胞,使用在2×2ml硅化管中制备的在4ml的
为了收集,转染后将细胞用1×pbs洗涤26小时并通过在2.4mlrna-stat-60
rna制备和cdna合成:这个方案着重于两个参数:i)全面去除rna样品中污染的dna,以及ii)大量(约4mg)总rna逆转录(rt)的效率最大化。为dnasei补充核酸外切酶i和iii的混合物可以完全去除双链和单链污染dna,因为dnasei对单链dna的效率较低。为经济高效地最大化rt,使用的rna是制造商建议的最大输入rna的15倍,而又不影响rt反应中的cdna产量。这个方案示意图在图9中示出。
为去除污染的dna,将分离的总rna(约4mg)重悬于1.7ml无核酸酶的水中,在37℃在包含1×dnasei缓冲液、100udnasei(
作为逆转录(rt)的质量控制,将含有约4000个细胞(约1μg)的等效体积的总rna用于cdna合成,根据制造商的方案使用高容量cdna逆转录试剂盒(applied
将剩余的总rna(约4mg)稀释至1.420ml,加入2000pmol的gramc_rt_oligo(nj-489)。将rna/引物混合物在65℃温育1分钟,然后在冰上冷却,随后加入200μl的10×高容量缓冲液、80μl的10mmdntp和100μl的multiscribe,不使用随机寡聚物。将反应在室温温育10分钟,然后在37℃4小时。与使用100个细胞/孔等效体积的标准rt对照相比,通过针对gfp的qpcr监测基因组规模cdna合成的进程。使反应继续进行直至ct值变得类似于标准rt反应。如果需要,将反应用m-mulv逆转录酶(
rt反应完成后,将样品用乙醇沉淀以减少体积。将rna/cdna重悬并用1000u的rnaseif(
制备用于ngs的表达的n25条形码:使用引物nj-141和nj-142在8次重复的50μl的
制备测序文库以进行
将条形码读取与条形码聚类(bcls)匹配:这个步骤的目的是针对每个条形码聚类(bcl)计数从表达的条形码或输入文库中读取的条形码数目。通过使用与上述相同的命令进行bwa搜索,将衔接子修剪的条形码读取与上述建立的代表性条形码读取匹配。当一个条形码读取匹配多于一个bcl时,每个匹配都计入相应bcl。因为对表达的条形码和输入文库都应用了相同的程序,所以中和了多次计数条形码读取的影响。
crm活性的计算:这个步骤基于从表达的条形码和输入文库计数的每个bcl的读取数目,计算每个插入物的顺式调节活性。当一个插入物与≥2个bcl相关联(99%的插入物)时,合并所述插入物的所有bcl的读取计数。首先,为避免由于输入计数过低而导致的假阳性crm,对于这两个批次的实验,保留了来自输入文库的≥10计数或表达的条形码≥50计数的插入物。这个过滤获得9,339,996个符合保留标准的插入物。其次,将表达的条形码的读取计数除以输入文库的读取计数,然后对所得数字进行排序。中间30%的数据用于计算背景活性(bg)(例如26)。将crm活性根据背景活性进一步标准化。当至少一个批次显示≥5×bg且另一个显示≥4.5×bg(90%的5×bg)时,插入物被认为是crm。总共鉴别出54,115个通过标准的插入物。在去除基因组其它部分中具有≥95%相同序列的插入物及合并重叠crm之后,最终一组包含41,216个独特且不重叠crm。散点图在图2a中示出,其是使用r软件包(cran.r-project.org)中ggplot2(wickham.ggplot2:elegantgraphicsfordataanalysis,springer-verlagnewyork,2009)产生的,使用500,000个随机选择的插入物。
crm的基因组分布
为比较crm和基因的基因组位置,使用来自ftp.ensembl.org的可公开获得的基因注释文件“grch38.89.gff3”和来自encodeproject.org的hepg2细胞“encff861gcr和encff640zbj”的rna-seq数据。在两个rna-seq数据中fpkm≥1的基因均被视为“表达”。为生成图2c和10a-10f中所示的图,使用r中的gridgraphicspackage(murrell.rgraphics.crcpress,2016),仓大小为1mb。
为计算基因组区域中关于基因的crm富集(图2d),将跨越超过2kb窗口的插入物/crm分配给与插入物最多重叠的窗口。从grch38.89.gff3文件中提取基因5’末端和3’末端的基因组坐标。一个基因的插入物/crm仅计数一次,但对于不同的基因则允许多次计数。
逐个报道子测定以进行验证
产生单个报道子构建体:通过pcr单独扩增20个基因组区域(11个crm,5个边缘活性区域和4个非活性区域)并通过gibson
将预先条形编码的scp-gramc载体进一步用于产生egfp内部对照载体,用于各个克隆的gfp报道子表达的qpcr。对于这个步骤,使用nj731和nj732通过反向pcr扩增载体。来自pegfp-c1的egfporf使用nj729和nj730扩增并使用gibson
单独报道子测定以验证gramc结果:将hepg2细胞以约60k个细胞/孔接种在24孔平板中的补充有10%fbs的500μlemem中。为与基因组规模测定一致,使用的细胞在从atcc收到第12-15代之间,在恢复后至少7代。使细胞附着24小时并用50μl的
在crm相对无活性插入物中encode注释的相对富集
encodechip-seq文件得自encodeproject.org。使用bedtools(quinlan,etal.bioinformatics26.6(2010):841-842)与命令“bedtoolsjaccard-f1e-09-f1e-09”计算crm和各个encode数据之间重叠。通过以下程序计算crm中encode注释的相对富集。i)首先,计算crm和encode注释之间重叠碱基对的基因组比例。ii)通过将两个数据集的基因组比例相乘来计算随机预期的重叠。iii)将i)的结果除以ii)的结果以计算富集。iv)按照相同程序,计算非活性区域(l1组)中相同encode注释的富集。v)通过获得iii和iv的比率来计算相对富集。
crm和预测的强增强子中的基序富集
gramc插入物的选择:将chromhmm(ernst,etal.nature473.7345(2011):43;ernst,etal.naturebiotechnology28.8(2010):817)预测的hepg2的强增强子与gramc数据比较crm活性和基序富集。染色质状态的基因组坐标通过liftover(hinrichs,etal.nucleicacidsresearch34.suppl_1(2006):d590-d598)转变为hg38。首先,随机选择与预测的强增强子长度≥90%重叠的非重叠gramc插入物。这种选择产生了18,898个gramc插入物,其对应于预测的强增强子。这个用于生成图3a。
为比较基序富集,在不考虑预测的增强子的情况下,随机采样了另外18,898个不重叠的gramccrm(≥5×bg或g5)。作为阴性对照,还采样了37,796个不重叠无活性插入物(≤1×bg或l1)。
基序富集测量:为测量推定的转录因子结合位点(tfbs)基序,同时分析了采样的75,592个插入物。使用hocomocov10数据库(kulakovskiy,etal.nucleicacidsresearch44.d1(2015):d116-d125)和fimo软件(cuellar-partida,etal.bioinformatics28.1(2011):56-62;bailey,etal.nucleicacidsresearch37(2009):w202-w208),e值截止值为1e-5。每个基序的丰度是给定集合中包含基序的插入物的比例。相对基序富集是通过将crm或预测的增强子中基序的丰度除以阴性对照集合中相同基序的丰度来计算的。
crm中基序富集和chip-seq峰的比较:以名称鉴别hocomocov10与encodechip-seq数据之间的58个常见转录因子。计算的相对富集评分用于生成图4b。
测量基因异位表达对crm的影响
gramc文库随机子集的制备:为获得gramc文库的小规模子集以通过pitx2或ikzf1的异位表达进行扰动实验,将约50μl冷冻甘油原液在2ml的lb培养基中稀释,在37℃以250rpm回转式振荡20分钟以回收。制备一系列2倍稀释液,其中1/100用于2个10倍稀释液进行铺板和集落计数,剩余的每个2倍稀释液用于接种150ml的lb-amp培养液过夜生长。使用
80k构建体文库的扰动测定:将细胞以约2m个细胞/10cm2平板一式两份种植,以进行以下3种共转染中的每种转染:80k文库+cmv::pitx2(genscriptohu17480d),80k文库+cmv::ikzf1(genscriptohu28016d)及80k文库+cmv::egfp(clontechpegfp-c1)。转染前将细胞培养约24小时。将细胞与9μg的80k文库和3μg各自的表达载体共转染,使用用于hepg2试剂的36μl的
转染后24小时,通过胰蛋白酶消化收集细胞并用1×dpbs洗涤。保存1/10的细胞用于western印迹分析,以确认pitx2和ikzf1的表达。裂解剩余细胞,针对dna和rna使用带有iiicg柱的zymo-duet试剂盒处理,无需柱上dnasei处理。在100μl中洗脱dna,在80μl中洗脱rna,并在37℃用dnasei(8u)/exoi(100u)/exoiii(100u)处理最少4小时,总反应体积为在1×dnasei缓冲液中100μl。假设每个样品有约10m个细胞,使用以gfp为靶的qpcr检测等价的约10,000个细胞的gdna和约5000个细胞的核酸酶处理的rna,以分别确认转染质量和rna中dna去除的完成。根据需要用另外2u的dnasei加标反应。使用zymo-iiic柱清洁rna,在50μl水中洗脱。如基因组规模方案中所述,将等价的约4000个细胞在标准rt反应中用作质量控制的量度。剩余rna与用于cdna合成的80pmole的gramc_rt_oligo(nj-489)在80μl的1×高容量cdna合成反应中保温,使用8μl的multiscribe和3.2μl的dntp但不使用随机引物在37℃温育4小时至过夜,在室温2小时后,用于质量控制qpcr。dna消化完成后,在37℃将4μl的
如上所述,初步扩增n25条形码,但是使用6个循环的单个50μl的
通过western印迹确认异位转录因子表达:将每种转染条件(80k文库+cmv::pitx2,80k文库+cmv::ikzf1,及80k文库+cmv::egfp)的等分试样在冰上在80μl的ripa缓冲液(150mmnacl,1%np40,0.5%脱氧胆酸钠,0.1%sds,50mmtris-hclph8.0,5mmedta)中间歇性轻弹裂解30分钟,缓冲液用1:100稀释的halt蛋白酶抑制剂混合物
将约25ng的每种样品一式两份加样(表达和对照),在12%聚丙烯酰胺凝胶上分离,移至pvdf膜上,并用抗flag抗体(1:500,santacruzsc-166355)或抗gapdh抗体(1:1000,santacruzsc-25778)印迹。辣根过氧化物酶缀合的二抗(1:5000)和增强的化学发光试剂(gehealthcare)用于在bio-radchemidocmp系统上检测条带。
实施例2
本实施例描述了gramc文库的构建。在这个实施例中,通过以下程序生成gramc文库(图1a-1d)。首先,对随机基因组dna片段进行大小选择,衔接子连接,然后连续稀释以达到预期的基因组覆盖(图1a)。为改良衔接子连接的准确性,将衔接子(图6)融合以形成环状连接产物,其可以抵抗针对线性dna的核酸外切酶i/iii处理,包括未连接的dna和线性串联体。核酸外切酶处理后,通过rnasehii使环状连接产物线性化,其切割在融合的衔接子内的核糖核苷酸位点(uu/aa)。然后将线性化的连接物连续稀释并使用衔接子特异性引物进行pcr扩增。通过qpcr计数11个随机选择的基因组区域的存在或不存在来鉴别预期的基因组覆盖的稀释度。对于包含约4m个随机取样的约800bp长的基因组dna片段(平均1×基因组覆盖)的稀释液,预期靶区域存在率为0.6。将5×稀释液(或任何所需的基因组覆盖)与两个常见dna组件组装在一起以形成线性dna产物文库,其中包含基因组测试片段、基础启动子、gfporf(arnone,etal.development124.22(1997):4649-4659)和载体主链(图7)。载体系统使用全部两个对称超级核心启动子(pan-bilateriansupercorepromoter)1(scp)(juven-gershon,etal.developmentalbiology339.2(2010):225-229)。
其次,使用一对可以扩增包括载体主链的整个文库的通用引物通过pcr,用过量的随机25mer(n25)对所得基因组dna文库进行条形编码(图1b)。常见的引物之一,即引物_r,含有在中间的一个随机n25和一个核心聚腺苷酸化信号(polya)(nag,etal.rna12.8(2006):1534-1544)。将条形编码的文库自连接,核酸外切酶i/iii处理,并电穿孔进大肠杆菌中以进行文库扩增和质粒提取。一小部分(例如1/1,000)未回收的转化体用于测量集落形成单位(cfu),其余用于在液体培养中的文库扩增和随后的质粒提取。由于pcr介导的条形编码导入过多的条形码,因此实际上所有单个转化体都含有独特的条形码。例如,在最终文库中未鉴别出用于集落计数的存在于转化体中的条形码。gramc文库中独特条形码报道子的数目可以通过电穿孔的规模来控制。在本文使用的方案中,具有约800bp的插入物的4-10ng环状连接产物始终产生约40mcfu,这与可商购的感受态细胞的广告效率相当。只要收获的独特条形码的数目远大于独特插入物的数目,就可以维持第一步中确定的文库的基因组覆盖。纯化的质粒用于文库鉴定。文库鉴定包括通过
使用所述方法,产生了约800bp长的插入物的人gramc文库。这个文库中独特基因组dna插入物和独特条形码的预期数目分别为20m(5×基因组覆盖)和200m(10个条形码/插入物)。在分析了作图为hg38组装的479.1m个成对序列后(在519m双末端读取中),鉴别出15.6m个基因组区域。与这些基因组区域相关的独特条形码的总数为191m。所述文库至少一次覆盖93.4%的人基因组(表1)。
表1:人gramc文库的基因组覆盖
尽管获得更多的测序读取将改善这些数目,但这些数目已经接近文库中插入物和条形码的预期数目。在检测的15.6m个基因组区域中,有13.8m个插入物是序列独特的(与其它基因组区域的序列相同性<95%)。另外,独特插入物的基因组分布或多或少是均匀的(图2c)。对于独特插入物(图1c),71%的插入物在750-850bp范围内,表明大小选择是有效的。此外,考虑到每个插入物的条形码数目(图1d),尽管大多数插入物的条形码数目与预期的数目10有明显偏差,但是99%和55%的独特插入物分别与≥2个条形码和≥10个条形码相关联。因此,在gramc文库中,条形码对报道子表达的特异性作用不明显。插入物及其相关条形码的基因组坐标的列表可从图6中获得。
实施例3
在这个实施例中,描述了gramc在hepg2细胞中的应用。将gramc文库在两个批次中测试:在种植时100mhepg2细胞或在转染时200m细胞。作为比较,先前基因组规模增强子筛选使用了300mlncap细胞(liu,etal.genomebiology18.1(2017):219)和800m的hela细胞(muerdter,etal.naturemethods15.2(2018):141),基因组规模启动子筛选使用了100m的k562细胞(vanarensbergen,etal.naturebiotechnology35.2(2017):145)。将gramc文库转染进细胞后,提取总rna并逆转录,对表达的条形码进行pcr扩增。为避免在二次富集mrna(muerdter,etal.naturemethods15.2(2018):141)或报道子转录本(tewhey,etal.cell165.6(2016):1519-1529)期间损失报道子转录,将总rna和gramc特异性寡聚物用于逆转录。通过pcr扩增表达的条形码,并通过
从每批表达的条形码中获得约200m读取,78-79%的条形码与具有相关基因组区域的条形码匹配。为说明拷贝数变化,从输入质粒中获得约450m条形码读取。由于99%的插入物驱动≥2个条形码,因此将同一插入物的多个条形码的读取合并在一起。从输入质粒中≥10个读取的约7.5m插入物用于数据分析。在两个独立的实验中,来自41,216个非重叠基因组区域的共50,993个插入物显示的活性比背景(bg)活性(红色点,≥5×bg)高≥5倍(图2a)。重复gramc数据显示pearson相关系数(r)为0.95,一个批次中的crm被认为是另一批次中的crm的可能性为0.80(80%的crm再现性)。当截止值降低至背景的3倍时(橙色和红色点,≥3×bg)时,活性区域的数目增加至150,011(crm的再现性62%)。
为验证gramc的准确性,随机选择11个crm(≥5×bg,红点)、5个边缘活性片段(3-5×bg,橙色点)和4个非活性片段(≤1×bg,黑色点),使用逐一报道子测定单独检测其调节活性(图2b)。通过qpcr测量相对于转染dna拷贝的gfp转录物水平。将报道子表达根据背景活性(bg)进一步标准化,这是4个非活性报道子构建体的平均水平。以黑色条示出各个插入物的4个独立测定的平均水平。在测试的11个crm中,8个插入物≥5×bg,而2个插入物和1个插入物分别为2.8×bg和1.9×bg。这个结果与gramc中crm的80%再现性相当(图2a)。对于5个边缘活性插入物,1个插入物为10×bg,3个插入物在预期的3-5×bg范围内,1个插入物为1.4×bg。总体而言,通过gramc测量的顺式调节活性在独立测定中可再现(r2=0.83)。这些结果表明,gramc是在基因组规模发现crm的可靠而有效的工具。
实施例4
这个实施例描述了具有预期crm特征的gramc鉴别的crm。由于gramc基于报道子构建体的标准构型,因此gramc鉴别的crm应具有通过传统的报道子测定法鉴别的crm的已知特征。首先,crm应主要位于hepg2中表达的基因附近。比较了hepg2、crm和输入文库中表达基因的基因组位置,表达的基因和crm具有相似的模式,而输入文库大致均匀分布(图2c和10a-10f)。
其次,已知crm富集在基因5’近端(启动子);但是其大多数位于近端区域之外(远端增强子)(26)。当针对在表达基因的上游或下游的滑动2kb窗口内测试的插入物的数目计算crm的比例时,5’近端2kb区域显示最高的富集(0.03)(图2d)。3’近端2kb区域显示第二最高峰,而基因组区域中的crm略微耗尽。尽管存在这些区域差异,但与0.0067的基因组平均值相比,crm在每个方向上一致富集在至少100kb区域内的表达基因周围。在未表达的基因附近也观察到类似模式,但是富集程度低于表达的基因附近。这些结果表明,gramc可以有效地鉴别近端启动子和远端增强子二者。
第三,预期crm与转录因子和其它积极影响crm功能的蛋白质结合相关。窄峰的相对富集(相对于随机预期共享的总碱基对)是根据来自crm相对于非活性片段中hepg2的167个encodechip-seq或dnase-seq数据计算得出的(图2e),有153个数据显示在crm相对于非活性区域≥2倍富集。这些包括一般转录因子(例如gtf2f1,taf1和tbp)、转录共激活因子(p300)和组蛋白修饰酶(例如h3k4me3和h3k9ac)。在crm中未富集或甚至耗尽的chip-seq峰包括转录因子(tcf12和bclaf1)、剪接体组分(plrg1和snrnp70)和组蛋白甲基化酶(h3k27me3,h3k36me3和h3k9me3)。有趣的是,尽管总体富集,但只有32%的gramc鉴别的crm与在crm中≥2倍富集的153个encode数据重叠,而58%的crm不与本分析中使用的任何encode数据重叠。尽管获得更多转录因子的chip-seq数据可能会增加重叠,但报道子测定可检测由于染色质沉默而在基因组中无活性的crm或可逃避chip-seq检测的crm。
实施例5
在这个实施例中,示出基序富集以解释chromhmm预测的增强子的差异活性。较早研究表明,尽管基于染色质标记的crm预测在经过功能验证的crm中富集,但大多数预测的crm在报道子测定中均未驱动显著表达(liu,etal.genomebiology18.1(2017):219;muerdter,etal.naturemethods15.2(2018):141;vanarensbergen,etal.naturebiotechnology35.2(2017):145)。与这些观察结果一致,在与在hepg2中chromhmm预测的强增强子重叠≥90%的gramc测试的片段的顺式调节活性测定中(ernst,etal.naturemethods9.3(2012):215),约80%预测的增强子显示出在gramc中背景活性的≤2倍(图3a)。如果预测的增强子是真正的增强子,则可以预期转录因子结合位点(tfbs)基序的富集。预测的强增强子是本文的重点,因为启动子固有地富含基序,而预测的弱增强子可能增加歧义。
使用fimo软件(cuellar-partida,etal.bioinformatics28.1(2011):56-62;bailey,etal.nucleicacidsresearch37(2009):w202-w208)比较在预测的增强子、gramc鉴别的crm和无活性片段中601个hocomoco_v10人基序的富集(kulakovskiy,etal.nucleicacidsresearch44.d1(2015):d116-d125)。总体而言,gramc鉴别的crm比预测的增强子表现出更强的基序富集(图3b)。在gramc中(图3c-3d)活性或边缘活性的预测增强子显示出与gramc鉴别的crm相当的基序富集或耗竭。相反,在具有较弱报道子表达的预测增强子中,基序的富集逐渐消失(图3e-3g)。由于其无法驱动显著报道子表达和弱基序富集,因此大多数预测的增强子可能不是真正的增强子。但是,这并不排除染色质标记可能指示增强子的邻域而不是确切位置的可能性,以及预测的增强子可能具有在报道子测定中无法测量的其它类型的顺式调节活性。
干扰素途径的激活导致在dna转染时错误鉴别干扰素反应性增强子(muerdter,etal.naturemethods15.2(2018):141),并且这种假象会减少gramc鉴别的crm和chromhmm预测之间的重叠。但是,与hepg2细胞不激活该途径的最初发现一致,干扰素刺激的转录因子包括irf1-9和hmx1的基序在gramc鉴别的crm中不富集。
实施例6
这个实施例表明,crm中富集的基序可以预测潜在的新型基因调节相互作用。通过小报道子构建体测量的报道子表达模式是宿主细胞中反式调节环境的直接解读。由于crm的dna序列包含转录因子的结合位点,因此通常使用计算基序分析来推断基因调节程序(例如xie,etal.nature434.7031(2005):338;mariani,etal.cellsystems5.3(2017):187-201;enuameh,etal.genomeresearch(2013):gr-151472;markstein,etal.development131.10(2004):2387-2394;halfon,etal.bmcgenomics12.1(2011):578)。基于通过fimo在crm和在无活性片段(阴性对照)中计算预测的601个hocomoco_v10human基序(kulakovskiy,etal.nucleicacidsresearch44.d1(2015):d116-d125),计算丰度(基序阳性crm或无活性片段的比例)和基序的相对富集(crm相对无活性片段中的基序的相对丰度)(图4a)。结果表明,与无活性片段相比,601个基序中有176个基序在crm中≥2倍富集。有趣的是,大多数(65%)富集的基序是针对表达的(fpkm≥1)转录因子,而其余是针对未表达或表达极低(fpkm<1)的转录因子(3)。
表达的转录因子的富集基序应预测在hepg2中鉴别的crm的阳性调节因子。为检测调节因子,将基序分析结果与来自hepg2细胞的encodechip-seq数据比较(3)。如果基于基序富集的预测转录因子是正确的,则相同转录因子的chip-seq峰也应富集。两个数据集共有总计58个转录因子。在58个因子中,相对无活性片段,31个基序和56个chip-seq峰在crm中富集≥2倍(图4b)。假定除了一个富集的基序以外,所有其它基序也都在chip-seq数据中富集,则基于基序富集的阳性调节因子预测具有非常低的假阳性率(<<0.1)。其它约50%的转录因子显示基序富集<2倍,但chip-seq峰仍然高度富集。尽管需要进行更详细的分析,但在保守的情况下,本文基于基序的预测显示出约0.5的假阴性率。
未表达的转录因子的基序富集表明其在其它细胞类型或条件下将hepg2-crm作为激活物或阻抑物来控制(图4c)。候选转录因子在hepg2中的异位表达被用于检测这种调节因子。检验两个转录因子基因pitx2(同源盒基因)和ikzf1(ikaros同源物)。在小鼠中,pitx2在胎肝中表达并且是胎肝的造血功能所必需的,而pitx2和胎肝的造血功能的关闭对于从胎肝分化成年肝脏至关重要(kieusseian,etal.blood107.2(2006):492-500)。类似地,ikzf1是造血发育的关键调节物(davis.therapeuticadvancesinhematology2.6(2011):359-368)并在胎肝中表达(roy,etal.pnasusa(2012):201211405);但是其在肝发育中的功能尚不清楚。将可以组成型表达pitx2(cmv::pitx2)或ikzf1(cmv::ikzf1)的mrna的质粒与来自完整gramc文库中的一组随机选择的约80,000个gramc报道子构建体共转染。作为对照实验,将可组成型表达gfpmrna(cmv::gfp)的质粒与同一组报道子构建体共转染。所有这三个实验的重复实验都是高度可再现的(pearson'sr≥0.99)(图14)。hepg2中pitx2的异位表达下调大多数crm≥2倍,这种下调在pitx2基序阳性crm中更加明显(双样本t检验,p=4.4e-16)(图4d)。在ikzf1的情况下,只有9个crm下调≥2倍,9个下调的crm中有6个对ikzf1基序呈阳性(双样本t检验,p=2.5e-4)(图4e)。通过western印迹证实了这两个重组基因的蛋白表达(图11)。这些结果表明,pitx2(和ikzf1在较小程度上)在胎肝中维持hepg2-crm抑制,而pitx2清除对于成年肝脏中hepg2-crm的激活和基因表达至关重要。这些结果表明,crm不仅可用于预测宿主细胞中的调节程序,而且可用于预测在时间和空间上分离的细胞之间的调节相互作用。
实施例7
这个实施例表明sine/alu元件在crm中富集。真核基因调节的早期模型提出重复元件是基因表达控制的关键参与者(mcclintock.pnasusa36.6(1950):344-355;britten,etal.science165.3891(1969):349-357)。这些预测随后得到有助于基因调节及其进化的alu和erv元件的多个实例支持(britten.pnasusa93.18(1996):9374-9377)。此外,染色质特征的基因组调查表明,sine/alu元件在推定的crm中富集(su,etal.cellreports7.2(2014):376-385;trizzino,etal.bmcgenomics19.1(2018):468)。但是,针对增强子(muerdter,etal.naturemethods15.2(2018):141)或启动子(vanarensbergen,etal.naturebiotechnology35.2(2017):145)的基因组规模报道子测定已检测到ltr/erv1和ltr/ervl-malr在crm中富集而不是sine/alu。为测定在gramc鉴别的crm中的这种富集,将本文的数据与人基因组中带注释的重复元件进行比较(smit,etal."repeatmaskeropen-4.0”(2015))。检测到三个重复元件家族,即随体/端粒、sine/alu和ltr/erv1,在crm中富集≥2倍(图5a中g5组);但是ltr/ervl-malr在crm中不富集。这三个元件在边缘活性g3l4和g4l5组中也较低程度富集。有趣的是,crm中的α随体耗竭了约8倍,表明其在hepg2中具有抑制功能或与其它crm的不相容性。然而,未检测到预期在肝脏中是转录阻抑物的逆转录子/sva元件的耗竭(trizzino.genomeresearch27.10(2017):1623-1633)。
使用gramc鉴别的crm,测定alu元件向增强子的进化与时间的关系(su,etal.cellreports7.2(2014):376-385)。crm中alu元件的富集应与年龄正相关。但是,检验了alu的三个主要亚家族(图5b),最年轻的亚家族(aluy)和中间亚家族(alus)示出在crm中≥3倍富集,而最老的亚家族(aluj)仅示出中度富集(1.3倍)。因为最初的研究基于hela细胞中的染色质注释,所以这种差异可以用细胞类型的差异来解释。因此,汇编了在hela细胞中用萤光素酶测定法测试的19个alu元件的亚家族(su,etal.cellreports7.2(2014):376-385)。与这些结果一致,8/10的aluy或alus元件是活性的,而仅4/9的aluj元件是活性的。因此,结果与可替换模型一致,即alu元件随着年龄增长而失去调节活性。
这些结果表明,gramc数据可用于测试多种进化基因组学假说,并且与早期基因组规模报道子测定或染色质注释所产生的数据相比,其可以得出不同结论。此外,有可能在gramc和先前报道子测定之间观察到的差异可能在很大程度上归因于所使用的不同细胞类型。表2提供了重复元件的完整列表的富集。
表2:重复元件的完整列表的富集
注意:富集评分在log2量度
鉴于可以将本公开的原理应用于其的许多可能的实施方案,应当认识到示例的实施方案仅是举例,不应被视为限制本发明的范围。本发明的范围是由所附权利要求书限定。因此,我们要求保护在这些权利要求的范围和精神内的所有内容作为我们的发明。
序列表
<110>罗格斯新泽西州立大学
<120>gramc:顺式调节模块的基因组规模报道子测定方法
<130>7213-101448-02
<150>62/753,608
<151>2018-10-31
<160>124
<170>patentinversion3.5
<210>1
<211>52
<212>dna
<213>人工序列
<220>
<223>示例线性衔接子序列
<400>1
ctgctgaatcactagtgaattattacccuucaagacactactctccagcagt52
<210>2
<211>52
<212>dna
<213>人工序列
<220>
<223>示例线性衔接子序列
<220>
<221>misc_rna
<222>(24)..(25)
<400>2
ctgctggagagtagtgtcttgaagggtaataattcactagtgattcagcagt52
<210>3
<211>41
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-95,gibson_scp1_amp1
<400>3
ctgctggagagtagtgtcttgtacttatataagggggtggg41
<210>4
<211>26
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-96,ecop15l_p1r_lin
<400>4
ctgctgaatcactagtgaattcgcgg26
<210>5
<211>19
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-101,3pofn25mershort
<400>5
ggcgcgccgctgagggagt19
<210>6
<211>23
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-126,nt_del_f
<400>6
aattcgccctatagtgagtcgta23
<210>7
<211>71
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-127,bn25_polya_r-1/primer_r
<220>
<221>misc_feature
<222>(1)..(1)
<223>5'生物素修饰
<220>
<221>misc_feature
<222>(22)..(46)
<223>nisa,c,g,toru
<400>7
tacagtccgacgatccagcagnnnnnnnnnnnnnnnnnnnnnnnnnggcgcgccgctgag60
ggagtctagag71
<210>8
<211>66
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-128,pn25_polya_r-2
<220>
<221>misc_feature
<222>(1)..(1)
<223>5'磷酸化修饰
<400>8
cacaaaccacaactagaatgcagtgaaaaaaatgctttatttgtttacagtccgacgatc60
cagcag66
<210>9
<211>23
<212>dna
<213>人工序列
<220>
<223>examplesequencelisting,nj-129,pnt_del_f
<400>9
aattcgccctatagtgagtcgta23
<210>10
<211>64
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-132,gramc_ion-a_ix7_p4s
<400>10
ccatctcatccctgcgtgtctccgactcagttcgtgattcgattacagtccgacgatcca60
gcag64
<210>11
<211>64
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-133,gramc_ion-a_ix8_p4s
<400>11
ccatctcatccctgcgtgtctccgactcagttccgataacgattacagtccgacgatcca60
gcag64
<210>12
<211>64
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-134,gramc_ion-a_ix9_p4s
<400>12
ccatctcatccctgcgtgtctccgactcagtgagcggaacgattacagtccgacgatcca60
gcag64
<210>13
<211>17
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-141,pgramc_np3_short
<220>
<221>misc_feature
<222>(1)..(1)
<223>磷酸化修饰
<400>13
tagactccctcagcggc17
<210>14
<211>21
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-142,pgramc_p4_short
<220>
<221>misc_feature
<222>(1)..(1)
<223>磷酸化修饰
<400>14
tacagtccgacgatccagcag21
<210>15
<211>19
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-145,s_3pofn25mershort
<220>
<221>misc_feature
<222>(1)..(7)
<223>至6-7的核苷酸键是核苷酸1-2的硫代磷酸酯键
<400>15
ggcgcgccgctgagggagt19
<210>16
<211>23
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-146,s_nt_del_f
<220>
<221>misc_feature
<222>(1)..(7)
<223>至6-7的核苷酸键是核苷酸1-2的硫代磷酸酯键
<400>16
aattcgccctatagtgagtcgta23
<210>17
<211>59
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-179,crsp_f_t7_backbone
<400>17
ttaatacgactcactataggtcgtagttatctacacgacggttttagagctagaaatag59
<210>18
<211>59
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-180,crsp_f_t7_gfp
<400>18
ttaatacgactcactataggcgcgctgaagtcaagttcgagttttagagctagaaatag59
<210>19
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-183,crsp_r
<400>19
aaaagcaccgactcggtgcc20
<210>20
<211>64
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-197,gramc_ion-a_ix1_p4s
<400>20
ccatctcatccctgcgtgtctccgactcagctaaggtaacgattacagtccgacgatcca60
gcag64
<210>21
<211>64
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-198,gramc_ion-a_ix2_p4s
<400>21
ccatctcatccctgcgtgtctccgactcagtaaggagaacgattacagtccgacgatcca60
gcag64
<210>22
<211>64
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-200,gramc_ion-a_ix3_p4s
<400>22
ccatctcatccctgcgtgtctccgactcagaagaggattcgattacagtccgacgatcca60
gcag64
<210>23
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-208,pgramc_p1s_not
<220>
<221>misc_feature
<222>(1)..(1)
<223>5'磷酸化修饰
<400>23
attcactagtgattcagcag20
<210>24
<211>18
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-209,pgramc_p2s_not
<220>
<221>misc_feature
<222>(1)..(1)
<223>5'磷酸化修饰
<400>24
gacactactctccagcag18
<210>25
<211>25
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-213,gibson_p1-t
<400>25
gcgaattcactagtgattcagcagt25
<210>26
<211>22
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-214,gibson_inbp-t
<400>26
caagacactactctccagcagt22
<210>27
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-268,hs-top1_qf
<400>27
acttcgtgtggagcacatca20
<210>28
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-269,hs-top1_qr
<400>28
cgtttctcaacagggacctt20
<210>29
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-270,hs-acta1_qf
<400>29
atggtcggtatgggtcagaa20
<210>30
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-271,hs-acta1_qr
<400>30
tctccatgtcatcccagttg20
<210>31
<211>19
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-276,hs-axl_qf2
<400>31
ctgtcagacgatgggatgg19
<210>32
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-277,hs-axl_qr2
<400>32
taaggggtgtgaggatggag20
<210>33
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-278,hs-dlx5_qf
<400>33
tacacaagtgcagccagctc20
<210>34
<211>22
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-279,hs-dlx5_qr
<400>34
gagtaagagagagcagcccatc22
<210>35
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-280,hs-notch2_qf
<400>35
aaatgcctcacaggcttcac20
<210>36
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-281,hs-notch2_qr
<400>36
cactggcactggtaggaacc20
<210>37
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-282,hs-rpp30_qf
<400>37
ctgcttccaggagacctgac20
<210>38
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-283,hs-rpp30_qr
<400>38
tttgtggtgatttcccccta20
<210>39
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-284,hs-adm_qf
<400>39
ggtcggactctggtgtcttc20
<210>40
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-285,hs-adm_qr
<400>40
cttgcgcgactattccttgt20
<210>41
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-286,hs-cfb_qf
<400>41
caagcagacaagcaaagcaa20
<210>42
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-287,hs-cfb_qr
<400>42
gataaagggcatcaggcaga20
<210>43
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-288,hs-kiss1_qf
<400>43
acctgccgaactacaactgg20
<210>44
<211>21
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-289,hs-kiss1_qr
<400>44
tttggggtctgaagttcactg21
<210>45
<211>19
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-292,hs-ncoa6_qf
<400>45
tggcttctcagcaggacag19
<210>46
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-293,hs-ncoa6_qr
<400>46
tgctggacattttgatttgc20
<210>47
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-294,hs-adam12_qf
<400>47
cagttgcagcaggaaggact20
<210>48
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-295,hs-adam12_qr
<400>48
tccacaaatctgttcccaca20
<210>49
<211>78
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-364,pe2_gramc_p4s
<400>49
caagcagaagacggcatacgagatgtgactggagttcagacgtgtgctcttccgatctac60
agtccgacgatccagcag78
<210>50
<211>75
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-399,pe2_gramc_p3s
<400>50
caagcagaagacggcatacgagatgtgactggagttcagacgtgtgctcttccgatctta60
gactccctcagcggc75
<210>51
<211>75
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-400,pe1_gramc_p3s
<400>51
aatgatacggcgaccaccgagatctacactctttccctacacgacgctcttccgatctta60
gactccctcagcggc75
<210>52
<211>75
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-401,pe2_gramc_p2s
<400>52
caagcagaagacggcatacgagatgtgactggagttcagacgtgtgctcttccgatctac60
actactctccagcag75
<210>53
<211>79
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-402,pe1_gramc_p4s
<400>53
aatgatacggcgaccaccgagatctacactctttccctacacgacgctcttccgatctta60
cagtccgacgatccagcag79
<210>54
<211>77
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-403,pe2_gramc_p1s
<400>54
caagcagaagacggcatacgagatgtgactggagttcagacgtgtgctcttccgatcttt60
cactagtgattcagcag77
<210>55
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-404,egfpc1_qf1
<400>55
aagggcatcgacttcaagga20
<210>56
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-405,egfpc1_qr1
<400>56
ggcggatcttgaagttcacc20
<210>57
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-443,gramc_gfp_qf2
<400>57
gccctgtctaaagatcccaa20
<210>58
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-444,gramc_gfp_qr2
<400>58
cttgtacagctcgtccatgc20
<210>59
<211>16
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-489,gramc_rt_oligo
<400>59
tacagtccgacgatcc16
<210>60
<211>58
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-497,pe1_adapter
<400>60
aatgatacggcgaccaccgagatctacactctttccctacacgacgctcttccgatct58
<210>61
<211>44
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-498,pe1s_gramc_2n_p4s
<220>
<221>misc_feature
<222>(22)..(23)
<223>nisa,c,g,toru
<400>61
tacacgacgctcttccgatctnntacagtccgacgatccagcag44
<210>62
<211>46
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-499,pe1s_gramc_4n_p4s
<220>
<221>misc_feature
<222>(22)..(25)
<223>nisa,c,g,toru
<400>62
tacacgacgctcttccgatctnnnntacagtccgacgatccagcag46
<210>63
<211>48
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-500,pe1s_gramc_6n_p4s
<220>
<221>misc_feature
<222>(22)..(27)
<223>nisa,c,g,toru
<400>63
tacacgacgctcttccgatctnnnnnntacagtccgacgatccagcag48
<210>64
<211>50
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-501,pe1s_gramc_8n_p4s
<220>
<221>misc_feature
<222>(22)..(29)
<223>nisa,c,g,toru
<400>64
tacacgacgctcttccgatctnnnnnnnntacagtccgacgatccagcag50
<210>65
<211>52
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-502,pe1s_gramc_10n_p4s
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,toru
<400>65
tacacgacgctcttccgatctnnnnnnnnnntacagtccgacgatccagcag52
<210>66
<211>54
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-503,pe1s_gramc_12n_p4s
<220>
<221>misc_feature
<222>(22)..(33)
<223>nisa,c,g,toru
<400>66
tacacgacgctcttccgatctnnnnnnnnnnnntacagtccgacgatccagcag54
<210>67
<211>40
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-504,pe1s_gramc_2n_np3s
<220>
<221>misc_feature
<222>(22)..(23)
<223>nisa,c,g,toru
<400>67
tacacgacgctcttccgatctnntagactccctcagcggc40
<210>68
<211>42
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-505,pe1s_gramc_4n_np3s
<220>
<221>misc_feature
<222>(22)..(25)
<223>nisa,c,g,toru
<400>68
tacacgacgctcttccgatctnnnntagactccctcagcggc42
<210>69
<211>44
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-506,pe1s_gramc_6n_np3s
<220>
<221>misc_feature
<222>(22)..(27)
<223>nisa,c,g,toru
<400>69
tacacgacgctcttccgatctnnnnnntagactccctcagcggc44
<210>70
<211>46
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-507,pe1s_gramc_8n_np3s
<220>
<221>misc_feature
<222>(22)..(29)
<223>nisa,c,g,toru
<400>70
tacacgacgctcttccgatctnnnnnnnntagactccctcagcggc46
<210>71
<211>48
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-508,pe1s_gramc_10n_np3s
<220>
<221>misc_feature
<222>(22)..(31)
<223>nisa,c,g,toru
<400>71
tacacgacgctcttccgatctnnnnnnnnnntagactccctcagcggc48
<210>72
<211>50
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-509,pe1s_gramc_12n_np3s
<220>
<221>misc_feature
<222>(22)..(33)
<223>nisa,c,g,toru
<400>72
tacacgacgctcttccgatctnnnnnnnnnnnntagactccctcagcggc50
<210>73
<211>40
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-523,gramc_ion-p_np3s
<400>73
cctctctatgggcagtcggtgattagactccctcagcggc40
<210>74
<211>44
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-575,gramc_test1_f
<400>74
ttcactagtgattcagcaggagtgccatcatgattcataaatag44
<210>75
<211>44
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-576,gramc_test1_r
<400>75
acactactctccagcaggtacttaatatttgaggttactcgtag44
<210>76
<211>37
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-577,gramc_test2_f
<400>76
ttcactagtgattcagcagcacctgaccactagtggg37
<210>77
<211>40
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-578,gramc_test2_r
<400>77
acactactctccagcagcactttggaatccaaatttccag40
<210>78
<211>40
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-579,gramc_test3_f
<400>78
ttcactagtgattcagcagcaagtacagcattgactgagc40
<210>79
<211>36
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-580,gramc_test3_r
<400>79
acactactctccagcagagacagagctgacacacac36
<210>80
<211>40
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-589,gramc_test8_f
<400>80
ttcactagtgattcagcagttattttgcttacagggccag40
<210>81
<211>46
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-590,gramc_test8_r
<400>81
acactactctccagcaggtgacacaggagcttatatatatataagc46
<210>82
<211>43
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-591,gramc_test9_f
<400>82
ttcactagtgattcagcagtacaatccacctacttaaagtgtg43
<210>83
<211>39
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-592,gramc_test9_r
<400>83
acactactctccagcagttaaatagagacggggtttcac39
<210>84
<211>43
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-691,g5_1_f
<400>84
ttcactagtgattcagcagcctttctaacttgggtcatttctg43
<210>85
<211>41
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-692,g5_1_r
<400>85
acactactctccagcagctttctttatctacagcaaacagg41
<210>86
<211>45
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-693,g5_2_f
<400>86
ttcactagtgattcagcagcacaagatacatgtagctgaatttag45
<210>87
<211>43
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-694,g5_2_r
<400>87
acactactctccagcagtatttttagtagagacggggtttcac43
<210>88
<211>40
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-695,g5_3_f
<400>88
ttcactagtgattcagcagaaaccctctaggtcctttaac40
<210>89
<211>37
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-696,g5_3_r
<400>89
acactactctccagcagggattacaggaatgtgccac37
<210>90
<211>39
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-697,g5_4_f
<400>90
ttcactagtgattcagcagaaaacaccacgtagtttggc39
<210>91
<211>37
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-699,g5_5_f
<400>91
ttcactagtgattcagcagaagccagcgttgcccatc37
<210>92
<211>36
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-700,g5_5_r
<400>92
acactactctccagcaggcctcagcctcctgagtag36
<210>93
<211>39
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-701,g5_6_f
<400>93
ttcactagtgattcagcaggtaaatccaatcccaggttg39
<210>94
<211>39
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-702,g5_6_r
<400>94
acactactctccagcaggccaccatgtttggctattttc39
<210>95
<211>43
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-705,g3_1_f
<400>95
ttcactagtgattcagcagagttttggtattttaatactcttg43
<210>96
<211>38
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-706,g3_1_r
<400>96
acactactctccagcagcattggttaagtgtagcaaac38
<210>97
<211>43
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-707,g3_2_f
<400>97
ttcactagtgattcagcagatcatttttctttccgagatgttg43
<210>98
<211>42
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-708,g3_2_r
<400>98
acactactctccagcagtattttttttgagatggagtttcgc42
<210>99
<211>40
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-709,g3_3_f
<400>99
ttcactagtgattcagcagcccgttccacaaggatctgtg40
<210>100
<211>38
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-710,g3_3_r
<400>100
acactactctccagcagctccggaatagctgggattac38
<210>101
<211>45
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-711,g3_4_f
<400>101
ttcactagtgattcagcagtctccttataaatatctttcacttcc45
<210>102
<211>38
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-712,g3_4_r
<400>102
acactactctccagcagagaattaagggggaaaagttg38
<210>103
<211>37
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-713,g3_5_f
<400>103
ttcactagtgattcagcaggtggaatctggaggccag37
<210>104
<211>40
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-714,g3_5_r
<400>104
acactactctccagcagttgttggctctggtttttctttg40
<210>105
<211>41
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-717,l1_1_f
<400>105
ttcactagtgattcagcagcttccttcctaccttctttttc41
<210>106
<211>37
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-718,l1_1_r
<400>106
acactactctccagcagaaaacctgggagtcccaaag37
<210>107
<211>41
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-719,l1_2_f
<400>107
ttcactagtgattcagcagaccttcttacttcttaaggggg41
<210>108
<211>40
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-720,l1_2_r
<400>108
acactactctccagcagtctgcgagtcctcctcttctttg40
<210>109
<211>41
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-723,l1_4_f
<400>109
ttcactagtgattcagcaggcaaccagcttggaaatttctc41
<210>110
<211>38
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-724,l1_4_r
<400>110
acactactctccagcagagacttcgacttcttcggatg38
<210>111
<211>41
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-727,l1_6_f
<400>111
ttcactagtgattcagcagaactaacatggctgatgccttg41
<210>112
<211>45
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-728,l1_6_r
<400>112
acactactctccagcagtatttggtttgcttagagtcctcctctg45
<210>113
<211>18
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-729,egfp_5p_f
<400>113
atggtgagcaagggcgag18
<210>114
<211>20
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-730,egfp_3p_r
<400>114
ttatctagatccggtggatc20
<210>115
<211>44
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-731,egfp_gramc_gibson_f
<400>115
gatccaccggatctagataagcctctagactccctcagcggcgc44
<210>116
<211>42
<212>dna
<213>人工序列
<220>
<223>示例引物序列,nj-732,egfp_gramc_gibson_r
<400>116
ctcgcccttgctcaccatttgtgattcacttgtaagatgacg42
<210>117
<211>17
<212>dna
<213>人工序列
<220>
<223>示例修剪衔接子序列,gramcp1s-se.fa
<400>117
tcactagtgattcagca17
<210>118
<211>16
<212>dna
<213>人工序列
<220>
<223>示例修剪衔接子序列,gramcp2s-se.fa
<400>118
acactactctccagca16
<210>119
<211>18
<212>dna
<213>人工序列
<220>
<223>示例修剪衔接子序列,gramcp3s-se.fa
<400>119
actccctcagcggcgcgc18
<210>120
<211>17
<212>dna
<213>人工序列
<220>
<223>示例修剪衔接子序列,gramcp4s-se.fa
<400>120
agtccgacgatccagca17
<210>121
<211>18
<212>dna
<213>人工序列
<220>
<223>示例修剪衔接子序列,gramcp1sr-se.fa
<400>121
ctgctgaatcactagtga18
<210>122
<211>17
<212>dna
<213>人工序列
<220>
<223>示例修剪衔接子序列,gramcp2sr-se.fa
<400>122
ctgctggagagtagtgt17
<210>123
<211>18
<212>dna
<213>人工序列
<220>
<223>示例修剪衔接子序列,gramcp3sr-se.fa
<400>123
gcgcgccgctgagggagt18
<210>124
<211>16
<212>dna
<213>人工序列
<220>
<223>示例修剪衔接子序列,gramcp4sr-se.fa
<400>124
tgctggatcgtcggac16
- 还没有人留言评论。精彩留言会获得点赞!